基于稀疏网络的可见光/近红外反射光谱土壤有机质含量估算  下载: 925次
下载: 925次
1 引言
土壤有机质(SOM)作为土壤的重要养分来源之一,能够提高土壤肥力、促进植物的生长以及实现农林业的可持续发展,而且及时有效地掌握SOM含量的变化,对湿地的保护与维护有重要的指导意义[1-3]。传统的化学测定方法有较高的测量精度,但需要消耗大量的时间成本且价格昂贵,在实际应用中难以大规模使用。由于SOM在可见光和近红外(VIS-NIR)光谱中有着独特的吸收特性,因此常用来估算SOM含量。VIS-NIR光谱技术作为研究已超过二十年的成熟技术,在估算SOM含量的准确性和经济效益上已得到科研工作者的广泛认可[4-6]。土壤光谱与SOM含量之间通常呈非线性相关,而光谱中包含若干噪声,从中探明与SOM有关的敏感变量仍存在一定难度,因此对土壤光谱进行特征变量的提取和干扰信息的剔除是模型具有较高准确性的重要保证[7-9]。
在VIS-NIR光谱数据的研究过程中,光谱的预处理方式与模型的构建是提高预测精度的关键。Hong等[10]采用了不同的分数阶导数对土壤反射率光谱进行预处理,发现随着导数阶数的增加,基线漂移的现象和重叠峰逐渐消失,但更容易受到噪声的干扰。章海亮等[11]应用了遗传算法与连续投影算法对波段进行优化,结合偏最小二乘回归建立有机质预测模型,结果表明对原始波段进行筛选优化能够显著提高模型的预测精度。栾福明等[12-13]通过相关分析和主成分分析(PCA)选择了特征变量,结合BP(Back Propagation)神经网络建立更为简洁的模型,从而提高了模型的预测性能。国内外学者采用不同的变量筛选与建模方法对SOM含量进行了大量的研究并取得了显著的成果,但这些模型难以充分探讨土壤光谱之间的线性与非线性关系,无法提取包含深层特征的光谱。由于模型收敛具有不稳定,常存在诸如局部最优和过拟合等问题[14],如何准确提取VIS-NIR光谱中的信息显得尤为重要。
深度学习算法在机器学习领域中带来了一系列突破,该算法可以自动学习和提取光谱数据中的固有和深层次特征[15]。因采用深度学习算法估算SOM的研究较少,为此提出一种新颖的深度学习算法以反演SOM。深度学习的核心是神经网络,如深度神经网络、卷积神经网络和自动编码器神经网络。与有监督的深度神经网络不同,自动编码器是一种无监督的特征学习神经网络,仅使用几层网络就可以从数据中提取特征[16],已成功应用于医学预测[17]和图像处理[18]等领域。基于此,本文针对SOM提出一种新的稀疏自编码-BP(SAE-BP)网络模型,以期能够最大程度地提高模型的预测性能及精度,为今后研制土壤光谱传感器提供理论依据。
2 数据与方法
2.1 土壤样品的采集
研究区域位于新疆北部的艾比湖湿地,地处亚欧大陆腹地(43°38'N—45°52'N,79°53'E—85°02'E),该区域的土壤质地以砂土、壤土、黏壤土和黏土为主。实验共采集103个样品,土样均匀分布于艾比湖湿地的周围,将样品带回实验室之后自然风干,研磨后过0.5 mm孔径的筛子过筛,采用重铬酸钾-硫酸溶液加热法来测定SOM。
2.2 光谱数据的采集
采用美国ASD公司的ASD Field Spec®3 HR便携式光谱仪(波长范围为350~2500 nm)来测定土壤的光谱反射率数据,重采样间隔为1 nm,每个土壤数据累计得到2151条波段。光谱测定过程是在暗室中进行的,光源为50 W功率的卤化灯,探头的入射角度为15°,探头距离样品表面为10 cm,光源距离样品表面为50 cm,每次反射率的测定均使用白板进行标定,每个样品均测量5次,取其平均值作为该样品的光谱反射率。实际的测定过程中,不同传感器之间的响应精度不同,使得位于两端的光谱数据易混入噪声信息,每份土样均对350~400 nm和2401~2500 nm波段的光谱进行剔除。
2.3 光谱数据的预处理及异常数据的剔除
为了突出原始数据的光谱信息、提高信噪比以及消除高频随机噪声对模型的影响,采用SG(Savitzky-Golay)平滑以及微分处理对原始光谱数据进行预处理,其中微分处理包含一阶微分处理(FDR)和二阶微分处理(SDR)。SG平滑可以提高光谱的平滑性,降低噪声的干扰;光谱微分处理能够部分消除外部环境的干扰,提高灵敏度与光谱的分辨率。此外,为了减少异常样本对模型性能产生干扰,使用PCA与马氏距离的结合方法对异常样本进行剔除[19]。
2.4 变量筛选方法
采用多种光谱变量筛选方法对原始光谱进行筛选,并结合PLSR(Partial least squares regression)和BP网络模型来预测SOM,目标算法主要包括连续投影算法(SPA)、PCA与SAE。
SPA是一种使矢量空间共线性最小化的前向变量选择算法,其在向量空间中执行简单的投影操作以获得共线性最小的有用变量子集,能够在有效消除变量间共线性的同时得到最低限度的冗余信息的变量组合,从而实现在较低的模型复杂度下以最大限度地获取解释信息。SPA的变量选择原则为新选择的变量是在原选择变量的正交子空间上选择具有最大投影值的变量,其可以在方均根误差(RMSE)最小的基础上确定最优的初始变量和变量数量,因此常用于光谱特征波长的筛选。PCA是一种常用的数据压缩算法,通过变换可以得到具有相同变量数量的新变量。这些新变量是原变量的线性组合且彼此正交,包含的信息不重叠,进而消除变量之间的多重共线性。理论上,采用PCA可以获得主成分的维度与原始变量数据相同,但由于前几个主成分的贡献率较大,则只需保留几个贡献较大的主成分,即可保留原始数据中大部分的信息。
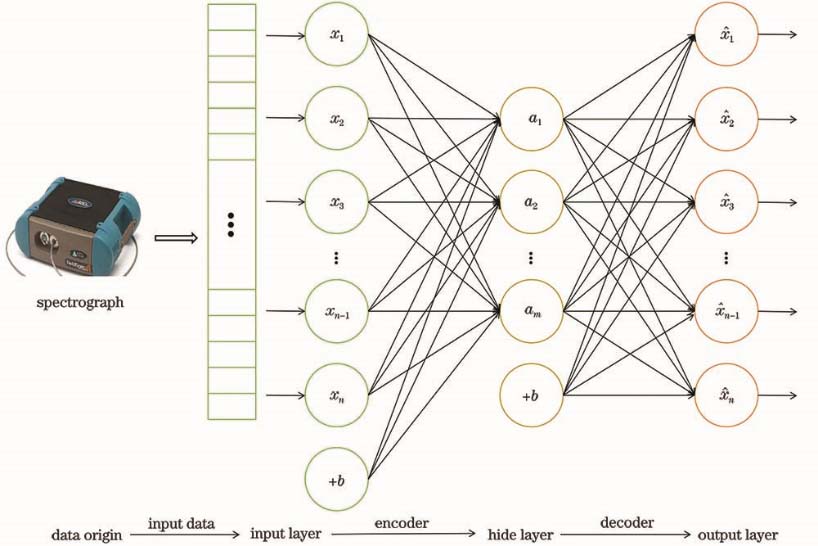
自编码器(AE)尝试学习一个Hw,b (x)≈x的函数,而实际输出变量
SAE是AE的改进,即在AE的优化目标函数中添加额外的惩罚因子,用来限制隐藏层中被激活的节点数,通过寻找一组超完备基来高效地表示数据的一种无监督学习方法,表达式为
式中:
式中:P为输入层神经元的总数;β为惩罚因子的权重。
2.5 模型评价
实验过程中,分别建立线性(多元线性回归模型和贝叶斯线性回归)和非线性(反向传播神经网络模型)两种不同的模型来预测SOM。模型评价指标包括决定系数(R2)、RMSE、剩余预测偏差(RPD)以及四分位数间隔(RPIQ)。R2用来表示模型的拟合程度,R2值越接近于1,模型的拟合效果越好;RMSE用来表示模型的估算能力,RMSE值越小越好;RPD用来表示模型预测的准确性,通常来说当xRPD<1.4时,意味着模型的量化能力较弱,当1.4≤xRPD≤2.0时,表示模型的量化能力尚可,当xRPD>2.0时,表示模型的量化能力极强;RPIQ为四分位间距与RMSE的比[20]。采用Kennard-Stone算法来划分数据集,选取59个样本点作为训练集,30个样本点作为验证集,分别建立预测模型。
3 结果与分析
3.1 SOM统计信息
实验过程中不可避免地存在误差,采用PCA与马氏距离的结合方法对异常样品进行剔除,共得到89个样本可以用于分析建模,如

图 2. PCA与马氏距离的结合方法剔除异常样本的结果
Fig. 2. Results of combined method of PCA and Mahalanobis distance to eliminate abnormal samples
表 1. SOM的统计信息
Table 1. SOM statistics
|
3.2 光谱特征分析
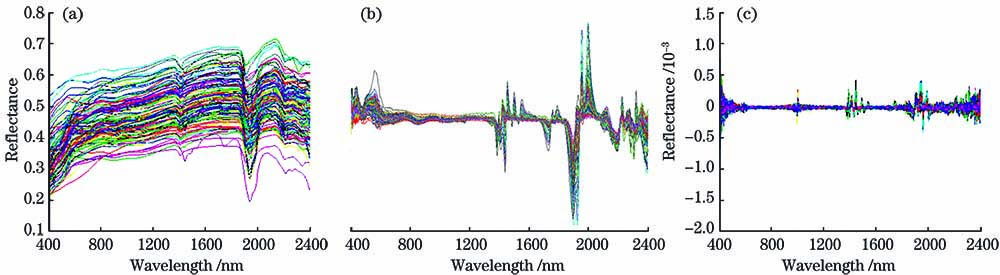
图 3. SOM与不同VIS-NIR光谱的反射率曲线。(a)原始光谱;(b)一阶光谱;(c)二阶光谱
Fig. 3. Reflectance curves of SOM and different VIS-NIR spectra. (a) Original spectrum; (b) first-order spectrum; (c) second-order spectrum
SOM与不同VIS-NIR光谱的相关系数如
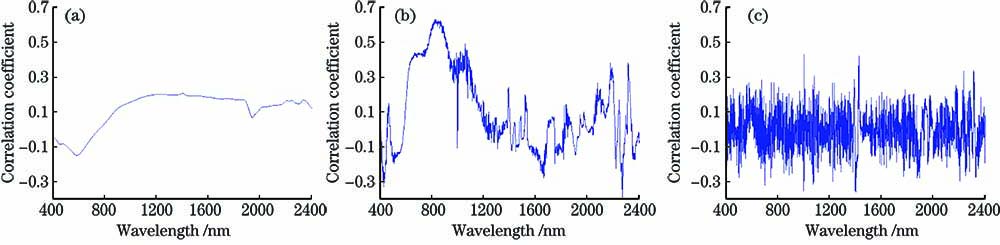
图 4. 不同VIS-NIR光谱与SOM的相关性曲线。(a)原始光谱;(b)一阶光谱;(c)二阶光谱
Fig. 4. Correlation curves of different VIS-NIR spectra and SOM. (a) Original spectrum; (b) first-order spectrum; (c) second-order spectrum
3.3 光谱特征变量的提取
为了获得具有稳健预测能力和少量输入变量的最优模型,采用三种变量选择方法(SPA、PCA和SAE)来筛选光谱特征,从而估算SOM。
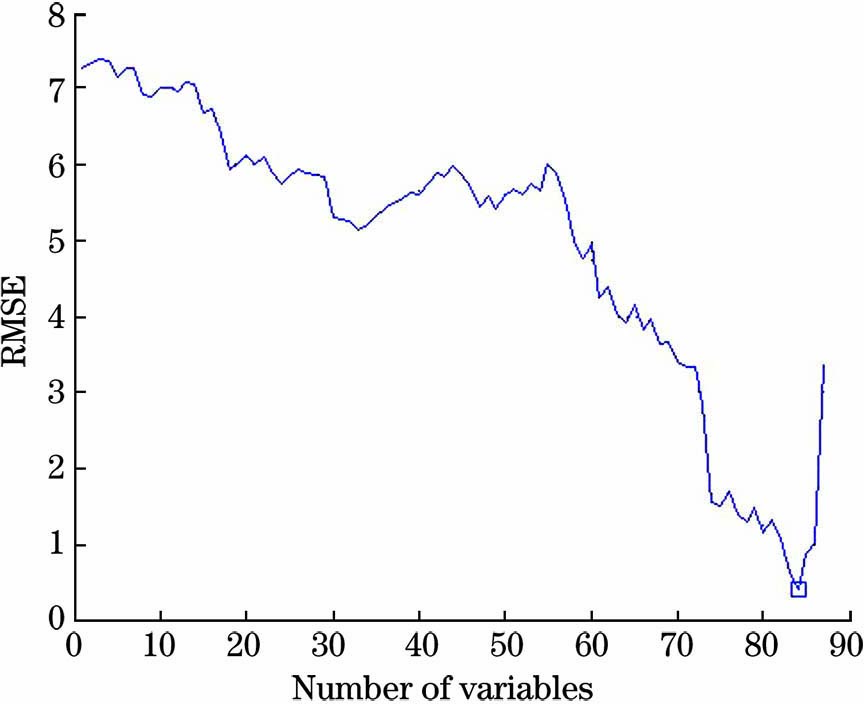
3.3.1 PCA和SPA方法提取特征变量
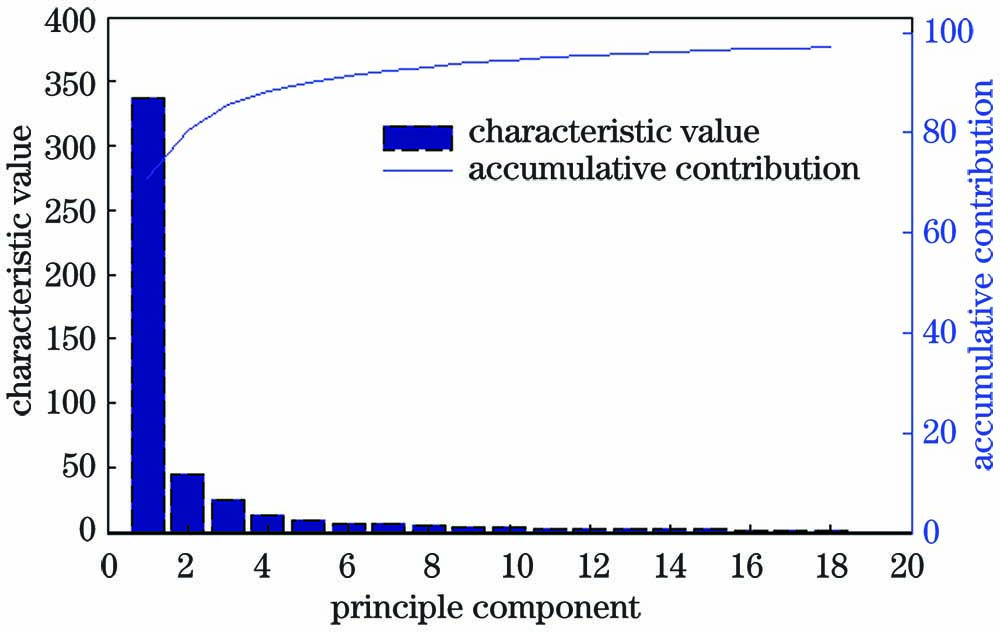
利用SPSS 22.0软件对一阶导数光谱进行PCA处理,选取特征值大于1的因子,结果如
3.3.2 SAE方法提取特征变量
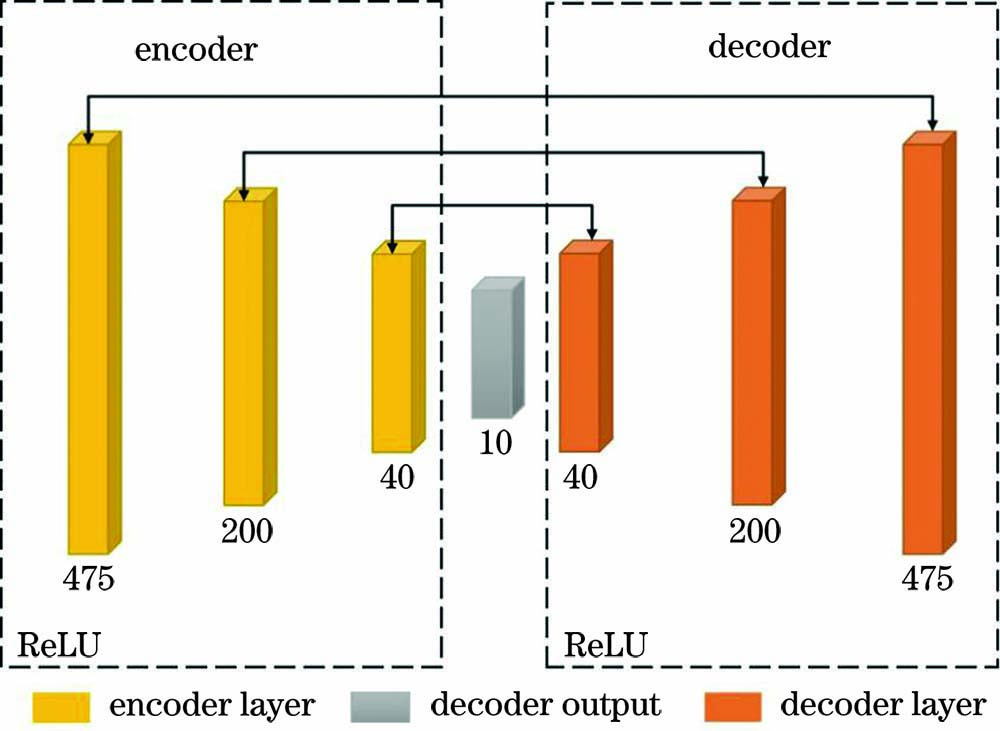
深度学习的学习能力受到参数的影响,准确选择参数对于模型的构建来说十分重要。传统的神经网络模型中,通常采用Sigmoid和Tanh函数作为激活函数,但是对于网络层数较多的神经网络往往存在计算量大、反向传播过程中梯度消失及信息丢失等问题,因此选择ReLU作为激活函数,可以减少运算量且降低过拟合发生的概率,优化器选用Adam,损失函数采用均方对数误差函数。一次训练所选取的样本数设置为30,迭代次数为100次,学习率为0.001。选择475个一阶导数光谱作为输入层数据,将数据压缩至200个,再转换到40维。为了选择最优的维度,在网络的第4层将数据维度分别压缩至4,6,8,10,12,14,16,18维进行实验,最后特征变量通过解码层依次重建为475个光谱波段,SAE网络结构及参数如
3.3.3 变量筛选方法的对比分析
变量筛选方法与SOM中R2的关系,如
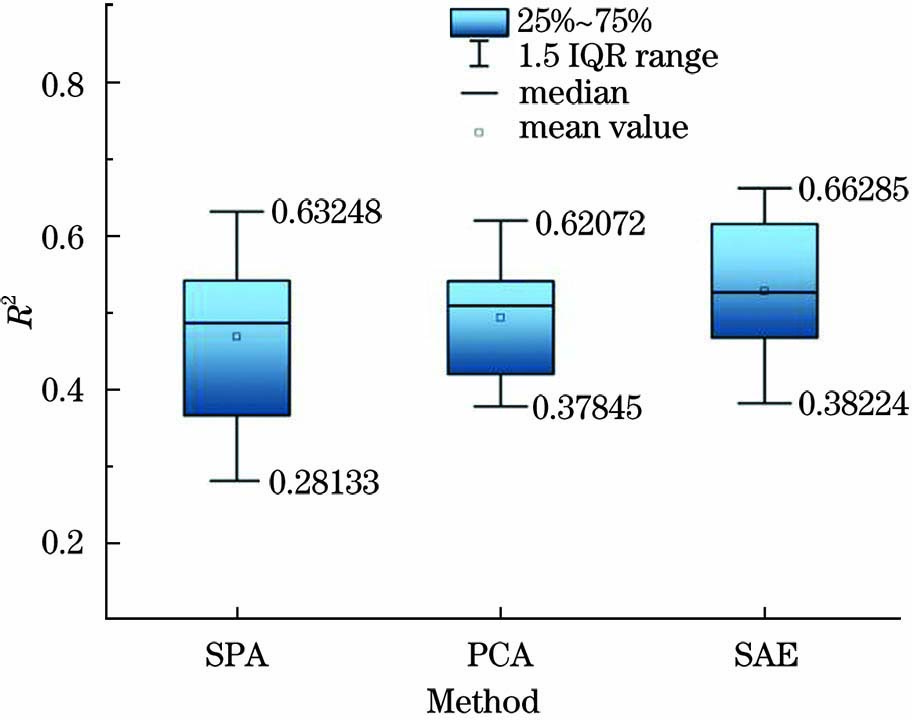
图 8. 变量筛选方法与SOM中R2的关系
Fig. 8. Relationship between variable selection method and R2 in SOM
3.4 模型的构建及对比分析
选择有效的变量能够大幅度提升模型的预测能力,采用SAE、PCA和SPA方法挑选不同的特征变量,并结合PLSR和BP神经网络来建立预测模型。
表 2. 不同特征变量筛选与建模方法的模型精度
Table 2. Model accuracy of different feature variable selections and modeling methods
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
4 讨论
光谱变量的筛选是研究土壤VIS-NIR光谱的重要步骤,其可以有效去除光谱中的冗杂信息,提高模型的预测精度。SPA方法在PLSR模型和BP模型中的效果都较差,但其在很大程度上可以避免光谱信息的重叠,当筛选变量时倾向于选择不稳定的波段并剔除一些重要的相关波段,使得部分波段的信息缺失,从而影响模型的预测精度[22-23]。此外,较长的计算时间和所选波长的数量(不能大于校准样品的数量)是SPA应用的两个主要难题。PCA方法在PLSR模型和BP模型中的表现则大不相同,其是通过降维的思想以及使用较少的综合变量来替代原本较多的变量,且这些综合变量之间相互独立,可以弱化变量自相关所引起的误差,但当光谱中存在较多的非线性信息时,数据降维的效果并不明显[24-25],即在线性的PLSR模型中取得的效果较好,而在非线性的BP网络中取得的效果一般。SAE方法通过计算输入光谱数据与输出光谱数据之间的误差,不断调节参数以学习数据内部隐藏的特征,从而压缩输入光谱以提取有用的光谱特征[26]。在SAE方法的基础上进行BP建模所得到的模型精度最高,说明SAE-BP方法可以有效地估算SOM。
从
实验采用不同的变量筛选方法,结合PLSR模型和BP神经网络算法对新疆艾比湖湿地的SOM进行反演估算,可以取得较好的效果。然而由于人为的实验误差以及复杂的土壤光谱特性,如何优选光谱以及去除土壤中的干扰因素,建立更稳健的模型仍需进一步研究。
5 结论
在土壤的有机质光谱的建模过程中,光谱数据的预处理方法以及建模方法都会影响建模的预测效果。以艾比湖湿地的89个土壤采样点作为研究对象,采用SAE、PCA和SPA方法从原始光谱中获取特征变量,建立基于特征变量的PLSR和BP预测模型。实验结果表明,在土壤的光谱数据中通常包含许多冗杂信息,对其进行有效剔除能够提升模型的预测精度。土壤的原始光谱与SOM之间的相关性较差,通过对原始光谱进行平滑微分处理能够取得显著效果,其中一阶导数的相关性高于二阶导数;SAE方法的降维效果优于SPA方法和PCA方法。对比分析SAE、PCA和SPA数据降维方法以及PLSR模型和BP模型,SAE-BP模型在估算SOM中取得的精度最高,得到R2=0.82、xRMSE=3.53、xRPD=2.32和xRPIQ=2.78,说明该模型能够较好地预测SOM,这与SAE方法和BP神经网络都是网络结构以及能够更好地处理光谱中非线性信息相关。
[2] Shi Z, Ji W. Viscarra Rossel R A, et al. Prediction of soil organic matter using a spatially constrained local partial least squares regression and the Chinese VIS-NIR spectral library[J]. European Journal of Soil Science, 2015, 66(4): 679-687.
[3] Wang X P, Zhang F, Kung H T, et al. New methods for improving the remote sensing estimation of soil organic matter content (SOMC) in the Ebinur Lake Wetland National Nature Reserve (ELWNNR) in northwest China[J]. Remote Sensing of Environment, 2018, 218: 104-118.
[4] Ben-Dor E, Banin A. Near-infrared analysis as a rapid method to simultaneously evaluate several soil properties[J]. Soil Science Society of America Journal, 1995, 59(2): 364-372.
[5] Zhang Z P, Ding J L, Wang J Z, et al. Prediction of soil organic matter in northwestern China using fractional-order derivative spectroscopy and modified normalized difference indices[J]. Catena, 2020, 185(10): 104257.
[6] 李冠稳, 高小红, 肖能文, 等. 特征变量选择和回归方法相结合的土壤有机质含量估算[J]. 光学学报, 2019, 39(9): 0930002.
[7] 何东健, 陈煦. 土壤有机质含量田间实时测定方法[J]. 农业机械学报, 2015, 46(1): 127-132.
He D J, Chen X. Real-time measurement of soil organic matter content in field[J]. Transactions of the Chinese Society for Agricultural Machinery, 2015, 46(1): 127-132.
[8] 王海峰, 张智韬, Arnon Karnieli, 等. 基于灰度关联-岭回归的荒漠土壤有机质含量高光谱估算[J]. 农业工程学报, 2018, 34(14): 124-131.
[9] Shi Z, Wang Q L, Peng J, et al. Development of a national VNIR soil-spectral library for soil classification and prediction of organic matter concentrations[J]. Science China Earth Sciences, 2014, 57(7): 1671-1680.
[10] Hong Y S, Chen S C, Liu Y L, et al. Combination of fractional order derivative and memory-based learning algorithm to improve the estimation accuracy of soil organic matter by visible and near-infrared spectroscopy[J]. Catena, 2019, 174: 104-116.
[11] 章海亮, 罗微, 刘雪梅, 等. 应用遗传算法结合连续投影算法近红外光谱检测土壤有机质研究[J]. 光谱学与光谱分析, 2017, 37(2): 584-587.
[12] 栾福明, 张小雷, 熊黑钢, 等. 基于不同模型的土壤有机质含量高光谱反演比较分析[J]. 光谱学与光谱分析, 2013, 33(1): 196-200.
[13] 叶勤, 姜雪芹, 李西灿, 等. 基于高光谱数据的土壤有机质含量反演模型比较[J]. 农业机械学报, 2017, 48(3): 164-172.
Ye Q, Jiang X Q, Li X C, et al. Comparison on inversion model of soil organic matter content based on hyperspectral data[J]. Transactions of the Chinese Society for Agricultural Machinery, 2017, 48(3): 164-172.
[14] Huang F M, Zhang J, Zhou C B, et al. A deep learning algorithm using a fully connected sparse autoencoder neural network for landslide susceptibility prediction[J]. Landslides, 2020, 17(1): 217-229.
[15] Ayinde B O, Inanc T, Zurada J M. Regularizing deep neural networks by enhancing diversity in feature extraction[J]. IEEE Transactions on Neural Networks and Learning Systems, 2019, 30(9): 2650-2661.
[16] FengJ, Zhou ZH. Autoencoder by forest[C]//Thirty-Second AAAI Conference on Artificial Intelligence. New York: AAAI, 2018.
[17] Bello G A. Dawes T J W, Duan J M, et al. Deep-learning cardiac motion analysis for human survival prediction[J]. Nature Machine Intelligence, 2019, 1(2): 95-104.
[18] ZhangR, IsolaP, Efros AA. Split-brain autoencoders: unsupervised learning by cross-channel prediction[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), July 21-26, 2017, Honolulu, HI, USA. New York: IEEE, 2017: 645- 654.
[19] 陈斌, 邹贤勇, 朱文静. PCA结合马氏距离法剔除近红外异常样品[J]. 江苏大学学报(自然科学版), 2008, 29(4): 277-279, 292.
Chen B, Zou X Y, Zhu W J. Eliminating outlier samples in near-infrared model by method of PCA-mahalanobis distance[J]. Journal of Jiangsu University (Natural Science Edition), 2008, 29(4): 277-279, 292.
[20] 葛翔宇, 丁建丽, 王敬哲, 等. 基于竞争适应重加权采样算法耦合机器学习的土壤含水量估算[J]. 光学学报, 2018, 38(10): 1030001.
[21] Hong Y S, Liu Y L, Chen Y Y, et al. Application of fractional-order derivative in the quantitative estimation of soil organic matter content through visible and near-infrared spectroscopy[J]. Geoderma, 2019, 337: 758-769.
[22] Xu S X, Zhao Y C, Wang M Y, et al. Determination of rice root density from Vis-NIR spectroscopy by support vector machine regression and spectral variable selection techniques[J]. Catena, 2017, 157: 12-23.
[23] 于雷, 洪永胜, 周勇, 等. 高光谱估算土壤有机质含量的波长变量筛选方法[J]. 农业工程学报, 2016, 32(13): 95-102.
Yu L, Hong Y S, Zhou Y, et al. Wavelength variable selection methods for estimation of soil organic matter content using hyperspectral technique[J]. Transactions of the Chinese Society of Agricultural Engineering, 2016, 32(13): 95-102.
[24] Ouyang Q, Chen Q S, Zhao J W. Intelligent sensing sensory quality of Chinese rice wine using near infrared spectroscopy and nonlinear tools[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2016, 154: 42-46.
[25] Morellos A, Pantazi X E, Moshou D, et al. Machine learning based prediction of soil total nitrogen, organic carbon and moisture content by using VIS-NIR spectroscopy[J]. Biosystems Engineering, 2016, 152: 104-116.
[26] 袁非牛, 章琳, 史劲亭, 等. 自编码神经网络理论及应用综述[J]. 计算机学报, 2019, 42(1): 203-230.
Yuan F N, Zhang L, Shi J T, et al. Theories andapplications of auto-encoder neural networks: a literature survey[J]. Chinese Journal of Computers, 2019, 42(1): 203-230.
Article Outline
冉思, 丁建丽, 葛翔宇, 刘博华, 张钧泳. 基于稀疏网络的可见光/近红外反射光谱土壤有机质含量估算[J]. 激光与光电子学进展, 2020, 57(24): 242803. Si Ran, Jianli Ding, Xiangyu Ge, Bohua Liu, Junyong Zhang. Estimation Method of VIS-NIR Spectroscopy for Soil Organic Matter Based on Sparse Networks[J]. Laser & Optoelectronics Progress, 2020, 57(24): 242803.