基于注意力U-Net的脑肿瘤磁共振图像分割  下载: 1772次
下载: 1772次
1 引言
神经胶质瘤是由于神经胶质细胞突变而产生的颅内原发性中枢神经系统肿瘤,其中几乎80%的恶性脑肿瘤是胶质瘤,恶性胶质瘤具有免疫逃逸能力,会使机体无法自然地对其产生特异性杀伤细胞和抗体,其中胶质母细胞瘤即使经过手术和放化疗,其生存时间也只有9.9个月[1]。由于磁共振(Magnetic Resonance,MR)图像具有较高的软组织对比度和出色的功能成像能力,可以生成T1加权像(T1 weighted)、T1ce(Contrast enhanced T1-weighted)、T2加权像(T2 weighted)及FLAIR(Fluid Attenuated Inversion Recovery)等多参数MR图像来为脑肿瘤的描述提供准确的信息[2]。医学界根据患者临床表现,结合MR图像对脑肿瘤分级,并对脑肿瘤患者进行手术治疗、放射治疗、化学治疗等综合治疗。MR扫描一次会得到大量不同模态的图像,分析、注释和分割图像的时间会影响对患者的诊断和治疗。因此,需要快速、准确的全自动方法用于脑肿瘤MR图像的分割,以为临床应用提供参考。
目前脑肿瘤MR图像分割方法分为三类:人工分割、半自动分割和自动分割。人工分割是在所有的肿瘤MR图像切片中手工勾画出肿瘤区域,人工分割的方法虽然准确,但是费时、费力且存在主观因素影响。半自动分割方法包括阈值分割[3]、分水岭分割[4]、区域分割[5]等。半自动分割方法具有快速、简单的优点,但分割结果依赖于用户指定的参数和MR图像的预处理。
自动分割方法一般采用全卷积神经网络(FCN)、U-Net等,在脑肿瘤MR图像分割中取得了很大的进展。Zhang等[6]利用原始脑MR图像作为训练样本,训练粗分割FCN模型得到肿瘤区域,再以肿瘤区域为训练样本进行FCN训练,精细分割肿瘤区域的内部结构,但是该过程中缺失了局部信息,得到的结果还是不够精细。Dong等[7]使用U-Net对脑肿瘤MR图像进行分割,通过跳过连接拼接扩展路径和收缩路径的特征向量,取得了较好的结果。但是简单的图像特征拼接依赖固定权重,且信息会随着分层传播而遗失,分割精度不高。针对上述方法的不足之处,本文引入注意力机制对U-Net进行改进,提高了对肿瘤区域分割的准确度。
2 网络结构
传统的基于卷积神经网络(CNN)的图像分割方法利用多层卷积操作自动提取图像的多个层次特征,在卷积层后连接若干全连接层得到固定长度的特征向量,并作为图像分类的概率。Long等[8]提出端对端的图像语义分割FCN方法,即把CNN最后的全连接层换成卷积层,用反卷积对卷积层提取的特征图进行上采样,使上采样图像尺寸恢复到与输入图像相同的尺寸,从而实现图像的分割,但是该方法只用到深层图像特征,分割精度低。
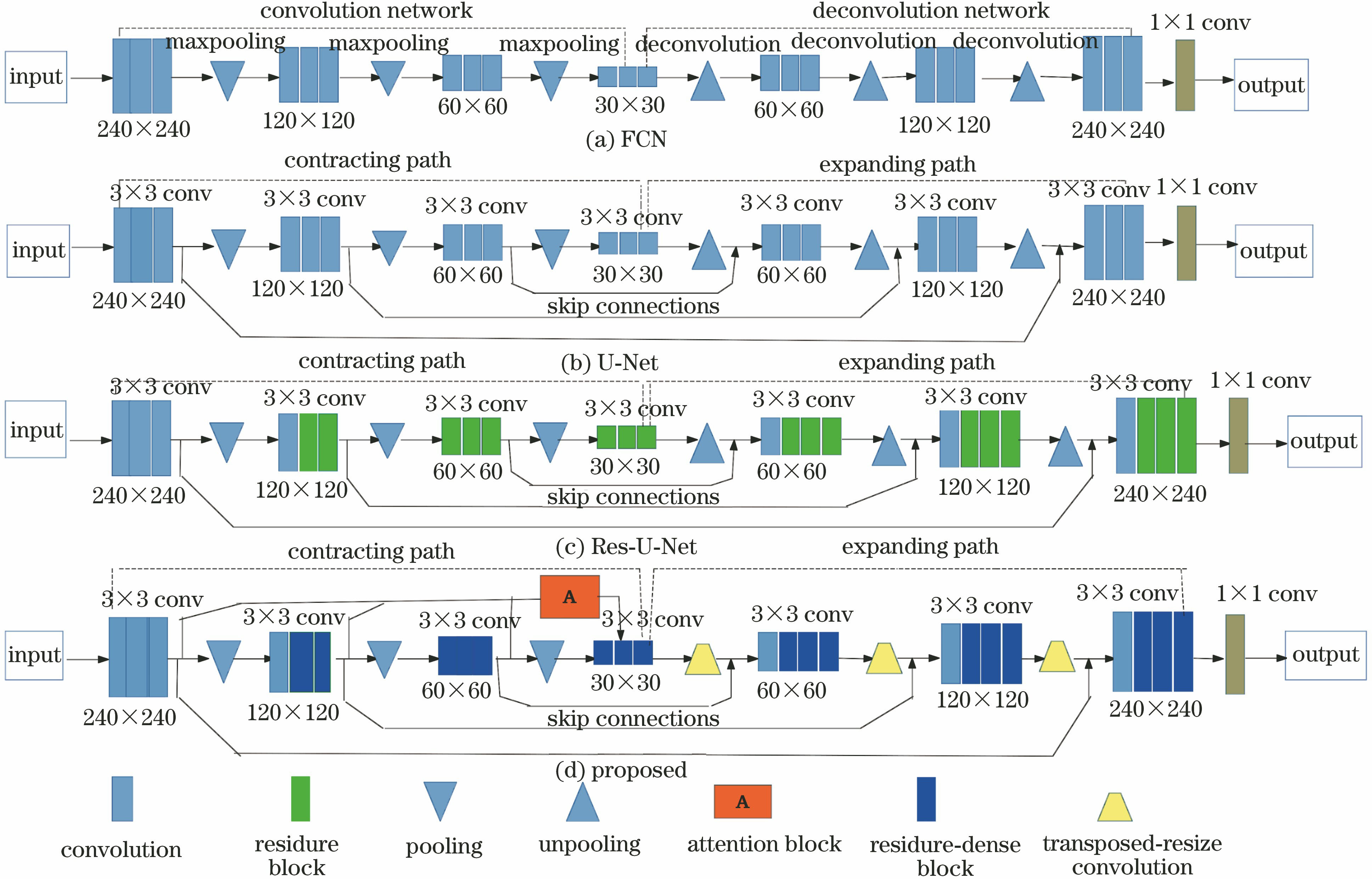
图 1. 网络结构图。(a) FCN;(b) U-Net;(c) Res-U-Net;(d)本文改进的方法
Fig. 1. Network structure. (a) FCN; (b) U-Net; (c) Res-U-Net; (d) proposed method
对神经网络的卷积模块进行设计时,残差连接通过将前后层特征相加实现了特征重用,结构如

图 2. 网络结构的卷积模块。(a) Residure模块;(b) dense模块;(c) residure-dense模块
Fig. 2. Convolution module of the network structure. (a) Residure modules; (b) dense module; (c) residure-dense module

图 3. 网络结构的注意力模块图。(a) SE-Net的注意力单元;(b)本文注意力单元
Fig. 3. Aattention module of SE-Net. (a) SE-Net attention unit; (b) proposed attention unit
视觉注意力机制从大量信息中快速筛选出高价值信息,SE网络(Squeeze-and-Excitation Networks,SE-Net)通过注意力自动获取每个特征通道的重要程度,然后依照重要程度,提升有用特征,抑制用处不大的特征,将权重加权到每个通道的特征上[14]。原始SE-Net的注意力SE模块如
3 实验设置
3.1 数据集与预处理
实验数据采用了由MICCAI提供的脑胶质瘤的公共数据集BraTS2017[15-18],数据集包括210名高级别胶质瘤(HGG)患者和75名低级别胶质瘤(LGG)患者的四种模态MR图像和真实分割标签,每位患者都有155张不同扫描层的图像,尺寸大小均为240 pixel×240 pixel,如

图 4. 实验数据图。(a) TI图像;(b) T1ce图像;(c) T2图像;(d) FLAIR图像;(e)真实分割标签
Fig. 4. Experimental data. (a) TI image; (b) T1ce image; (c) T2 image; (d) FLAIR image; (E) ground truth
3.2 损失函数
本文选择由交叉熵损失和广义Dice损失[20]组合的混合损失函数,损失的交叉熵部分能够确保背景被正确分割,提高了分割精度。广义的骰子损失将Dice系数最大化到接近于零的值,从而使模型收敛更加快速。交叉熵损失函数定义为
式中:N为所有样本的集合;L为所有标签的集合;yil为第i个样本和第l种标签的一个热编码(0或1);
式中:ξ是防止出现分母为0导致计算错误而设置的一个极小数,在实验中将ξ固定到0.1410301;Wl表示每个类的权重,为了确保所有类都被平等对待,最大化每个类的Dice系数,设计每个类的权重与其频率平方成反比,即
当出现分母为零的情况,实验中会将Wl设为零。最后的混合损失函数的表达式为
式中:β为超参数,用于控制LD和Lc之间的平衡。通过实验可知,将超参数β固定到β=0.7时,网络训练损失最小。
3.3 实验评估
为了评估所提出方法的性能,使用图像分割最常用的标准性能指标(Dice系数、灵敏度和特异度)进行分割,其表达式分别为
式中:TP、TN、FP和FN分别对应真阳性、真阴性、假阳性和假阴性。
这些指标用于评估肿瘤增强区域(简写为WT,仅增强区域被认为是阳性,其余均被认为是阴性)、肿瘤核心区域(简写为TC,坏死核心、增强型肿瘤和非增强型肿瘤合并为阳性)和肿瘤整体区域(简写为ET,所有肿瘤区域为阳性)。
4 实验结果与分析
实验硬件实验环境为:Ubuntu18.04 LST64 位操作系统,64G内存,显卡NVIDIA GeForce GTX1080Ti,处理器intel(R) Core(TM) i7-7400K CPU 3.60GHz。软件环境采用TensorFlow作为后端进行数值实验,在CUDA 9.0架构平台上进行并行计算,调用深度神经网络计算库CuDNN 7.5对运算进行加速。网络训练模型时,输入的图像包括FLAIR、T1ce加权像和T2加权像三个MRI模态的图像,以及专家分割好的真实分割标签。使用完全相同的样本分别在FCN网络、U-Net 网络、Res-U-Net网络进行对比实验,最后采用本文提出的网络里进行实验。学习率初始时为0.005,在卷积层里使用dropout来防止过拟合,提高泛化能力,dropout取值为0.5,权重衰减系数为0.0001。激活函数使用参数化修正线性单元PReLU,批量大小设置为20,训练迭代次数为10000,优化方法采用随机梯度下降(SGD)法。最后,该网络输出包括增强肿瘤区、肿瘤核心、整体肿瘤区和背景区域4类分割标签。
表 3. 本文方法与其他先进分割方法的Dice对比
Table 3. Dice comparison between proposed method and other advanced segmentation methods
|
表 2. 在U-Net结构添加不同模块的评估结果
Table 2. Results of adding different blocks to the U-NET structure%
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 1. 四种模型的评估结果
Table 1. Results of the four models%
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||

图 5. 四种模型在LGG和HGG的三个不同水平切面的实验结果图。(a) LGG;(b) HGG
Fig. 5. Experimental results of the four models at three different levels of LGG and HGG. (a) LGG; (b) HGG

图 6. 不同Epoch权重系数下网络损失变化对比图
Fig. 6. Comparison of network loss changes under different epoch weighting factors
4 结论
针对基于全卷积神经网络为代表的U-Net和Res-U-Net分割方法计算复杂度高且精度不高的缺陷,提出了一种基于注意力U-Net的脑肿瘤MR图像分割算法。用残差密集模块替代原有卷积层,用转置卷积和缩放卷积模块进行上采样,同时利用注意机制处理不同层次的特征层。通过实验,将FCN、U-Net、Res-U-Net和本文所提方法进行了比较,结果表明,本文提出的网络结构更加精细,能够有效地解决脑肿瘤MR图像的过分割与欠分割问题,分割预测效果好,可实现较高的分割精度,其中Dice评分结果为肿瘤整体区域0.90分、肿瘤核心区域0.79分和肿瘤增强区域0.78分。同时,在U-Net结构添加不同模块进行对比实验,结果表明残差密集模块、转置卷积和缩放卷积模块和注意力模块都对网络性能的提升有促进作用。因此,本研究将对脑肿瘤MR图像分割中学习注意机制的研究提供新的思路。
[1] Louis D N, Perry A, Reifenberger G, et al. The 2016 world health organization classification of tumors of the central nervous system: a summary[J]. Acta Neuropathologica, 2016, 131(6): 803-820.
[2] 任璐, 李锵, 关欣, 等. 改进的连续型最大流算法脑肿瘤磁核共振成像三维分割[J]. 激光与光电子学进展, 2018, 55(11): 111011.
[3] 赖小波, 许茂盛, 徐小媚. 多模态MR图像和多特征融合的胶质母细胞瘤自动分割[J]. 计算机辅助设计与图形学学报, 2019, 31(3): 421-430.
Lai X B, Xu M S, Xu X M. Automatic segmentation for glioblastoma multiforme using multimodal MR images and multiple features[J]. Journal of Computer-Aided Design & Computer Graphics, 2019, 31(3): 421-430.
[4] 李永焯, 戴曙光. 改进分水岭算法在脑肿瘤CT图像分割中的应用[J]. 软件导刊, 2018, 17(6): 157-159.
Li Y Z, Dai S G. Application of improved watershed algorithm in segmentation of brain tumor CT images[J]. Software Guide, 2018, 17(6): 157-159.
[5] 蒋秋霖, 王昕. 基于区域生长算法的脑肿瘤图像分割[J]. 长春工业大学学报, 2018, 39(5): 490-493.
Jiang Q L, Wang X. Brain tumor image segmentation based on region growing algorithm[J]. Journal of Changchun University of Technology, 2018, 39(5): 490-493.
[6] Zhang C J, Fang M C, Nie H H. Brain tumor segmentation using fully convolutional networks from magnetic resonance imaging[J]. Journal of Medical Imaging and Health Informatics, 2018, 8(8): 1546-1553.
[7] DongH, YangG, Liu FD, et al.Automatic brain tumor detection and segmentation using U-net based fully convolutional networks[M] ∥Communications in Computer and Information Science. Cham: Springer International Publishing, 2017: 506- 517.
[8] LongJ, ShelhamerE, DarrellT. Fully convolutional networks for semantic segmentation[C]∥2015 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), June 7-12, 2015, Boston, MA, USA. New York: IEEE, 2015: 3431- 3440.
[9] RonnebergerO, FischerP, BroxT. U-net: convolutional networks for biomedical image segmentation[M] ∥Lecture Notes in Computer Science. Cham: Springer International Publishing, 2015: 234- 241.
[10] He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition[C]∥2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), June 27-30, 2016, Las Vegas, NV, USA. New York: IEEE, 2016: 770- 778.
[11] HuangG, SunY, LiuZ, et al.Deep networks with stochastic depth[M] ∥Computer Vision - ECCV 2016. Cham: Springer International Publishing, 2016: 646- 661.
[12] Zeiler MD, KrishnanD, Taylor GW, et al. Deconvolutional networks[C]∥2010 IEEE Computer Society Conference on Computer Vision and Pattern Recognition, June 13-18, 2010, San Francisco, CA, USA. New York: IEEE, 2010: 2528- 2535.
[13] OdenaA, DumoulinV, Olah C.Deconvolution and checkerboard artifacts[J/OL].Distill, 2016[ 2019-10-28]. http: ∥distill. pub/2016/deconv-checkerboard.
[14] HuJ, ShenL, SunG. Squeeze-and-excitation networks[C]∥2018 IEEE/CVF Conference on Computer Vision and Pattern Recognition, June 18-23, 2018, Salt Lake City, UT. New York: IEEE, 2018: 7132- 7141.
[15] Menze B, Jakab A, Bauer S, et al. The multimodal brain tumor image segmentation benchmark (BRATS)[J]. IEEE Transactions on Medical Imaging, 2015, 34(10): 1993-2024.
[16] Bakas S, Akbari H, Sotiras A, et al. Advancing the cancer genome atlas glioma MRI collections with expert segmentation labels and radiomic features[J]. Scientific Data, 2017, 4: 170117.
[17] BakasS, AkbariH, SotirasA, et al. Segmentation labels and radiomic features for the pre-operative scans of the TCGA-GBM collection[EB/OL]. [2019-11-30]. https: ∥doi. org/10. 7937/K9/TCIA.2017. KLXWJJ1Q.
[18] BakasS, AkbariH, SotirasA, et al. and radiomic features for the pre-operative scans of the TCGA-LGG collection[EB/OL]. [2019-11-30]. https: ∥doi. org/10.7937/K9/TCIA. 2017. GJQ7R0EF.
[19] Tustison N J, Gee J C. N4ITK: Nick's N3 ITK implementation for MRI bias field correction[J]. Insight Journal, 2009: 1-8.
[20] Sudre CH, Li WQ, VercauterenT, et al. Generalised dice overlap as a deep learning loss function for highly unbalanced segmentations[C]∥Deep learning in medical image analysis and multimodal learning for clinical decision support. Cham: Springer, 2017: 240- 248.
[21] IsenseeF, KickingerederP, WickW, et al.Brain tumor segmentation and radiomics survival prediction: contribution to the BRATS 2017 challenge[M] ∥Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Cham: Springer International Publishing, 2018: 287- 297.
[22] PuchS, SánchezI, HernándezA, et al. Global planar convolutions for improved context aggregation in brain tumor segmentation[C]∥ Crimi A, Bakas S, Kuijf H, et al. International MICCAI Brainlesion Workshop. Cham: Springer, 2019, 11384: 393- 405.
艾玲梅, 李天东, 廖福元, 石康珍. 基于注意力U-Net的脑肿瘤磁共振图像分割[J]. 激光与光电子学进展, 2020, 57(14): 141030. Lingmei Ai, Tiandong Li, Fuyuan Liao, Kangzhen Shi. Magnetic Resonance Brain Tumor Image Segmentation Based on Attention U-Net[J]. Laser & Optoelectronics Progress, 2020, 57(14): 141030.






