基于混合损失函数的改进型U-Net肝部医学影像分割方法  下载: 2361次
下载: 2361次
1 引言
肝癌是一种发生在人体肝脏部位的恶性肿瘤,对肝癌进行早期诊断可以显著降低患者死亡率[1]。在对肝病进行精确诊断和治疗中,肝部医学影像分割是目前研究的热点之一,由于肿瘤的形态和外观比较复杂,并且人工标注工作量过大,其在医学图像处理领域仍然是一项比较艰巨的任务[2]。因此探索一种具有较高准确率的自动分割方法对临床诊断有着重要意义。
目前,大多数关于肝部医学图像的分割方法分为人工分割、半自动分割和自动分割。人工分割时需要专家依靠经验进行手动标注,不仅耗时,而且存在肿瘤边缘特征标注不明显的问题;半自动分割方法简单快速,但分割结果依赖于初始的设置参数,其算法的主观性较大;自动分割方法一般采用全卷积神经网络,在肝部医学图像分割中取得了很大进展。Vorontsov等[3]提出了一种对肝脏和肝脏病变区域进行联合分割的模型,该模型包括两个完全卷积网络(FCN),一个负责输出图片的感兴趣区域,另一个负责检测感兴趣区域并对其进行分割。类似于U-Net,该网络在内部使用跳跃连接,经过端到端训练对二维(2D)轴向切片进行分割,但在分割过程中忽略了Z轴上的低层信息和高层信息,分割结果不够精细。Chlebus等[4]开发了一种基于2D全卷积神经网络和对象的后处理分割方法。该方法采用类似于U-Net的网络结构,使其可以在四个分辨率级别上进行工作,并允许学习局部特征和全局特征,通过添加短跳跃连接来合理更新参数并加快训练速度,同时添加Dropout层防止过拟合,但对输出结果只进行了阈值处理,没有考虑复杂的图像细节。Li等[5]提出了一种新颖的混合紧密连接型U-Net,它由2D DenseUNet和3D DenseUNet组成,2D DenseUNet用于有效地提取切片内特征,而3D DenseUNet则对低层特征和高层特征进行分层聚合,用于肝脏和肿瘤的分割。通过混合的特征融合层,连接型U-Net可以共同优化片内表示和片间特征,但其模型不易训练,且对硬件要求较高。
针对上述方法的不足,本文提出一种改进的U-Net肝部医学图像分割方法,并引入改进的残差模块对边界细节进行优化,同时利用混合损失函数获得具有高置信度的清晰边界,以提高分割精确度。
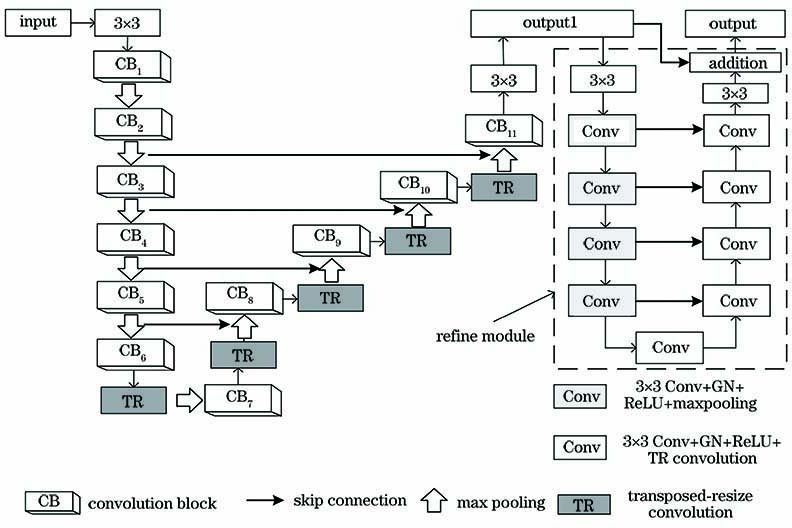
2 改进的网络结构
受U-Net结构和残差优化模块的启发,提出一种基于混合损失函数的改进型U-Net结构,如
2.1 改进的U-Net
U-Net是用于医学影像语义分割的U形全卷积神经网络[6-8],在网络内部将卷积层代替全连接层实现图像的端到端训练输出,并且能够结合底层信息和高层信息减小特征提取时的损失,是一种编码-解码结构[9]。U-Net的编码路径为首先将输入图像编码为特征向量,然后通过解码路径将其解码为输出分割图。由于上采样过程中提取到的高层特征非常抽象,因此网络利用跳跃连接将下采样过程中具有较高分辨率的低层特征图连接起来,这有助于通过编码路径恢复损失的信息。但是传统U-Net的网络层数过少,可学习的参数量过多,并且在上采样过程中会损失较多语义特征,难以满足日益复杂的诊断需求,因此需要对网络结构进行改进。
2.2 优化模块
优化模块通常被设计为残差模块,通过学习目标分割图像和专家标注图像之间的残差来精炼初始分割图。一般的初始分割结果存在边界细节丢失和预测区域概率不准确等问题,需要对其进行进一步优化处理。
目前对边界细节进行优化的残差模块通常为金字塔池化模块。该模块将其中的三级信息串联在一起,使用具有不同内核大小的空洞卷积捕获上下文的各级特征,并将所有残差连接在一起进行输出,这有助于合并在主网络层被编码的高级特征和在调整网络中被编码的低级特征,从而生成更精细的边界细节,其结构如
为了完善初始分割图中的边界缺陷,在改进的U-Net后添加一种新型的残差优化模块,其类似于残差编码-解码结构,如

图 3. 网络结构的残差优化模块。(a)传统残差优化模块;(b)改进的残差优化模块
Fig. 3. Residual refine module of network structure. (a) General residual refine module; (b) improved residual refine module
2.3 混合损失函数
为了获得高置信度分割图像的清晰边界,网络采用一种混合损失函数以减小分割图像和标注图像之间的差距,其定义为
式中:
BCE损失函数是二进制分类和分割中使用最广泛的损失函数,定义为
式中:GT(a,b)为像素(a,b)的专家标注标签;SEG(a,b)为成为分割物体的预测概率。BCE损失函数是逐像素的,它不考虑邻域的标签,并且对分割像素和背景像素均进行加权,有助于损失函数的收敛。
IOU通常用于目标检测和图像分割的标准评估。最近,它被用于网络的损失函数中来反映初始分割图与手动标注图之间的差距,表达式为
SSIM可以用于评估图像质量,可以获得分割图像的结构信息,因此,如果将其整合到网络的损失函数中,可获得标注图像的结构信息。令两张图片的对应像素点分别表示为x和y,其中x={xj:j=1,…,N2}和y={yj:j=1,…,N2}分别表示分割图片和专家标注图片对应区域的像素值,图片尺寸为N×N,x和y的SSIM定义为
式中:μx和μy为x和y的均值;σx和σy为x和y的标准差;σxy为协方差;C1设定为0.022,C2设定为0.042,防止分母为0。SSIM损失函数考虑了每个像素的局部邻域,可以将较高的权重分配给边界。在训练开始时,沿边界位置的损失最大,因此使用SSIM损失函数有助于优化边界特征的提取。
当结合这三个损耗时,BCE损失函数可使所有像素保持平滑的梯度,IOU损失函数更加关注相似度,SSIM损失函数则通过边界附近的较大损耗来对分割细节进行优化,提高图像的分割精度。
3 实验设置
3.1 数据集与数据预处理
实验数据采用了CodaLab组织提供的公共肝肿瘤数据集Liver Tumor Segmentation(LITS),其由4名放射科医生进行手动标注。由于提前测试了该网络对肝脏图像和肝肿瘤图像的分割精度,因此训练集和测试集均被分为两部分。肝脏部分的数据集包含18298张大小为512×512的图片,其中14198张图片作为训练集,4100张图片作为测试集;肝肿瘤数据集则在肝脏数据集上进行了进一步筛选,其训练集图片数量为3624,测试集图片数量为918。考虑到原始图像尺寸相比肝肿瘤图像太大,可能不利于训练,因此将包含肿瘤部分的原始图像重新调整为128×128像素大小作为网络输入。所有数据均通过随机裁剪、缩放及旋转等方式进行数据增强,以提高训练精度。
3.2 实验评估
为了评估所提方法的性能,使用图像分割常用的标准性能指标,包括体积重叠误差(VOE)、相对体积误差(RVD)、敏感度(SEN)、Dice系数及Jaccard系数。计算方式分别为
1) Dice系数的计算公式为
2) SEN的计算公式为
3) VOE的计算公式为
4) RVD的计算公式为
5) Jaccard系数的计算公式为
式中:SEG和GT分别为网络分割图像和专家标注图像。Dice系数、Jaccard系数和SEN接近1时,表示分割结果更接近专家标注图;RVD和VOE接近0时,表示分割误差较小或基本不存在分割误差。其中Dice系数用于表示网络分割图与专家标注图之间的相似程度,是一种十分重要的分割图像评价系数。
4 实验结果与分析
4.1 实验环境和参数设置
该网络框架基于公共框架Pytorch 0.4.0,训练和测试时均使用具有Intel Core i5-9400F 2.9 GHz CPU(具有16GB 内存)和GTX 2080 GPU(具有8GB显存)的计算机,在CUDA 10.0架构平台上进行并行计算,并调用CuDNN 7.5进行运算加速。所提网络采用Adam优化算法,其超参数设置为默认值,其中初始学习率设为10-3,矩估计的指数衰减速率ρ1和ρ2分别为0.9和0.999,用于数值稳定的小常数为1×10-8,权重衰减为0,并使用Dropout策略防止网络过拟合,设置其值为0.5。在对肝脏图像进行分割时,通过多次实验将损失函数的超参数分别设置为∂1=0.7,∂2=0.2,∂3=0.1,batch_size设置为4;在对肝肿瘤图像进行分割时,其初始学习率设置为5×10-4,通过多次实验将损失函数的超参数设置为∂1=0.5,∂2=0.3,∂3=0.2,其余参数设置不变。由于肝脏样本数量和肝肿瘤样本数量存在较大差距,所提网络在对肝脏进行分割训练时设置epoch为200,对肝肿瘤进行分割训练时设置epoch为800。相比肝脏,肝肿瘤的形状受呼吸、运动等外界影响较为严重,其形状变化非常不规律,导致难以对其分割准确。在对肝肿瘤进行分割训练时,当经过200000次迭代后,模型总体上趋于稳定状态;对肝脏进行分割训练时,模型趋于稳定时的迭代次数约为100000。

图 4. 改进U-Net的训练误差和测试准确率。(a)肝肿瘤分割;(b)肝脏分割
Fig. 4. Training error and test accuracy of improved U-Net. (a) Liver tumor segmentation; (b) liver segmentation
4.2 肝脏图像分割
为了证明所提网络对肝脏分割具有较优结果,利用U-Net和FCN-8s网络同时对该数据集进行分割处理。

图 5. 不同网络对肝脏图像分割的结果
Fig. 5. Segmentation results of liver images obtained by different networks
为了更加直观地对分割结果进行分析,对卷积块下的卷积层数进行了不同数量的设定,并结合U-Net与FCN-8s的分割结果,绘制了Dice系数箱型图,如
表 1. 不同网络对肝脏图像进行分割时的性能对比
Table 1. Performance comparison of different networks for liver image segmentation
|
4.3 肝肿瘤图像分割

图 7. 不同网络对肝肿瘤图像分割的结果
Fig. 7. Segmentation results of liver tumor images obtained by different networks
从
表 2. 不同网络对肝肿瘤图像进行分割时的性能对比
Table 2. Performance comparison of different networks for liver tumor image segmentation
|
4.4 其他医学图像分割
为了证明所提网络对其他医学图像的分割结果,利用U-Net和FCN-8s网络同时对肺部CT图像进行分割处理。数据集为LIDC-IDRI肺结节数据集,该数据集记录了对肺癌的筛查、诊断及标记的病变信息,共包含1018个病例。考虑到肺结节大小的差异,给出了大结节(尺寸大于等于3 mm)和小结节(尺寸小于3 mm)分割结果,如
从
表 3. 不同网络的性能对比
Table 3. Performance comparison of different networks
|
5 结论
针对目前肝部医学图像自动分割方法的不足,提出了一种改进的分割网络用于分割肝部医学图像。为了提高分割精度,对原始U-Net进行了改进,通过在上采样阶段加入池化层复制对应下采样阶段的特征图,利用转置缩放卷积和组归一化减小信息损失;同时采用了新型残差优化模块精炼初始分割图像,并通过混合损失函数赋予边界像素较高权重,提高了分割图像边界的清晰度。实验结果表明,在肝脏分割和肝肿瘤分割实验中,所提网络在精度和稳定性上均大幅优于传统深度学习方法,并且与目前先进分割网络相比,其性能也互有优劣,证明改进的网络结构、残差优化模块及混合损失函数均可以提升网络分割性能。对于其他医学图像的分割,所提网络结构虽然不能与先进网络相比,但依然可以证明残差优化模块结合混合损失函数对分割网络性能的提升潜力,表明所提网络可以推广到所有U形网络和FCN中,并在未来得到更好的发展。
[3] VorontsovE, TangA, PalC, et al. Liver lesion segmentation informed by joint liver segmentation[C]//2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018), April 4-7, 2018, Washington, DC, USA. New York: IEEE, 2018: 1332- 1335.
[4] Chlebus G, Schenk A, Moltz J H, et al. Automatic liver tumor segmentation in CT with fully convolutional neural networks and object-based postprocessing[J]. Scientific Reports, 2018, 8(1): 15497.
[5] Li X M, Chen H, Qi X J, et al. H-DenseUNet: hybrid densely connected UNet for liver and tumor segmentation from CT volumes[J]. IEEE Transactions on Medical Imaging, 2018, 37(12): 2663-2674.
[6] RonnebergerO, FischerP, BroxT. U-Net: convolutional networks for biomedical image segmentation[M] //Navab N, Hornegger J, Wells W M, et al. Medical image computing and computer-assisted intervention-MICCAI 2015. Lecture notes in computer science. Cham: Springer, 2015, 9351: 234- 241.
[7] 苗光, 李朝锋. 二维和三维卷积神经网络相结合的CT图像肺结节检测方法[J]. 激光与光电子学进展, 2018, 55(5): 051006.
[8] 马其鹏, 谢林柏, 彭力. 一种基于改进的卷积神经网络在医学图像分割上的应用[J]. 激光与光电子学进展, 2020, 57(14): 141020.
[9] 任欣磊, 王阳萍, 杨景玉, 等. 基于改进U-net的遥感影像建筑物提取[J]. 激光与光电子学进展, 2019, 56(22): 222801.
[10] SzegedyC, VanhouckeV, IoffeS, et al. Rethinking the inception architecture for computer vision[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), June 27-30, 2016, Las Vegas, NV, USA. New York: IEEE, 2016: 2818- 2826.
[12] MáttyusG, Luo WJ, UrtasunR. DeepRoadMapper: extracting road topology from aerial images[C]//2017 IEEE International Conference on Computer Vision (ICCV), October 22-29, 2017, Venice, Italy. New York: IEEE, 2017: 3458- 3466.
[13] WangZ, Simoncelli EP, Bovik AC. Multiscale structural similarity for image quality assessment[C]//The Thrity-Seventh Asilomar Conference on Signals, November 9-12, 2003, Pacific Grove, CA, USA. New York: IEEE, 2003: 1398- 1402.
[14] Christ P F. Elshaer M E A, Ettlinger F, et al. Automatic liver and lesion segmentation in CT using cascaded fully convolutional neural networks and 3D conditional random fields[J]. Medical Image Computing and Computer, 2016: 415-423.
[15] Li S. Tso G K F, He K J. Bottleneck feature supervised U-Net for pixel-wise liver and tumor segmentation[J]. Expert Systems with Applications, 2020, 145: 113131.
[16] Nasiri N, Foruzan A H, Chen Y W. Integration of a knowledge-based constraint into generative models with applications in semi-automatic segmentation of liver tumors[J]. Biomedical Signal Processing and Control, 2020, 57: 101725.
[18] Foruzan A H, Chen Y W. Improved segmentation of low-contrast lesions using sigmoid edge model[J]. International Journal of Computer Assisted Radiology and Surgery, 2016, 11(7): 1267-1283.
[20] Cao H C, Liu H, Song E M, et al. Dual-branch residual network for lung nodule segmentation[J]. Applied Soft Computing, 2020, 86: 105934.
[21] Liu H, Cao H C, Song E M, et al. A cascaded dual-pathway residual network for lung nodule segmentation in CT images[J]. Physica Medica, 2019, 63: 112-121.
Article Outline
黄泳嘉, 史再峰, 王仲琦, 王哲. 基于混合损失函数的改进型U-Net肝部医学影像分割方法[J]. 激光与光电子学进展, 2020, 57(22): 221003. Yongjia Huang, Zaifeng Shi, Zhongqi Wang, Zhe Wang. Improved U-Net Based on Mixed Loss Function for Liver Medical Image Segmentation[J]. Laser & Optoelectronics Progress, 2020, 57(22): 221003.