二维和三维卷积神经网络相结合的CT图像肺结节检测方法  下载: 2880次
下载: 2880次
ing at the problems that traditional lung nodules detection methods can only get low sensitivities and a lot of false positives, this paper presents a retrieval method for lung nodules CT image based on end-to-end two-dimensional full convolution object recognition network (2D FCN) and three-dimensional target classification convolution neural network (3D CNN). Firstly, the method builds the 2D CNN for candidate selection to detect and locate the suspected regions on axial slices, and outputs an image that is the same size as the original image and is marked. Secondly, the three-dimensional patches of each candidate are extracted to train the 3D CNN. Finally, the trained 3D model is used to classify the false positive nodules. Experimental results on the LIDC-IDRI dataset show that the proposed method can achieve the recall rate of nodules of 98.2% at 36.2 false positives per scan. In the false positive reduction, the method respectively achieves high detection sensitivities of 87.3% and 97.0% at 1 and 4 false positives per scan. Experimental results on the LIDC-IDRI dataset show that the proposed method is highly suited to be used for lung nodules detection, achieves high recall rate and accuracy and outperforms the current reported method. Meanwhile, the proposed framework is general and can be easily extended to many other 3D object detection tasks from volumetric medical images, and it has an important application value in clinical practice with the aid of radiologists and surgeons.
1 引言
肺癌是目前对人类健康和生命威胁最大的恶性肿瘤之一[1]。研究发现,如果能及早发现和治疗,肺癌患者的5年存活率将提高50%左右。肺癌早期有相当部分表现为结节,肺部结节为肺内呈圆形或不规则的病灶,其中约34%为肺癌,其余为良性病变。所以肺部结节的检测对发现早期肺癌具有极大的重要性。
低剂量胸部电子计算机断层扫描(CT)是目前公认的检测肺部结节最可靠的方法,但CT检测结果中依然存在大量的假阳性。仅依靠人工对肺结节进行查找和诊断,不仅工作量大,而且容易出错,因此对组织病理学图片进行自动检测和分析是目前非常热门的一个研究方向。目前,对病理学图片进行自动检测和分析的常用方法是传统的机器学习方法[2-5],如阈值分割法、支持向量机(SVM)方法等。阈值分割法可对肺部不同区域的灰度值进行层层分割,如可对血管、气泡、骨架等进行分离,该方法比较简单,但肺结节的灰度值与血管、肺边缘、气泡等的灰度值非常相似,会导致模型生成大量的假结节。SVM方法需要人工提取并计算结节特征,如纹理、大小、边缘等特征。这些算法的准确度依赖于基于人工设计的特征提取方法,如果特征选择不好,就会很容易遗漏结节信息,这将直接影响结节检测的准确率。
深度学习是机器学习的一个重要分支,近几年得到了迅猛发展。大量的可用数据和高效的图形处理器(GPU)计算能力使得卷积神经网络(CNN)在图像领域中取得了不错的成就[6-13],并且在医疗图像上有了一定应用[7-13],可对脑部、细胞、肺部组织等进行分割与识别。卷积神经网络以一种端到端的形式识别特征,与传统的机器学习方法相比,其优势在于特征提取不需要人工参与,各层的特征提取都是在通用的学习过程中进行,从数据中学习得到。
本文提出了一种基于深度卷积神经网络的肺结节检测方法,通过深度卷积神经网络对肺结节进行自动的特征学习和检测。每一副CT图像约有500张序列切片,每一张切片大小为512 pixel×512 pixel,最大的结节尺寸约为34 mm,最小的结节尺寸约为4 mm,此次检测为小目标检测,且数据量较大。3D全卷积模型对小目标的检测效果并不好,主要用于大目标的定位与分割,同时数据量过大会导致机器不能以批量样本的形式将多副完整的CT图像送入模型进行训练。本文采用2D全卷积神经网络对CT图像中每一张切片的候选结节进行初步检测,之后合并切片间的结节,最后在保证高检出率的同时生成少量的候选结节[4-5]。候选结节生成后在CT立体图像上构建3D卷积神经网络框架,在候选结节中抽取3D样本块进行假阳性去除,经过3D样本训练后模型能更加充分地提取结节中原有的空间信息,同时生成更具有代表性的特征。与传统方法相比,所提方法可大大提高肺部结节的检出率和准确率,对辅助医师诊治有着重要的应用价值。
2 二维和三维卷积神经网络相结合的肺结节检测
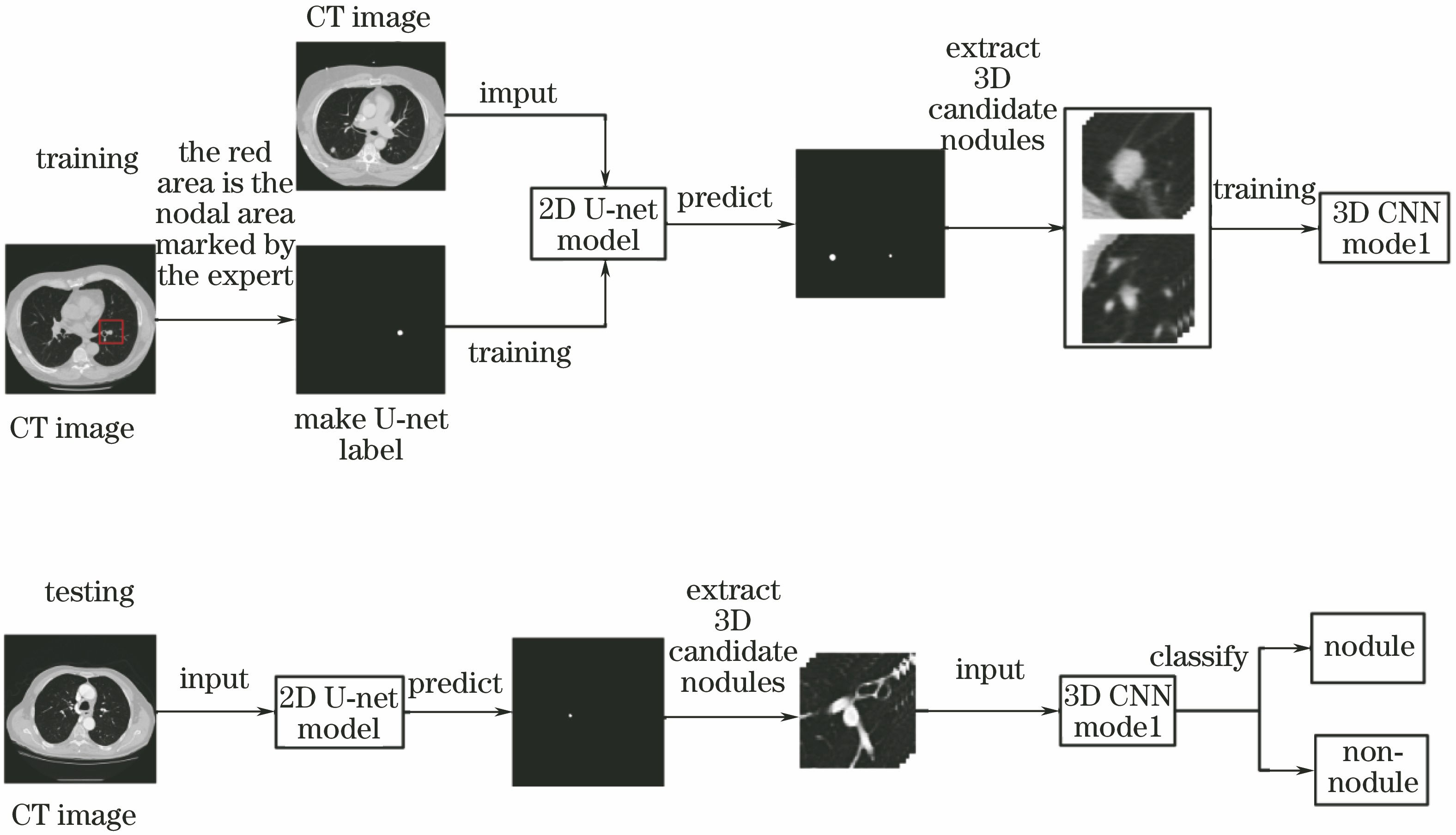
系统的功能模块由两部分组成:1) 改进的2D U-net候选结节检测;2) 3D卷积神经网络假阳性去除。具体方法是先采用改进的全卷积神经网络U-net[7]以一种端到端的形式快速完成候选结节对象的识别与定位,然后在CT立体图像上对所有的候选结节抽取3D样本,构建并训练3D卷积神经网络模型,最终使用模型对每一个候选结节做二分类,得到结节的一个数值描述(概率值)。系统的实现分为训练和测试两个阶段,
2.1 改进的2D U-net候选结节检测
候选结节检测算法在整个检测方案中扮演着重要角色,它决定了结节的召回率。理想的候选结节检测方法应该检测出所有的真结节,但是由于结节检测为小目标对象识别,并且结节形态各异,所以要想达到100%的召回率较困难。本研究采用改进的全卷积神经网络U-net作为候选结节的检测方法,全卷积网络是一种端到端、像素到像素的对象识别与定位网络。它可以接受任意尺寸的输入图像,采用反卷积层对最后一个卷积层的特征映射图进行上采样,使它恢复到与输入图像相同的尺寸,从而可以对每个像素都产生一个预测,最后输出一张已标记好的图片,效率高且结果比较直观。参照原始的U-net模型在训练集上会出现过拟合的趋势,所以对U-net进行了一些改进。
2.1.1 改进U-net网络的结构
由于CT图像的大小为512 pixel×512 pixel,所以将原始网络的输入大小由572 pixel×572 pixel改为512 pixel×512 pixel。网络本身较深的层数会导致计算量过大,从而导致网络只能接受较小的批处理图片,原始的网络会出现不收敛的情况。所以在网络的收缩部分添加了Batch-Normalization[14],激活函数采用Leaky-ReL[15]代替原有的ReLU[16],最后加上空间dropout策略[17]。
2.1.2 U-net结果输出
在U-net的输出中候选结节将被抽取,一共生成62809个候选结节。对预测图采用二值化、去噪声处理,最后使用形态学方法得到中心坐标。
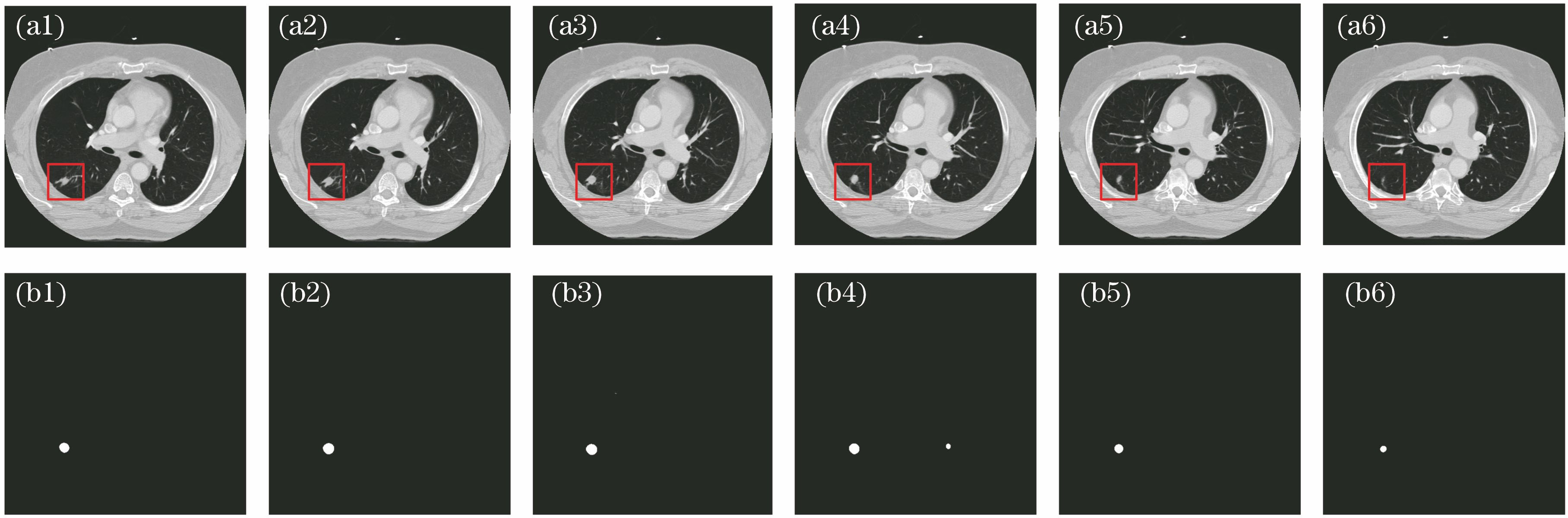
图 2. CT切片序列原图与U-net预测图。(a)原图;(b)预测图
Fig. 2. CT slice original images and U-net prediction images. (a) Original images; (b) prediction images
2.1.3 结节合并
U-net抽取候选结节后,多层切片之间的许多结节是需要合并的。首先采用距离合并,再采用结节合并。采用距离合并时,由于结节的抽取是基于2D的,因此合并切面之后有很多结节距离很近。为了减少这些候选结节,通过计算坐标之间的欧氏距离将其合并,然后将所有的合并结节坐标的均值作为新的坐标值。结节合并这一环节是基于真结节的距离进行合并的,在注释文件中有真结节的坐标以及半径注释。在真结节的半径内或距离结节中心坐标30 mm范围内将其合并,新的坐标采用均值坐标。
2.1.4 U-net模型评价指标--召回率
候选结节初步检测的标准由召回率来衡量,召回率先是候选结节的中心坐标与注释文件中真结节的真实坐标之间的距离,如果这个距离在真结节的半径之内,则认为检测到结节。之后统计候选结节的总数来计算召回率,召回率的计算公式为
式中:
2.2 3D卷积神经网络假阳性去除
在系统完成肺结节初步检测之后,检测范围得以缩小,但依然生成了较多的候选结节,之后需要对这些结节做假阳性去除处理。采用基于3D卷积神经网络的框架完成假阳性去除的任务。与传统的阈值分割法[18]、机器学习方法[19-20]和2D卷积神经网络模型[12-13]相比,3D卷积神经网络能够更加充分地提取结节原有的空间信息,同时可以学习到更具代表性的特征。这些特征对物体的大小、位置和方向等的敏感性更低,有助于识别性能的提高。系统首先在原有CT立体图像上抽取结节的立体块,然后通过构建的3D卷积神经网络网络结构更深入地学习结节潜在的特征。在3D图像的研究中,该框架能够更容易和更精确地探索检测类任务。
2.2.1 网络结构
3D卷积神经网络假阳性去除系统网络的结构如
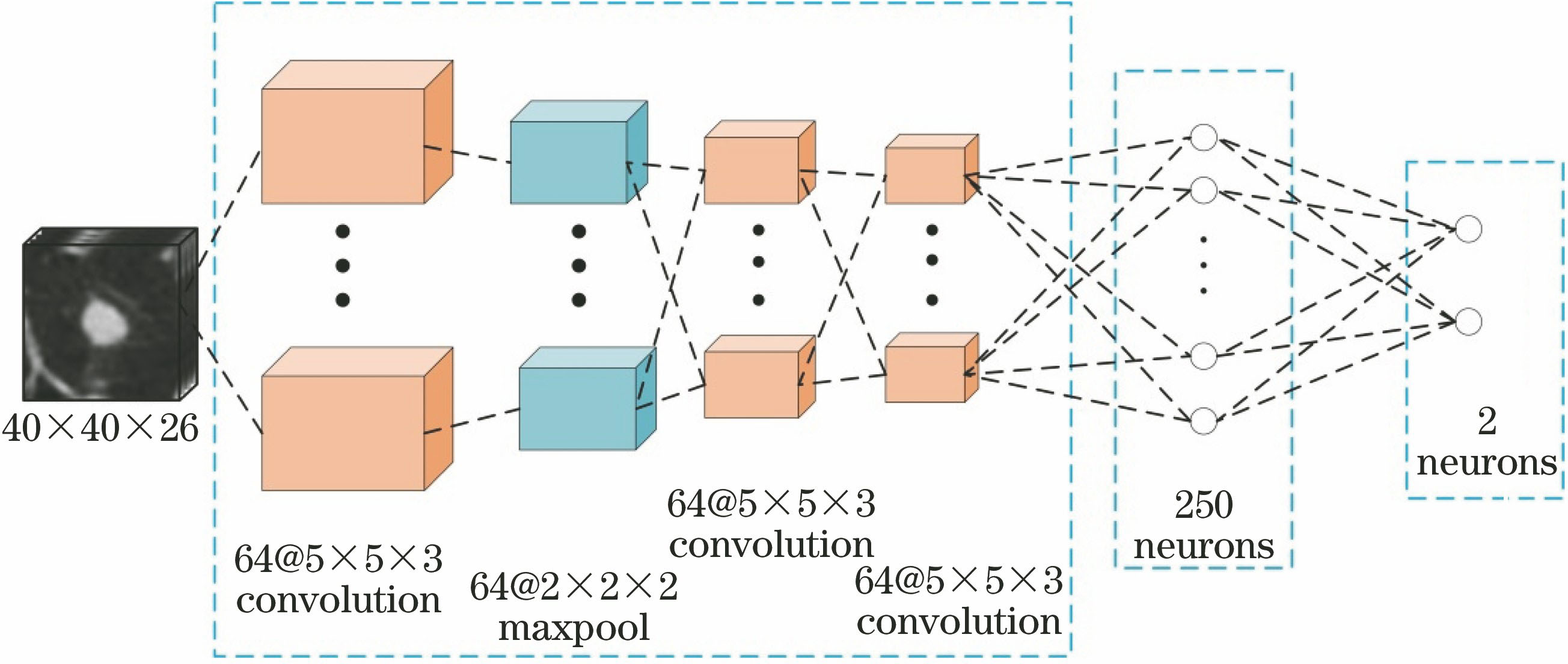
图 3. 3D卷积神经网络假阳性去除系统网络的结构
Fig. 3. Structure of 3D convolution neural network false positive removal system network
2.2.2 3D卷积神经网络的构造
一般的3D卷积神经网络由3D卷积层、3D池化层和全连接层抽取图像的特征,最后接一个softmax层得到一个分数[10-11]。每一层有许多通道,每个通道代表一种特征。对于3D卷积神经网络而言,卷积池化实际上是一个立体的3D特征块。与2D不同的是,3D的特征是以立体形式呈现的一组神经元。
3D卷积层:构造一个3D卷积层,首先要建立一系列的3D特征抽取块来扫描所有的输入,也就是3D卷积核。为了生成一个立体特征块,使用不同的3D卷积核来卷积不同的特征输入块,每一个特征块对应一个独立的卷积核;然后添加偏置项和一个非线性激活函数,最后对不同的3D卷积核的操作求和。3D卷积的公式为
式中:
3D池化层:在连续的卷积操作之后插入一个3D池化层。采样3D特征块是为了在保留原有特征信息的情况下,不断地通过降低图像维度后缩小特征图尺度来减少数据量的运算。假设第
全连接层:与卷积层相比,全连接层有更多的连接。每一个神经元将会和邻近层中所有的神经元连接,这些神经元会增强抽取特征的代表能力。要实现全连接层,首先需要将立体特征块拉成的一维向量与神经元做乘积运算,然后加上一个偏置项,最后应用一个非线性激活函数,即:
式中:
2.2.3 3D图像块的抽取
3 实验设计与结果分析
3.1 实验数据集
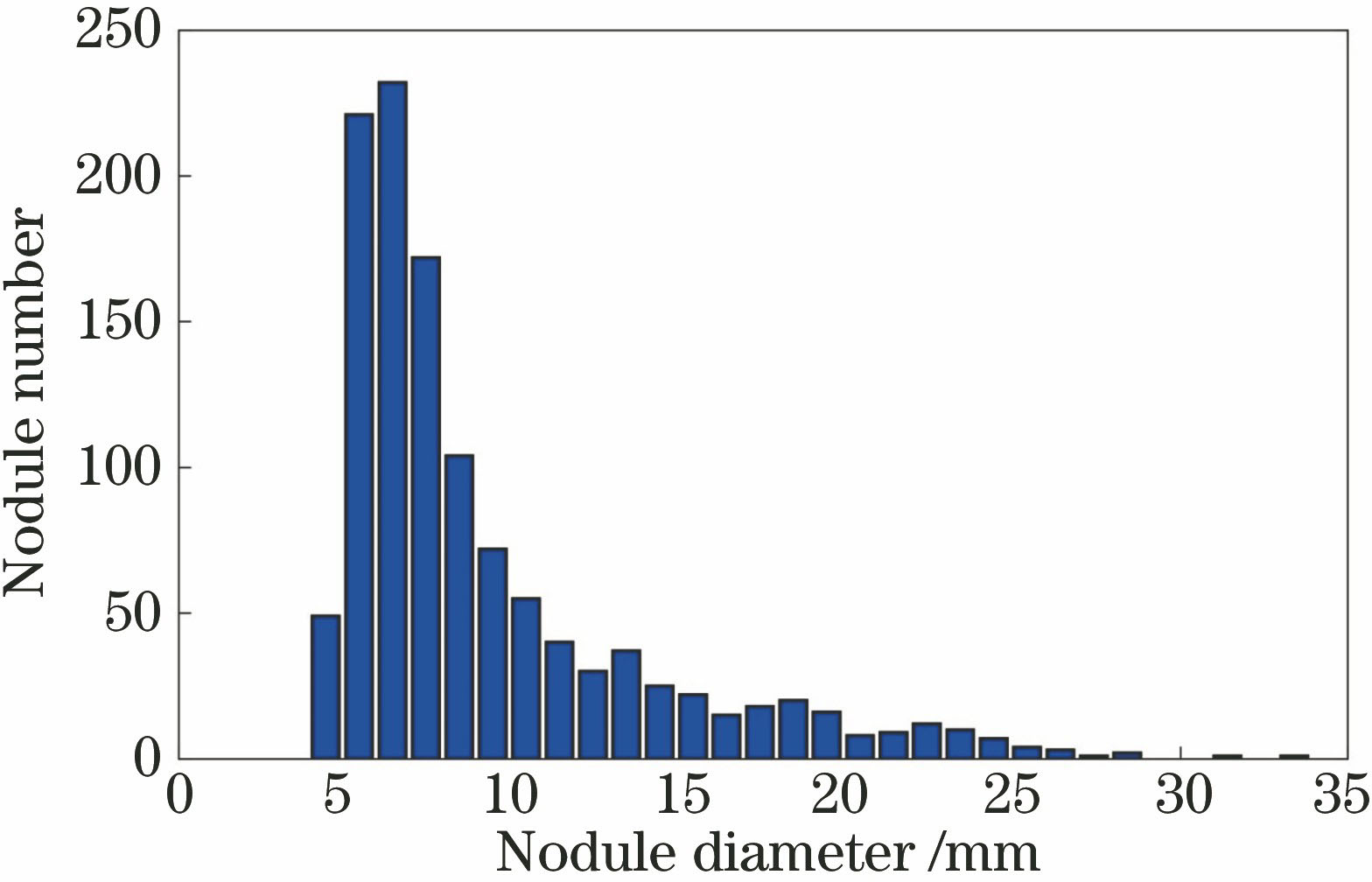
实验数据集取自公开的LIDC-IDRI数据库[21],数据库中包含1018张CT图像。这些CT由7个不同的机构制作而成。切片的厚度为0.6~5.0 mm,中间值为2.0 mm。参照标准由4位专家手工注释。为了保证注释的准确性,专家对每一幅CT图像审查两遍。第一遍判断肺结节的尺寸是小于3 mm还是大于或等于3 mm,然后手动分割出尺寸大于等于3 mm的结节。第二遍由4名放射科医师共同评审结节的注释,每位放射科医师都可以否定其他专家对结节的标注,然后进行重新标注。挑出尺寸大于3 mm并由3名或4名专家共同注释的结节。
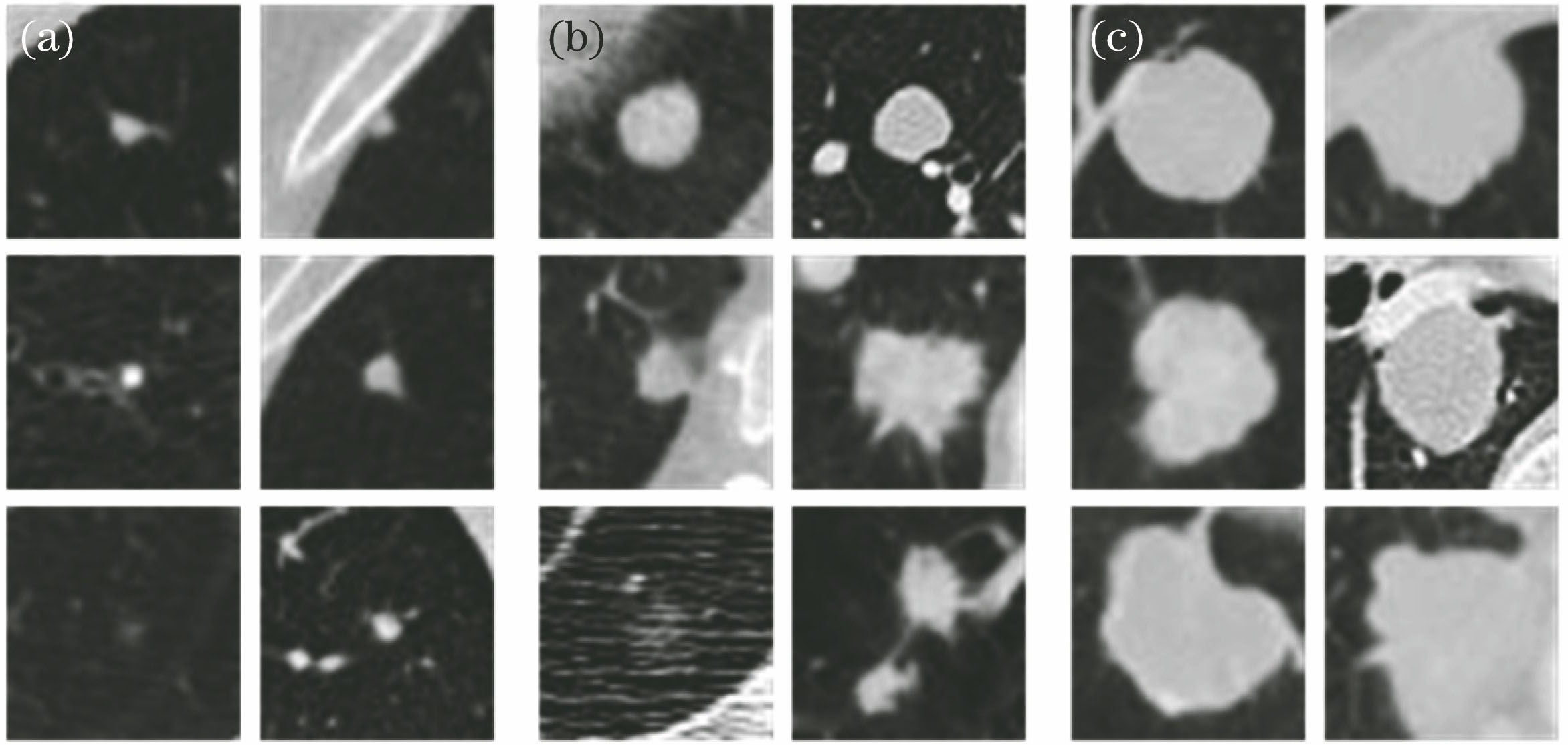
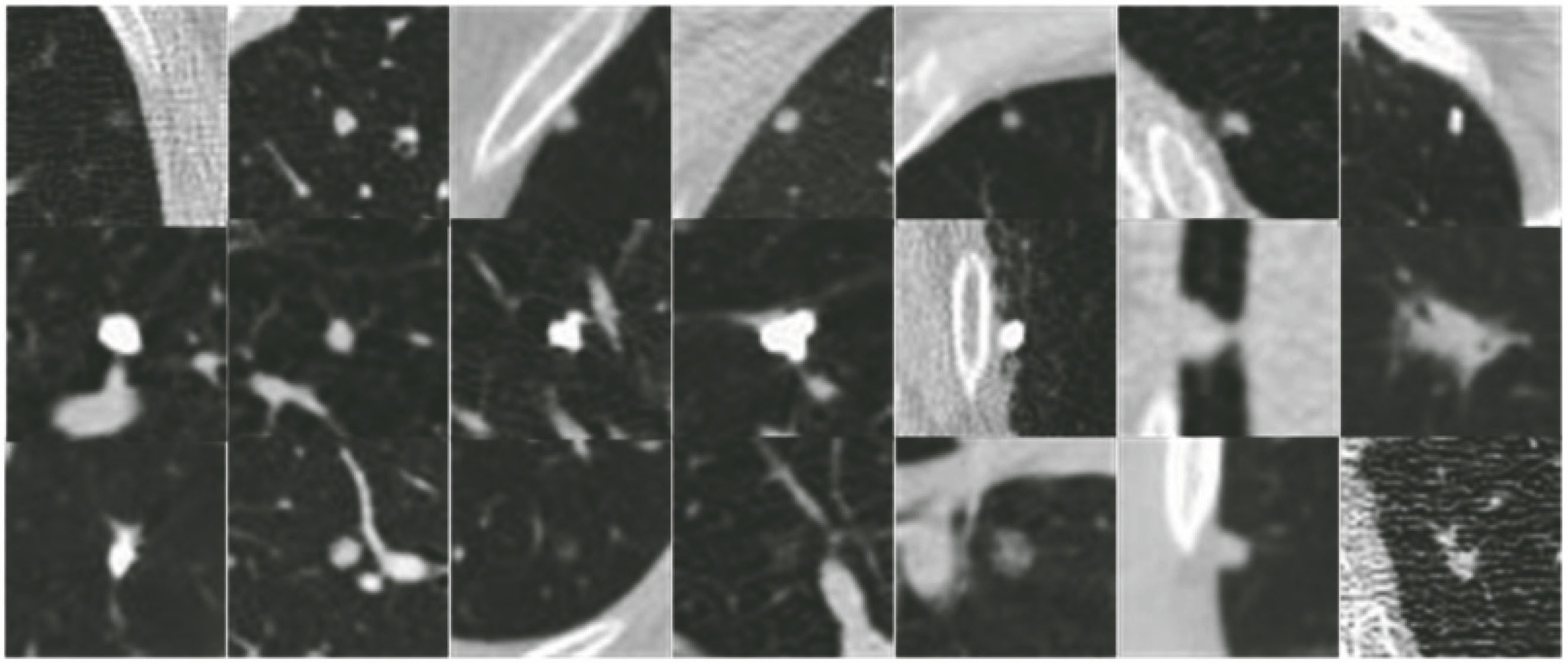
图 5. 数据库中不同尺寸结节的图像。(a)小结节;(b)中等结节;(c)大结节
Fig. 5. Images of nodules with different sizes in the database. (a) Small nodules; (b) middle nodules; (c) big nodules
3.2 模型训练方法
为了验证模型的性能,将数据划分为5份,采用5折交叉验证实验,4份用于训练,1份用于测试,直到所有的数据都被测试。针对3D模型训练,由于其候选结节中的阳性样本较少,为了保持样本的均衡,使用增援技术。首先将数据在(
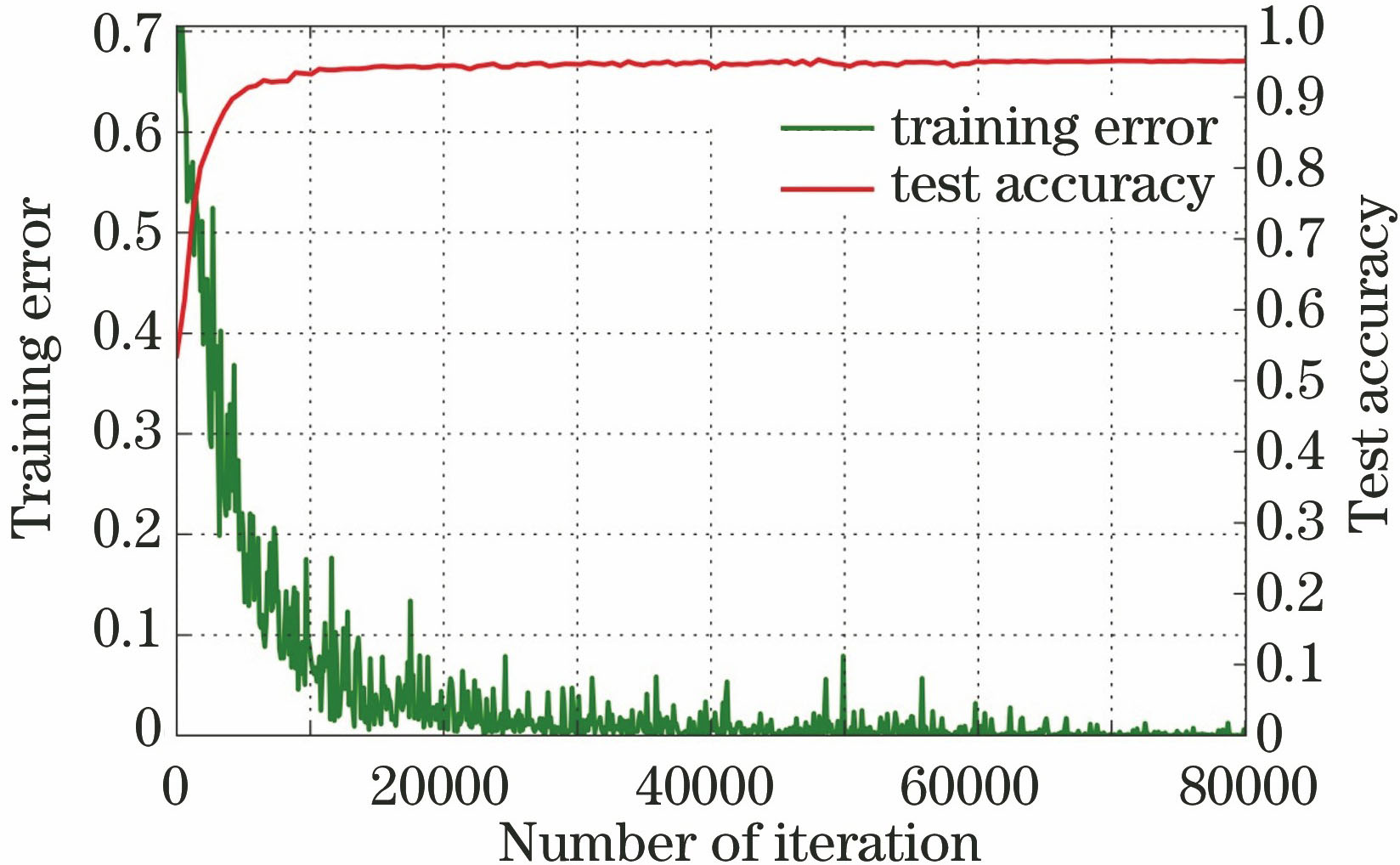
图 6. 3D卷积神经网络模型的训练误差和测试准确率曲线
Fig. 6. Training error and test accuracy curves of 3D convolution neural network model
3.3 实验结果与分析
采用改进的U-net全卷积网络模型作为候选结节的初步检测方法,最终生成62809个候选结节,合并后为35597个结节。在1186个真结节中检测到了1165个,召回率为98.2%,U-net表现出了出色的检测性能。之后提出3D卷积神经网络假阳性去除模型,通过对结节原有立体信息的特征学习来强化系统的检测性能。针对3D假阳性去除模型的验证,通过受试者特征(FROC)[24]曲线来定量分析检测系统对结节的检测性能,FROC曲线可以反映肺结节的检出率与平均每幅图像假阳性个数的关系。
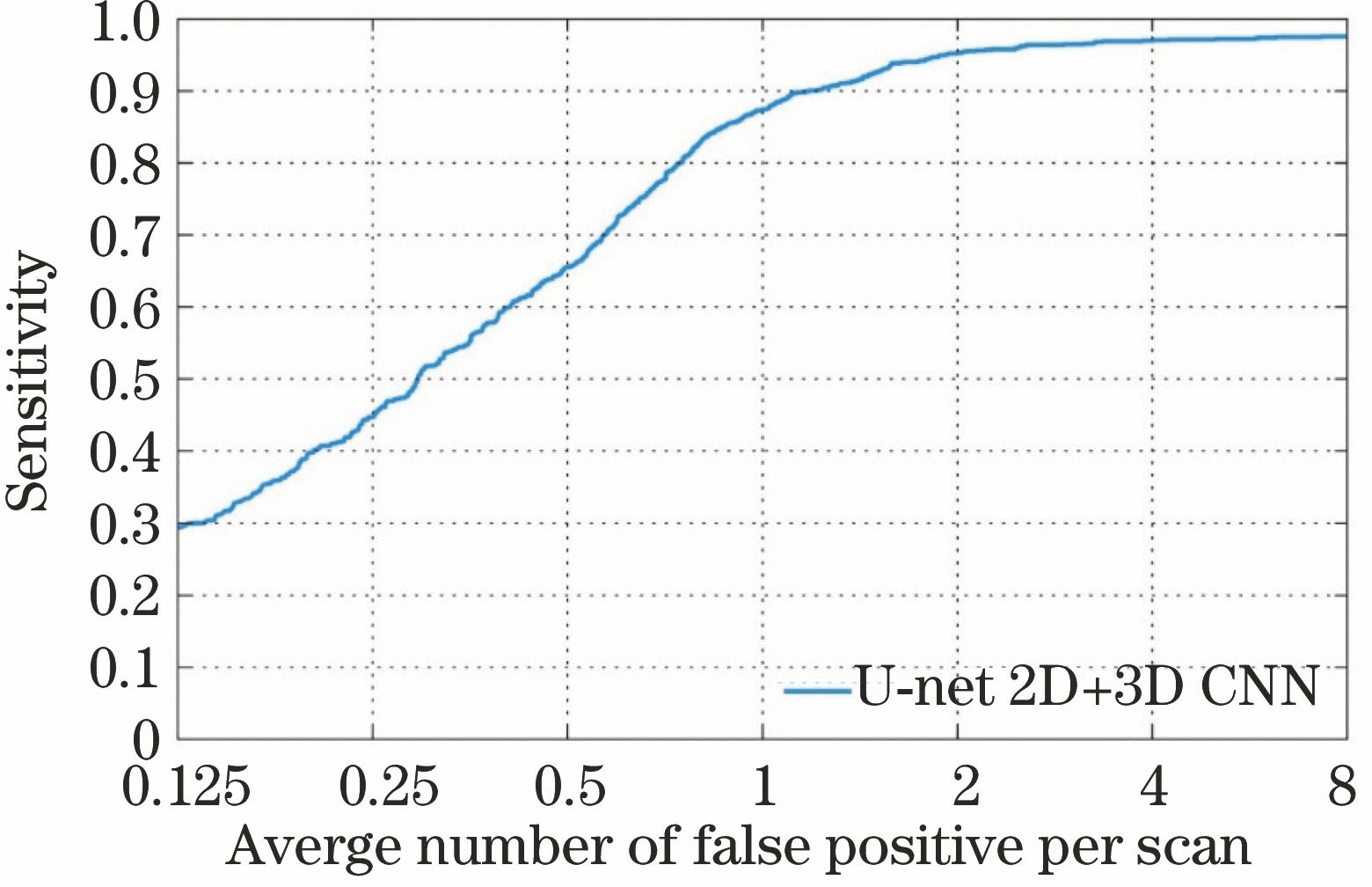
图 7. 平均每幅CT图像假阳性比例的准确率
Fig. 7. Accuracy of average number of false positives per CT image
3.3.1 模型尺度的选择对实验结果的影响
根据尺寸将结节分为大、中、小3类,如
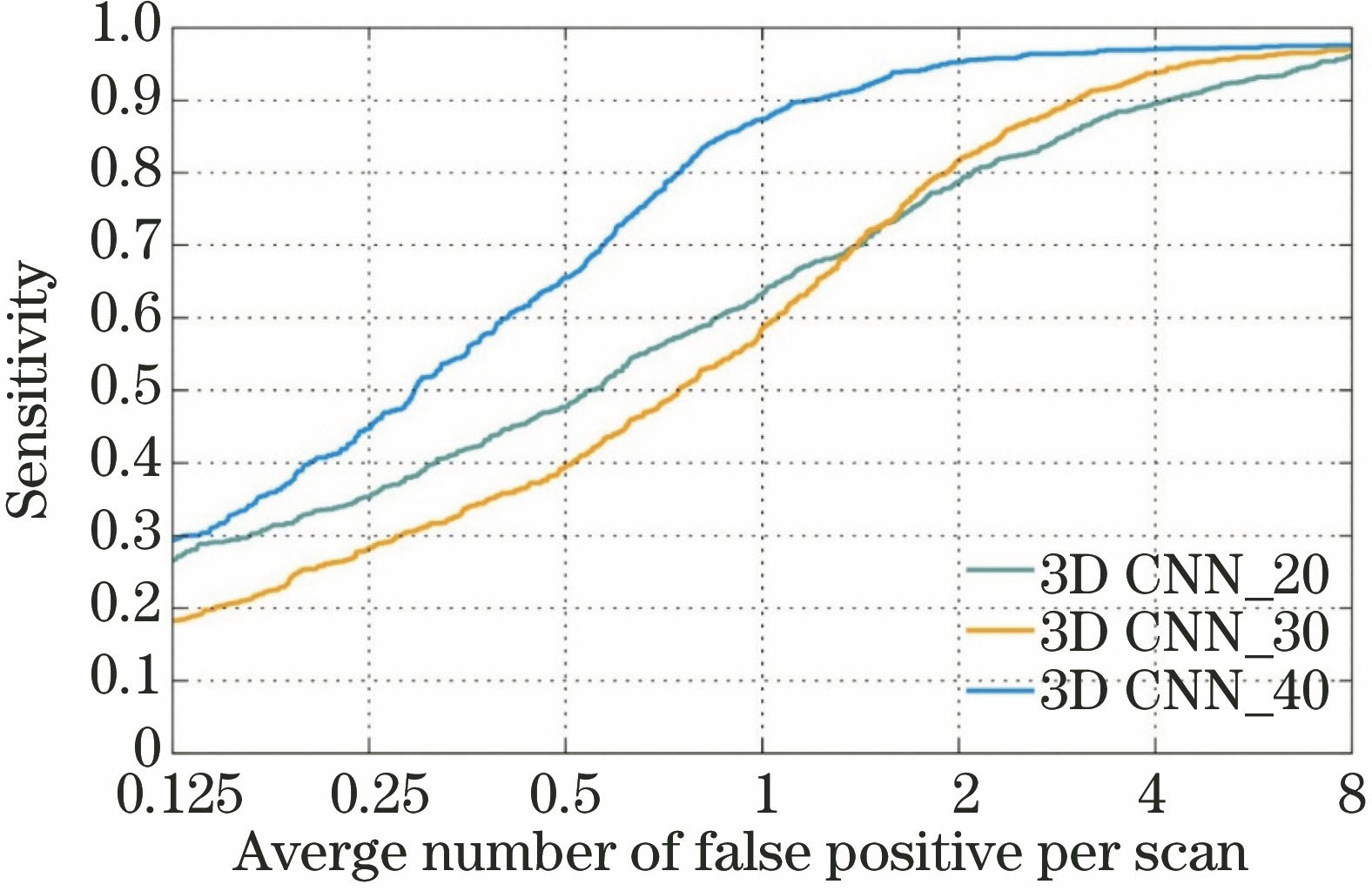
图 8. 输入图像块的尺度对实验结果的影响
Fig. 8. Effect of the dimensions of the input image block on the experimental results
3.3.2 网络模型对实验结果的影响
在假阳性去除任务中,测试了2D切面网络结构对实验结果的影响。2D模型对本研究中的3D模型进行了一些调整,主要将样本输入由3D图像块改为(
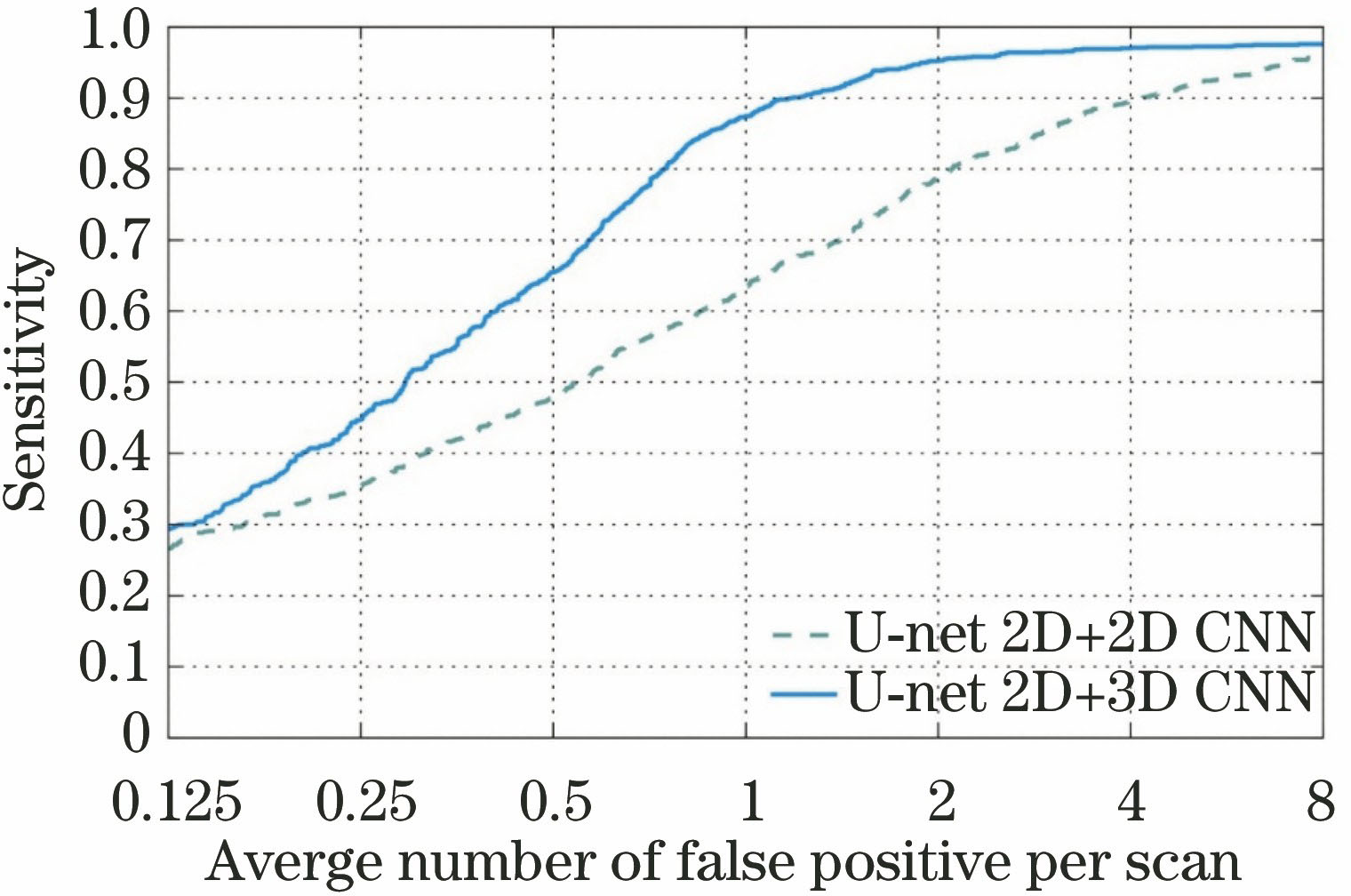
图 9. 网络模型的选择对实验结果的影响
Fig. 9. Effect of the selection of network model on experimental results
3.3.3 分析与对比
表 1. LIDC-IDRI数据库中肺结节的检测算法比较
Table 1. Comparison of detection algorithms of pulmonary nodules in LIDC-IDRI database
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
提出的基于2D对象识别与3D目标分类相结合的卷积神经网络肺结节检测方法的准确率均优于其他算法。该方法不需要人工干预,既不需要切取三维立体图像块多个方向的切面图像来保持原有的立体信息,也不需要进行特征选取,直接将原图像输入到全卷积网络中检测出候选结节,再将候选结节的3D样本块送入3D网络模型,充分提取结节的空间信息,利用深度学习发现图像块中的潜在特征,为图像块进行分类,检测出目标对象。所提方法的稳健性强,而且可以精确检测出CT图像中的结节。
在检测中有一些结节未被检出,
4 结论
研究了卷积神经网络在肺结节检测中的应用。使用改进的U-net全卷积分割网络作为结节的初步检测,达到了98.2%的召回率。提出的基于3D卷积神经网络的肺结节假阳性去除模型在假阳性为4时达到了97.0%的精度。基于LIDC-IDRI数据库的实验结果表明,3D卷积神经网络在不同大小和变异的立体医疗数据中获得了最优的分类精度,同时也获得了优于目前国内外其他方法的分类结果。在3D医疗图像的研究中,该模型能够更容易和更精确地执行检测类任务。
[1] Sluimer I, Schilham A, Prokop M, et al. Computer analysis of computed tomography scans of the lung: a survey[J]. IEEE Transactions on Medical Imaging, 2006, 25(4): 385-405.
[2] 高婷, 龚敬, 王远军, 等. 检测肺结节的3维自适应模板匹配[J]. 中国图象图形学报, 2014, 19(9): 1384-1391.
Gao T, Gong J, Wang Y J, et al. Three dimensional adaptive template matching algorithm for lung nodule detection[J]. Journal of Image and Graphics, 2014, 19(9): 1384-1391.
[3] Murphy K, Ginneken B V. Schilham A M R, et al. A large-scale evaluation of automatic pulmonary nodule detection in chest CT using local image features and k-nearest-neighbour classification[J]. Medical Image Analysis, 2009, 13(5): 757-770.
[4] Jacobs C. Rikxoort E M V, Twellmann T, et al. Automatic detection of subsolid pulmonary nodules in thoracic computed tomography images[J]. Medical Image Analysis, 2014, 18(2): 374-384.
[5] 陈胜, 李莉. 一种全新的基于胸片计算机辅助检测肺结节方案[J]. 电子学报, 2010, 38(5): 1211-1216.
Chen S, Li L. A new computer aided diagnostic scheme for lung nodule detection on chest radiograph[J]. Acta Electronica Sinica, 2010, 38(5): 1211-1216.
[6] 王民, 刘可心, 刘利, 等. 基于优化卷积神经网络的图像超分辨率重建[J]. 激光与光电子学进展, 2017, 54(11): 111005.
[7] Krizhevsky A, Sutskever I, Hinton G E. ImageNet classification with deep convolutional neural networks[J]. Communications of the ACM, 2017, 60(6): 84-90.
[8] RonnebergerO, FischerP, BroxT. U-Net: convolutional networks for biomedical image segmentation[C]. International Conference on Medical Image Computing and Computer-Assisted Intervention, [S.l.]: Springer, 2015: 234- 241.
[9] 黄鸿, 何凯, 郑新磊, 等. 基于深度学习的高光谱图像空-谱联合特征提取[J]. 激光与光电子学进展, 2017, 54(10): 101001.
[10] ChenH, QiX, YuL,et al. DCAN: deep contour-aware networks for accurate gland segmentation[C]. IEEE Conference on Computer Vision and Pattern Recognition, 2016: 16541200.
[11] Kamnitsas K, Ledig C. Newcombe V F J, et al. Efficient multiscale 3D CNN with fully connected CRF for accurate brain lesion segmentation[J]. Medical Image Analysis, 2017, 36: 61-78.
[12] Setio A A A, Ciompi F, Litjens G, et al. . Pulmonary nodule detection in CT images: false positive reduction using multi-view convolutional networks[J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1160-1169.
[13] van Ginneken Bv, Setio A AA, JacobsC, et al. Off-the-shelf convolutional neural network features for pulmonary nodule detection in computed tomography scans[C]. IEEE 12th International Symposium on Biomedical Imaging, 2015: 15309667.
[14] IoffeS, SzegedyC. Batch normalization: accelerating deep network training by reducing internal covariate shift[C]. International Conference on Machine Learning, 2015: 448- 456.
[15] HeK, ZhangX, RenS, et al. Delving deep into rectifiers: surpassing human-level performance on ImageNet classification[C]. IEEE International Conference on Computer Vision, 2015: 1026- 1034.
[16] GlorotX, BordesA, BengioY. Deep sparse rectifier neural networks[C]. 14th International Conference on Artificial Intelligence and Statistics, 2011: 315- 323.
[17] Oliveira GL, ValadaA, BollenC, et al. Deep learning for human part discovery in images[C]. IEEE International Conference on Robotics and Automation, 2016: 16055460.
[18] Brown M S, Lo P, Goldin J G, et al. Toward clinically usable CAD for lung cancer screening with computed tomography[J]. European Radiology, 2014, 24(11): 2719-2728.
[19] Torres E L, Fiorina E, Pennazio F. et al. Large scale validation of the M5L lung CAD on heterogeneous CT datasets[J]. Medical Physics, 2015, 42(4): 1477-1489.
[20] Choi W J, Choi T S. Automated pulmonary nodule detection system in computed tomography images: a hierarchical block classification approach[J]. Entropy, 2013, 15(2): 507-523.
[21] Lii S G A, Mclennan G, Bidaut L, et al. . The lung image database consortium(LIDC) and image database resource initiative (IDRI): a completed reference database of lung nodules on CT scans[J]. Medical Physics, 2011, 38(2): 915-931.
[22] van Ginneken B, Lii S G A, de Hoop B, et al. Comparing and combining algorithms for computer-aided detection of pulmonary nodules in computed tomography scans: the ANODE09 study[J]. Medical Image Analysis, 2010, 14(6): 707-722.
[23] 刘帆, 刘鹏远, 李兵, 等. TensorFlow平台下的视频目标跟踪深度学习模型设计[J]. 激光与光电子学进展, 2017, 54(9): 091501.
[24] Egan J P, Greenberg G Z, Schulman A I. Operating characteristics, signal detectability, and the method of free response[J]. Journal of the Acoustical Society of America, 1961, 33(8): 993-1007.
[25] Cascio D, Magro R, Fauci F, et al. Automatic detection of lung nodules in CT datasets based on stable 3D mass-spring models[J]. Computers in Biology & Medicine, 2012, 42(11): 1098-1109.
Article Outline
苗光, 李朝锋. 二维和三维卷积神经网络相结合的CT图像肺结节检测方法[J]. 激光与光电子学进展, 2018, 55(5): 051006. Guang Miao, Chaofeng Li. Detection of Pulmonary Nodules CT Images Combined with Two-Dimensional and Three-Dimensional Convolution Neural Networks[J]. Laser & Optoelectronics Progress, 2018, 55(5): 051006.