多模态磁共振脑肿瘤图像自动分割算法研究  下载: 1790次
下载: 1790次
1 引言
从医学图像中准确分割脑肿瘤区域对于疾病诊断、制定放射治疗计划等意义重大。磁共振成像(MRI)因其具有亚毫米级的空间分辨率,常用于肿瘤检测[1]。MRI有4种成像模态:T1、T1c、T2和Flair,不同的模态用于突出显示不同的肿瘤区域[2]。手动分割肿瘤区域是一项耗时且高度依赖医生个人经验的工作。其难点在于肿瘤形态各异,且可能分布于大脑的任意区域,快速、准确、自动地描绘出患者的肿瘤区域难度较大。
医学图像分割的根本问题是提取像素或体素的特征,然后对其分类。技术发展路线可分为手动分割、基于强度的分割、基于地图集的分割以及深度学习的方法。
手动分割是最原始的方法,耗时、繁琐,且存在人工差异。基于强度的分割主要是将像素的灰度值与某一个或多个阈值相比较来区分不同的组织[3]。常用的方法有区域分割法和聚类分割法,区域分割法具有较低的计算负荷,但对噪声很敏感,聚类分割法对初始化参数敏感,且难以考虑空间信息或像素间的依赖关系[4]。
“地图集”就是人工标记完备的数据库,基于地图集的分割就是将测试图像与当前数据库的数据进行非刚性配准,然后用标签传播的方法将地图集的标签映射到测试图像中。但非刚性配准的准确度不高,且计算复杂度高[5]。
2012年以来,以深度神经网络为代表的深度学习方法性能大幅超过传统算法的性能。深度学习方法不依赖手工制作的特征,可以直接从数据中自动地学习特征的各个层次结构[6]。Shelhamer等[7]提出全卷积网络(FCN),将原始卷积神经网络中最后用于分类的全连接层换成卷积层,以处理任意输入尺寸的图像。Ronneberger等[8]在FCN的基础上设计了2D-Unet网络,该网络由对称的“收缩路径(Contracting Path)”和“扩展路径(Expanding Path)”组成,收缩路径用于捕捉上下文信息,扩展路径用于定位感兴趣的结构,且在同一深度的收缩层和扩展层之间添加跳跃连接。此模型达到了2015年2D医学图像分割的最先进水平。然而大部分医学图像是3D的,2D切片会丢失相邻切片之间的三维空间信息,降低分割精度,且单纯地将2D分割结果拼接成3D分割结果,可能会出现锯齿、断层等问题[9]。Çiçek等[10]利用3D卷积核将其拓展为3D-Unet网络。Milletari等[11]在3D-Unet的基础上加入残差模块,缓解了网络过深时梯度消失的问题。Kayalibay等[12]在最先进的3D-Unet架构下采用Jaccard损失函数,改善了类别不平衡问题,取得了BraTS 2015数据集的最好成绩。
本文以BraTS 2017脑肿瘤数据集作为实验数据,在3D-Unet架构基础上,构建4通道全卷积神经网络。在收缩路径中加入混合膨胀卷积模块(HDC)以扩大每一层的感受野。在扩展路径中,将上采样得到的特征映射(Feature Maps)与本层的特征映射进行拼接,生成不同分辨率下的预测图,然后通过逐元素相加得到最终预测图。损失函数方面,受文献[ 13]的启发,将多类别Dice损失函数和Focal损失函数[14]相结合,得到混合损失函数(HLF),加强稀疏类分类错误对模型的惩罚,有助于改善类别不平衡问题。
2 自动分割方法
2.1 数据集与预处理
实验数据选取BraTS 2017数据集[15],其中包含210位高级别胶质瘤(HGG)和75位低级别胶质瘤(LGG)患者的4种MRI模态图像,原始尺寸为240 mm×240 mm×155 mm,分别来自于19个机构的临床数据。每位患者的肿瘤图像都由4位放射科医生手动标注出4类标签,分别代
BraTS 2017数据集图像已经剔除了头骨部分并配准到相同的解剖模板,分辨率为1 mm×1 mm×1 mm。经过观察,将图像裁剪为128 mm×128 mm×128 mm,进一步减少了无关信息但并未减少有用的肿瘤区域信息。为了避免数据不足可能导致的过拟合风险,在每次训练开始之前应用随机旋转、缩放、弹性变形、镜像等数据增强技术对数据集进行扩充。
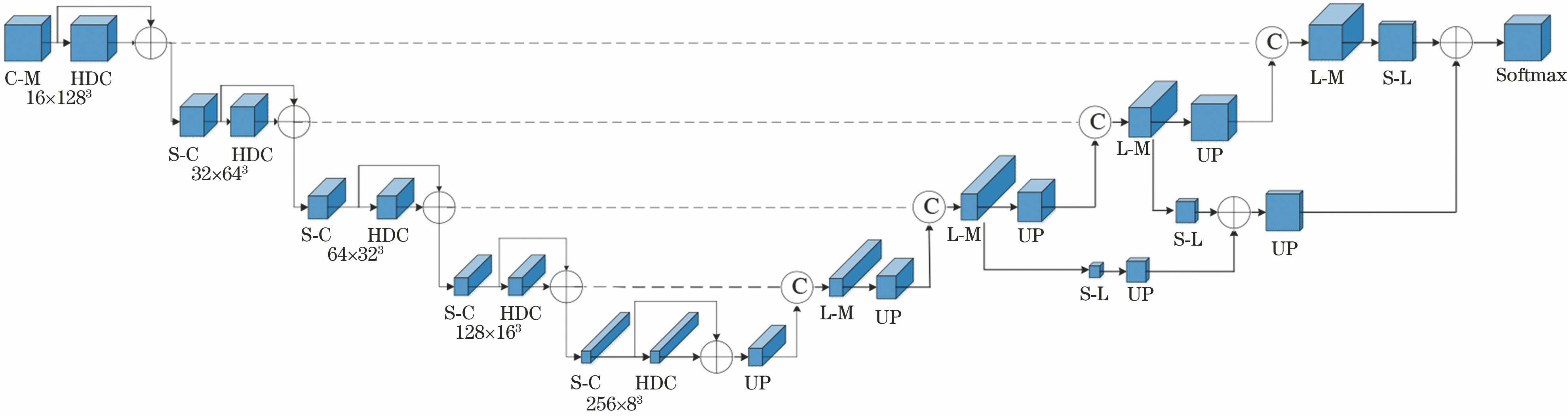
2.2 3D-HDC-Unet模型架构
3D-HDC-Unet模型如
模型的左半部分为收缩路径。第一个C-M模块是由16个尺寸为3×3×3的卷积层、实例标准化(Instance Normalization, IN)层和PReLU层组成的卷积模块。卷积层将输入的(4,128,128,128)体素块转换为16个的128 mm×128 mm×128 mm的体素块,相比起全连接网络,卷积操作的稀疏交互和参数共享两个特性,使得需要优化的参数数量大大缩减,每一层的神经元只响应前一层局部区域范围内的神经元,提高了模型的训练效率,提升了模型的可扩展性。普通3D卷积运算如

图 2. 3D卷积与输入体素块的点乘运算示意图。(a)普通3D卷积运算;(b)膨胀率r=2的3D膨胀卷积运算
Fig. 2. Dot product operation diagram of 3D convolution with input voxel block. (a) Original 3D convolution operation; (b) 2-dilated 3D convolution operation
IN层是针对整幅输入图像的所有像素值的灰度值作归一化处理,使所有体素块的灰度值服从均值为0、方差为1的正态分布,避免网络中每一层的输入的数据分布差异过大。
PReLU层实质为带参数的线性整流单元(Parametric Rectified Linear Unit),定义了来自上一层神经网络的某通道输入变量Xm的非线性输出结果,即
式中:am是可学习的参数,采用带动量的更新方式使其在反向传播中逐渐确定[16]。然后数据被送入混合膨胀残差模块,进行特征提取。前向传播时第l层的3D卷积核的权重Wl与第l-1层体素块Xl-1卷积之后,加上偏置项b后经过PReLU函数f处理,即可得到第l层的特征映射
式中:b为偏置项b组成的矩阵。
混合膨胀残差模块如
首先是在常规卷积的基础上引入变化的膨胀率(Dilation)。膨胀卷积(Dilated Convolution)可以增加单个神经元的感受野(Receptive Field),如
本研究采用膨胀率变化的混合膨胀卷积核来减弱棋盘效应。例如对于原来r=2的层,将3个级联的膨胀卷积层作为一个组,分别将其膨胀率设置为1、2、3,这样顶层可以访问更大范围的体素信息。这一操作对于模型识别较大的对象非常重要。混合膨胀卷积能在模型参数个数保持不变、下采样次数较少的情况下,大大增加三维卷积层的感受野[19],如

图 4. 收缩路径中输入图片降采样与混合膨胀卷积模块的感受野示意图
Fig. 4. Sketch map of input images sub-sampling and HDC receptive fieldNote:In layer 3 4 5, the size of images is enlarged to scale for better demonstration.
最后再将数据送入下一层步长为2的3×3×3的跨步卷积层进行下采样,将体素块3个维度的尺寸减半,特征映射层数加倍,一方面可以提升卷积层的感受野,另一方面可以降低存储空间需求,最终实现效率和精度的平衡。随着网络的加深,输入图像块的分辨率越低,特征的抽象程度越高。抽象程度低的特征映射用于捕捉边缘、转角等特征,抽象程度高的特征映射用于捕捉相邻体素的非线性强度关系。
扩展路径中,首先利用双线性插值法对低分辨率的特征映射进行上采样,将3个维度的尺寸扩大一倍,然后利用3×3×3的卷积将其特征映射的数量减半。同时,将此特征映射与同一层级的收缩路径中的特征映射在通道维度上进行拼接,再通过定位模块将其重新组合,以低空间分辨率编码上下文信息,并将该信息传送到更高空间分辨率的特征中。定位模块由两组重复的C-M模块组成。
在扩展路径的第3、2、1层分别进行不同分辨率的预分割。分割层首先由卷积层将特征映射的通道维数降至4,然后再接IN层和PReLU层。经过上采样后分别与上一层的特征映射进行矩阵相加。通过整合网络不同层级上的分割层,生成最终的预测图,最后利用Softmax层进行体素级预测。Softmax函数也称为归一化指数函数,用于计算输入张量X属于某类标签Y的概率P(y=Y|X),定义为
式中:exp(
2.3 混合损失函数
训练数据中的类别不平衡是阻碍模型精度提高的一大障碍,
表 1. BraTS 2017数据集中各类别体素占总体素的比值
Table 1. Ratio of different types of voxels to total voxel in the BraTS 2017 dataset
|
为此,本研究构造了一种结合Dice损失和Focal损失的混合损失函数来克服这个问题。Dice相似系数(DSC)是一种集合相似度的度量函数,用于计算预测区域与真实分割区域的重叠程度。Dice损失函数为负的DSC,定义为
式中:PTP,x(y)、PFN,x(y)和PFP,x(y)分别是将体素x预测为类别y的真阳、假阴和假阳的概率,定义为
其中px(y)是将体素x预测为类别y的预测概率,tx(y)是体素x对应的真实标签的one-hot编码值,X为输入MRI图像的体素总数,K为标签类别总数,本研究中取4。多类别分类时的Dice损失函数为
Focal Loss是通过降低易分类样本的损失权重,提高难分类样本(比如稀疏样本)的权重,来迫使模型更好地学习分类较差的样本,定义为
(9)式相当于在标准交叉熵损失前添加一个调节因子[1-px(y)]γ,γ∈N为聚焦参数,其功能在于:1)当样本分类错误,px(y)值较小时,调节因子接近于1,该体素对损失函数贡献正常的惩罚值;当样本分类容易,px(y)较大时,调节因子接近于0,该体素对损失函数的贡献降低。2)聚焦参数γ可以调整易分类体素对损失函数权重降低的速率。当γ=0时,LFocal相当于标准交叉熵损失,γ越大,易分类体素对损失函数的贡献越小。例如γ=1,px(y)=0.8时易分类样本与标准交叉熵损失相比损失值降低为1/5,这也变相地增加了纠正错误分类样本的重要性[14]。γ的取值对损失函数f的影响如
本文的损失函数由LDice和LFocal混合而成,即
混合比例由λ调节。由实验可得,当λ=0.5,γ=1时,模型的综合性能最佳。
3 仿真实验
3.1 评价指标
为了定量评估此模型的性能,选取医学图像分割中常用的4种评价指标:Dice系数(Dsc)、灵敏度(Sen)[21]、特异度(Spe)、Hausdorff距离(DHD)。Dsc表示预测肿瘤区域与真实肿瘤区域的重叠程度;灵敏度又称为真阳性率、召回率,表示真实肿瘤区域中被预测为肿瘤的比例;特异度又称为真阴性率,表示健康组织区域中被预测为健康的比例;Hausdorff距离用于度量两个点集的最大不匹配程度,对分割出的边界比较敏感。其定义为
(14)式中的P和T分别表示模型预测的肿瘤区域和真实肿瘤区域在二维图像上的像素集,p和t分别表示各自像素集中的像素点。
3.2 实验结果
从BraTS 2017数据集的285名患者中随机选出5名作为测试集,将剩余280名患者均分成5组,每组56人,在BraTS 2017数据集上进行5次交叉验证(5-fold Cross-validation),如
每次训练一位患者的MRI图像,总共训练300轮,每一轮训练224幅图像。优化器选用Adam,在反向传播时,首先利用
计算第l层的损失函数关于前一层l-1的梯度gl,然后根据
定义的一阶动量mt和二阶动量vt,分别初始化为m1=g1,v1=
对其进行偏差纠正后,再根据
对权重值进行更新,步长α=0.001。
利用Keras+TensorFlow深度学习框架搭建此模型,在Nvidia GTX 1070 GPU上进行约36 h的训练,训练好的模型大约需要5 s来完成一名患者的MRI图像的分割。
为了探究HDC模块和HLF各自对脑肿瘤分割性能的提升程度,在原始3D-Unet模型基础上分别添加HDC模块和HLF,训练出两个模型:3D-Unet+HDC和3D-Unet+HDC+HLF,连同使用普通Dice损失函数的双通道3D CNN网络,普通3D-Unet网络,K均值聚类(K-means Clustering),混合水平集(Hybrid Level),结合了FCM、自动阈值法和混合水平集的混合算法(Hybrid Algorithm)、随机森林(Random Forests),记录他们在测试集上的4种参数性能,分割的区域包括WT(标签1、2、4)、TC(标签1、4)和ET(标签4)。结果记录于
表 2. 8种模型的评价指标数据对比
Table 2. Comparison of evaluation index data of eight models
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
本文提出的模型算法流程图如
以患者Brats17_TCIA_167_1为例,分割结果如
结合

图 8. 算法流程图与各阶段图像处理结果
Fig. 8. Algorithm flowchart and image processing results of each stage
由于BraTS 2017的在线评估端口已经关闭,无法测试此模型在官方测试集上的具体性能结果,根据文献[ 22]中的数据,可知模型在官方未公开发布的测试集上的性能相比从训练数据中分割出的测试集的性能来说,会有5%以内的下降。但至少从训练数据中得到的性能参数可以看出,3D-HDC-Unet仍然取得了领先成绩。
4 结论
在原始3D-Unet的基础上,本研究加入HDC模块以增加神经元的感受野,可在较浅的网络中实现多尺度特征提取,并大大降低模型参数规模。用混合损失函数替换单一的Dice损失函数,提高了稀疏类对损失函数的贡献,迫使模型更好地学习不易分类的样本,从而提升了3D-HDC-Unet在BraTS 2017数据集上的分割性能。经过36 h的训练,模型在训练数据分割出的验证集上的WT、TC、ET的平均Dice系数为0.90、0.80、0.77,均优于原始3D-Unet模型。
[1] Niessen W J. MR brain image analysis in dementia: from quantitative imaging biomarkers to ageing brain models and imaging genetics[J]. Medical Image Analysis, 2016, 33: 107-113.
[2] 任璐, 李锵, 关欣, 等. 改进的连续型最大流算法脑肿瘤磁核共振成像三维分割[J]. 激光与光电子学进展, 2018, 55(11): 111011.
[3] Shanthi KJ, Kumar MS. Skull stripping and automatic segmentation of brain MRI using seed growth and threshold techniques[C]∥2007 International Conference on Intelligent and Advanced Systems, November 25-28, 2007, Kuala Lumpur, Malaysia. New York: IEEE, 2007: 422- 426.
[4] Saman S, Narayanan S J. Survey on brain tumor segmentation and feature extraction of MR images[J]. International Journal of Multimedia Information Retrieval, 2019, 8(2): 79-99.
[5] Bach Cuadra M, de Craene M, Duay V, et al. Dense deformation field estimation for atlas-based segmentation of pathological MR brain images[J]. Computer Methods and Programs in Biomedicine, 2006, 84(2/3): 66-75.
[6] Pereira S, Pinto A, Alves V, et al. Brain tumor segmentation using convolutional neural networks in MRI images[J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1240-1251.
[7] Shelhamer E, Long J, Darrell T. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651.
[8] RonnebergerO, FischerP, BroxT. U-net: convolutional networks for biomedical image segmentation[M] ∥Navab N, Hornegger J, Wells W, et al. Medical image computing and computer-assisted intervention-MICCAI 2015. Lecture notes in computer science. Cham: Springer, 2015, 9351: 234- 241.
[9] 褚晶辉, 李晓川, 张佳祺, 等. 一种基于级联卷积网络的三维脑肿瘤精细分割[J]. 激光与光电子学进展, 2019, 56(10): 101001.
[10] ÇiçekÖ, AbdulkadirA, Lienkamp SS, et al. 3D U-net: learning dense volumetric segmentation from sparse annotation[M] ∥Ourselin S, Joskowicz L, Sabuncu M, et al. Medical image computing and computer-assisted intervention-MICCAI 2016. Lecture notes in computer science. Cham: Springer, 2016, 9901: 424- 432.
[11] MilletariF, NavabN, Ahmadi SA. V-net: fully convolutional neural networks for volumetric medical image segmentation[C]∥2016 Fourth International Conference on 3D Vision (3DV), October 25-28, 2016, Stanford, CA, USA. New York: IEEE, 2016: 565- 571.
[13] Zhu W, Huang Y, Tang H, et al. Anatomynet: deep 3D squeeze-and-excitation U-Nets for fast and fully automated whole-volume anatomical segmentation[J]. bioRxiv, 2018, 392969.
[14] Lin T Y, Goyal P, Girshick R, et al. Focal loss for dense object detection[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2018, 1.
[15] Menze B H, Jakab A, Bauer S, et al. The multimodal brain tumor image segmentation benchmark (BRATS)[J]. IEEE transactions on medical imaging, 2014, 34(10): 1993-2024.
[16] He KM, Zhang XY, Ren SQ, et al. Delving deep into rectifiers: surpassing human-level performance on ImageNet classification[C]∥2015 IEEE International Conference on Computer Vision (ICCV), December 7-13, 2015, Santiago, Chile. New York: IEEE, 2015: 1026- 1034.
[17] Liu Y, Stojadinovic S, Hrycushko B, et al. A deep convolutional neural network-based automatic delineation strategy for multiple brain metastases stereotactic radiosurgery[J]. PLoS One, 2017, 12(10): e0185844.
[18] YuF, KoltunV, FunkhouserT. Dilated residual networks[C]∥2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), July 21-26, 2017, Honolulu, HI, USA. New York: IEEE, 2017: 636- 644.
[19] Wang PQ, Chen PF, YuanY, et al. Understanding convolution for semantic segmentation[C]∥2018 IEEE Winter Conference on Applications of Computer Vision (WACV), March 12-15, 2018, Lake Tahoe, NV, USA. New York: IEEE, 2018: 1451- 1460.
[20] He KM, Zhang XY, Ren SQ, et al. Identity mappings in deep residual networks[M] ∥Leibe B, Matas J, Sebe N, et al. Computer vision-ECCV 2016. Lecture notes in computer science. Cham: Springer, 2016, 9908: 630- 645.
[21] Havaei M, Davy A, Warde-Farley D, et al. Brain tumor segmentation with deep neural networks[J]. Medical Image Analysis, 2017, 35: 18-31.
[22] Menze B H, Jakab A, Bauer S, et al. Proceedings of the MICCAI challenge on multimodal brain tumor image segmentation (BRATS) 2017[J]. MICCAI Challenge on Multimodal Brain Tumor Image Segmentation, 2017, 10: 1-347.
[23] 童云飞, 李锵, 关欣. 改进的多模式脑肿瘤图像混合分割算法[J]. 信号处理, 2018, 34(3): 340-346.
Tong Y F, Li Q, Guan X. An improved multi-modal brain tumor segmentation hybrid algorithm[J]. Journal of Signal Processing, 2018, 34(3): 340-346.
[24] Zhang C, Shen X J, Cheng H, et al. Brain tumor segmentation based on hybrid clustering and morphological operations[J]. International Journal of Biomedical Imaging, 2019, 2019: 7305832.
Article Outline
何承恩, 徐慧君, 王忠, 马丽萍. 多模态磁共振脑肿瘤图像自动分割算法研究[J]. 光学学报, 2020, 40(6): 0610001. Cheng'en He, Huijun Xu, Zhong Wang, Liping Ma. Automatic Segmentation Algorithm for Multimodal Magnetic Resonance-Based Brain Tumor Images[J]. Acta Optica Sinica, 2020, 40(6): 0610001.