整体器官的光透明成像方法综述  下载: 2893次特邀综述
下载: 2893次特邀综述
1 引言
生物体是由复杂多样的细胞构成的,其结构与功能通常是作为一个整体表现出来的。在器官乃至全身水平上,高分辨地获取生物组织三维结构信息对诸多生物医学相关的研究至关重要,例如神经科学中大脑神经元连接信息的绘制、发育生物学中器官的形态发生等[1-3]。
常用的医学影像技术,例如MRI(Magnetic Resonance Imaging)、CT(Computed Tomography)、PET(Positron Emission Tomography)等,虽能够获取组织的三维结构与功能信息,但其空间分辨率十分有限,仅能获得宏观尺度的图像信息,难以获得细胞的精细结构。电子显微镜技术虽然能够进行纳米尺度的分辨成像,但仅适用于获取极小组织样本的微观结构,而将其应用到厘米尺度的样本时,耗费的时间不可估量。
现代光学成像技术的不断发展填补了上述宏观成像与微观成像之间的空白,为生物组织器官介观尺度的成像提供了重要工具[4]。如单光子/双光子显微成像技术[5-7]、光片照明成像技术[8-12]等,能够通过光学切片层析成像的方法实现生物组织的三维成像。然而,大多数生物组织对光具有较强的散射与吸收,这限制了光在组织中的穿透深度[13-16],导致成像深度局限在几十到几百微米。为了提高光学成像深度、改善成像的质量,使大家能够看得更深、更清晰,研究人员从多个方面开展了工作:发展新型光学成像技术,例如三光子显微成像技术等[17-18];发展长波长的荧光探针,如近红外探针等[19-20]。但要实现对厘米尺度的大体积组织器官的整体高分辨成像仍然十分困难。
传统组织切片的方法(切片-染色-薄片成像-计算重构)[21-22]虽然在生物学研究中起到了非常重要的作用,能够获得足够高的横向分辨率,但是将其应用于大组织成像时,费时费力,且切片易丢失或变形,从而导致大量信息丢失,令后续图像配准及三维重建变得极为复杂与困难[23]。近年发展的基于自动连续切片和成像的技术,如序列双光子成像(STP)[24]、显微光学断层成像(MOST)[25-27]等,有效解决了上述问题,显著地提高了成像的通量。
近年兴起的组织光透明技术则从另一个角度出发,通过采用多种物理、化学的手段,减小组织对光的衰减,使得生物组织变得对光“透明”,从而提高了光学成像的成像深度与成像质量[2, 28-29]。该方法与多种荧光标记技术、光学成像技术的结合为高分辨获取组织器官的三维整体结构信息提供了重要的手段,在生命科学特别是在神经科学领域产生了巨大的影响[30-31]。
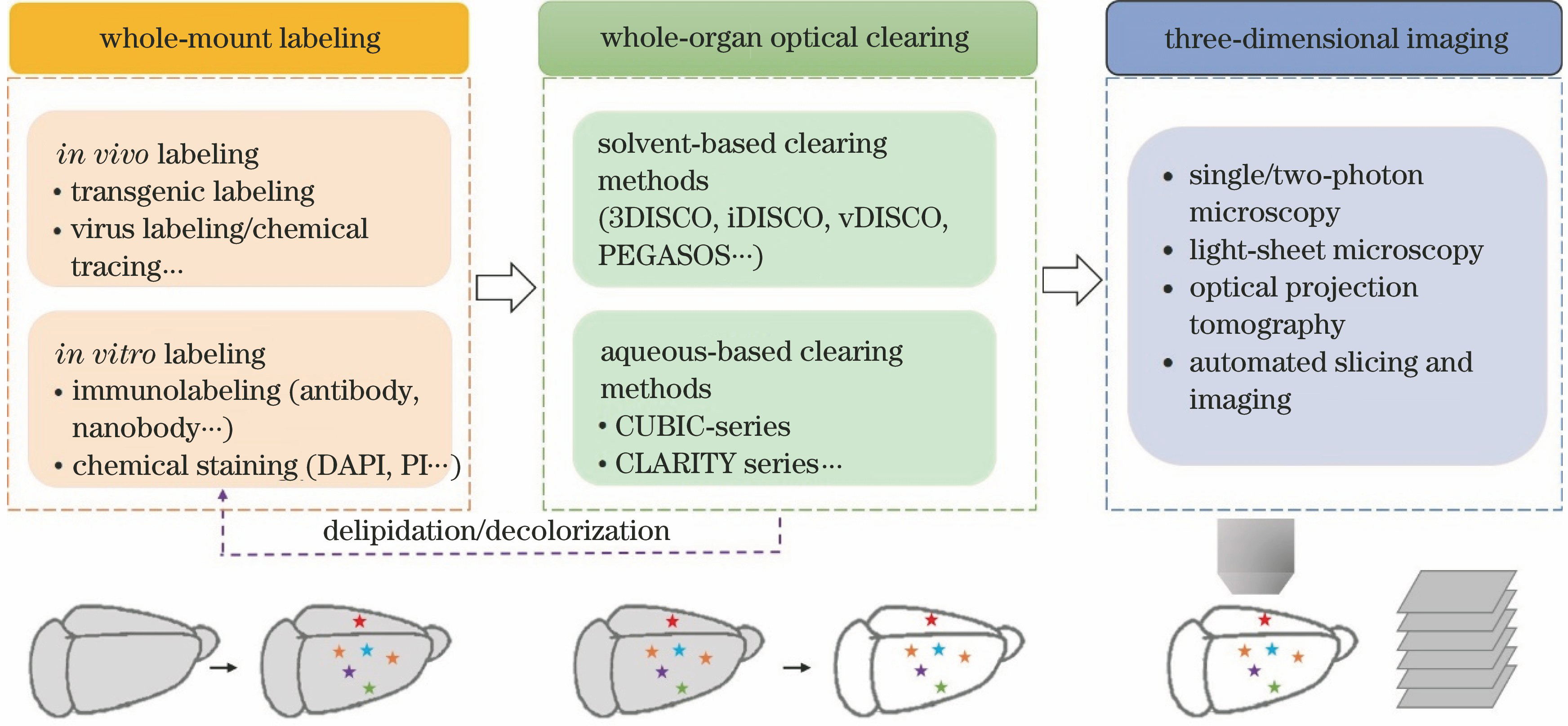
在整体器官的光透明成像研究中,组织光透明、光学成像方法及细胞结构或分子的特异性标记缺一不可(
2 离体组织光透明的基本策略
生物组织是由多种具有不同光学特性的复杂成分组成的,当光穿过组织时,组织对光的衰减主要来自两个方面:光散射和光吸收[2-3]。光散射使光偏离原来的方向,不能穿过组织向前方传播,使光分布变宽,从而使光学成像的图像变得模糊;光吸收则会将光能转换为其他形式的能量(如:热),导致光强衰减从而使光学成像的信号强度变弱。这种现象在组织深层位置表现得更为明显。
因而,组织光透明技术的主要目的是减小组织对光的散射和吸收,增加光穿透深度,以提高光学成像的深度与对比度。组织光透明方法的基本策略也是基于这两个方面(
![组织光透明方法的两大策略:减小光散射与光吸收[3]](/richHtml/zgjg/2020/47/2/0207007/img_2.jpg)
图 2. 组织光透明方法的两大策略:减小光散射与光吸收[3]
Fig. 2. Two strategies for tissue optical clearing methods: reduction of light scattering and light absorption[3]
2.1 减小光散射
生物组织的高散射主要是由其组成成分折射率之间的差异导致的[13]。一方面,生物组织大多由水(n=1.33)、脂质(n≈1.44)、蛋白质(n≈1.43)、矿物质等多种物质组成,这些折射率不同的物质非均匀地分布在组织内,导致组织内各个区域的折射率存在差异,从而引起较强的光散射;另一方面,细胞内外的不同结构成分的折射率也存在较大差异,例如细胞液、细胞间液等成分的折射率较低,而细胞膜、细胞器等成分的折射率较高[32]。
组织光透明技术的策略之一就是减小组织散射。一方面,通过向组织中引入高渗、高折射率的试剂,提高组织内部的折射率匹配程度,达到减小组织对光的散射的目的,进而提高光在组织中的穿透深度[13]。近年,组织光透明技术发展迅速,多种光透明方法不断涌现,不同方法中使用的折射率匹配试剂也有所不同,按照试剂的化学性质,可大致将折射率匹配试剂分为两类:有机溶剂和亲水性试剂[2,28,33]。使用这些高折射率试剂替代组织中折射率低的成分,使组织变得透明。
另一方面,去除组织中高折射率、高散射的成分,例如脂质[34-37]、蛋白质[38]等,也有利于提高折射率匹配程度,是实现组织光透明的另一个重要手段。光透明方法中,常用具有溶脂能力的有机试剂[34]、高浓度的表面活性剂对组织进行脱脂[37,39-40],也有利用表面活性剂结合组织电泳的方式实现了高效脱脂[35,41-42],提高了组织的透明程度。在全身成像时,骨组织的透明十分必要。骨组织与软组织的最大差异是其组成成分具有矿物特性,其折射率明显高于周围组织,对光具有较高的散射。上述的处理方法能够一定程度地减小骨组织的散射并提高成像的质量,但效果十分有限,而在光透明处理过程中引入脱钙处理会显著提高骨组织的透明度[43-45]。
2.2 减小光吸收
阻碍光穿透的另一个重要因素是组织对光的吸收。生物组织内有诸多的吸收成分,特别是血红素和黑色素,这些成分在组织内分布广泛,在可见光范围内对光具有很强的吸收。因而,去除这些吸收成分将有利于实施组织深层成像,通常称这一过程为脱色。
血红蛋白和肌红蛋白中的血红素是吸收可见光的主要色团,它会显著降低血液对可见光的透过率。因而,对于一些血红素含量丰富的组织器官,例如脾、肾等,有效地去除其中的血红蛋白对于提高整体器官光学成像的质量至关重要。
过氧化氢是一种传统的脱色试剂[46],它能够破坏蛋白结构,具有较强的脱色能力;此外,使用酸丙酮或者强碱处理样本也可以使血红素解离,从而达到脱色的效果[47]。但这些方法不利于保存细胞内的荧光蛋白,因而在荧光成像中并不常用。近年有研究发现氨基醇类试剂能够去除血红素[40],可能的机制是其与球蛋白链结构相似,可取代血红素与球蛋白链的配位点,与血红素结合。
除上述化学手段以外,一些物理手段也被用来去除血红素。简单的缓冲液和固定液灌注可以有效地去除血管中的红细胞,也就去除了大多数的血红素。但在某些特定的组织器官中,例如脾脏,灌注后血液的残留尤其严重。此外,现有的一些光透明方法采用十二烷基硫酸钠(SDS)[35,42,48],或结合组织电泳的电势差去除组织中残留的血红素成分,从而达到脱色的效果。
2.3 现有光透明方法的主要化学过程
如2.1与2.2节所述,目前所有的光透明方法具有相似的物理原理,最终是要降低组织散射或组织吸收,但使用的化学方法各异[32]。下面将简要介绍现有组织光透明方法的主要化学过程。从化学方法上,目前的组织光透明方法可分为两大类:基于有机溶剂的透明方法与基于亲水性试剂的透明方法。
基于有机溶剂的光透明方法的透明步骤主要包括脱水、脱脂/漂白及折射率匹配几个部分。光透明前通常采用多聚甲醛(PFA)固定样本。脱水过程主要基于醇类试剂,如乙醇(EtOH)、1-丙醇、叔丁醇(t-BuOH)、四氢呋喃(THF)等;脱脂主要利用四氢呋喃与二氯甲烷(DCM);部分不关心内源荧光蛋白的方法也需要对样本进行漂白处理,主要使用过氧化氢;折射率匹配主要基于醚类试剂,如水杨酸甲酯、苯甲醇-苯甲酸苄酯(BABB)、二苄醚(DBE)、二苯醚(DPE)、肉桂酸乙酯(ECi)、聚(乙二醇)甲基丙烯酸酯(PEGMMA500)等。这些试剂均可与有机溶剂混溶,因此具有高折射率的芳香烃溶剂(n≈1.56)能够渗透进组织中,起到均质化的作用。可能是由于所用试剂的分子较小,这类方法的透明速度通常较快。
基于亲水性试剂的光透明方法的透明步骤主要包含脱脂/脱色及折射率匹配。不同透明方法对样本的固定要求不同,除了最常规的PFA固定以外,也有用戊二醛(GA)固定的方式,或者引入水凝胶对样本进行包埋,用于后续的脱脂、脱色等处理。脱脂主要基于去污剂,如Triton X-100[37,39,49-50]、三乙醇胺(TEOA)[39-40,49]、SDS [35,42]等;脱色主要基于Quadrol[40]、SDS[43],还有抑制氧化反应的硫代甘油[51]或亚硫酸钠[52]等;折射率匹配试剂主要是高浓度的糖类或基于造影剂的高折射率溶液,如甘油[35]、蔗糖[39,53]、果糖[51,54]、山梨醇[48,55]、聚乙二醇[56]、葡甲胺[52,57-58]、碘海醇[48,57,59-61]、碘克沙醇[52]、二甲基亚砜[55]等,它们的折射率一般大于1.40。按照试剂作用的过程,该类方法可再细分为三类:一类是完全基于折射率匹配,利用梯度浓度的糖醇溶液实现透明;另一类是基于尿素或甲酰胺的水合作用增加组织的通透性,结合脱脂/脱色试剂,最后再进行折射率匹配;还有一类是基于水凝胶包埋增加组织结构的稳定性,结合SDS试剂(以及电泳、温升)等实现脱脂/脱色,最后用高折射率试剂进行折射率匹配,实现组织透明[2,62]。
![组织光透明方法的化学过程[32]。(a)基于有机溶剂透明方法的化学过程;(b)基于水溶性试剂透明方法的化学过程](/richHtml/zgjg/2020/47/2/0207007/img_3.jpg)
图 3. 组织光透明方法的化学过程[32]。(a)基于有机溶剂透明方法的化学过程;(b)基于水溶性试剂透明方法的化学过程
Fig. 3. Overview of the main steps of tissue optical clearing methods[32]. (a) Main steps of organic solvent-based clearing methods; (b) main steps of hydrophilic reagent-based clearing methods
3 整体组织器官透明方法
近年,离体组织光透明方法层出不穷,被广泛用于多种生物组织的透明与深层成像,不同光透明方法的适用性不同。一些基于简单试剂交换的透明方法,例如FocusClear、 SeeDB(See Deep Brain)、FRUIT(一种基于果糖和尿素的光透明方法)、ClearT/T2、DMSO/Sorbitol等,多适用于小组织块,能一定程度地提高组织光学成像的深度和成像质量,但用于大组织成像时,这些方法的透明效果仍十分有限,通常不足以满足完整器官整体成像所要求的透明度[29]。当然,也有将这些基于简单交换的透明方法(例如DMSO/Sorbitol)与自动切片技术结合,用于实现大组织的三维结构成像。本文将着重介绍用于完整器官或大样本透明的离体组织光透明方法。
根据前述化学过程,可将光透明方法分为基于有机溶剂的方法和基于水溶性试剂的方法。下面将从这两个方面依次进行介绍(

图 4. 适用于整体组织器官透明的离体光透明方法
Fig. 4. Classification of tissue optical clearing methods for imaging whole organs
3.1 基于有机溶剂的方法
为了实现完整器官的透明,Spalteholz大约在一个世纪以前就发明了一种基于有机溶剂的方法,但该方法会明显破坏组织表面结构,且不能保存荧光信号。近些年,Dodt课题组在这方面开展了大量的研究工作。基于有机溶剂的光透明技术通常包含两个部分,一是伴随溶脂的脱水过程,二是伴随溶脂的折射率匹配过程。他们最开始使用乙醇及BABB作为透明试剂,但会使荧光信号快速淬灭[63-66];而利用四氢呋喃替代乙醇进行脱水后,荧光保存的问题在一定程度上得到了改善[67-68];接着,为了获得更好的透明度,进一步使用二苄醚(DBE)替代BABB,最后形成了一种新的透明方法3DISCO (3D Imaging of Solvent-Cleared Organs)[34]。得益于3DISCO方法的高透明度,结合光片照明显微成像技术,研究者对全脑与脊髓节段的典型神经元与血管结构进行了研究。
Schwarz等[69]提出的FluoClearBABB是另一种基于BABB的方法,该方法使用1-丙醇或叔丁醇进行脱水,其优点是在长时间保存过程中样本荧光不会明显衰减。Ethanol-ECi方法使用pH调节的乙醇进行脱水,并利用ECi进行折射率匹配,所用的试剂毒性极小,被用于多种组织器官的透明,包括骨、心及肾[70-71]。
近期,Pan等[72] 利用发明的uDISCO(Ultimate DISCO)方法展示了全身的透明结果以及成像结果。该方法用叔丁醇进行脱水,用BABB与二苯醚混合物进行折射率匹配,并在试剂中引入维生素E等抗氧化剂,使荧光蛋白的保存期长达数月。Zhao课题组[36]最新提出了一种PEGASOS(A Polyethylene Glycol-Associated Solvent System)方法,同样也在大鼠及小鼠上展示了高效的全身透明结果。它引入乙二胺四乙酸(EDTA)与Quadrol分别进行脱钙及脱色预处理,利用Quadrol调过酸碱度的叔丁醇溶液进行梯度脱水,并在脱水与透明试剂中引入PEGMMA500,将苯甲酸苄酯与PEGMMA500用于折射率匹配过程,不仅能有效地透明软组织,对硬组织(如人的牙齿组织)也十分有效。上述这两种方法在实施全身透明过程中均利用了心脏循环系统进行灌注。
大多数光透明方法秉承保存内源荧光(如:绿色荧光蛋白)的原则,而Renier等[46]提出的iDISCO(Immunolabeling-Enabled Three-Dimensional Imaging of Solvent-Cleared Organs)方法不执着于样本内源荧光的保存,他们从另一个角度出发,尝试用整体免疫标记对这些蛋白进行染色,然后用3DISCO方法进行透明成像。研究人员利用iDISCO方法对啮齿类动物的完整器官(如:脑、肾),甚至人类胚胎及胎儿标本进行了成像研究[73],在多种复杂的大组织上展示了几个前所未有的分子标记结构。在后续的研究中,Renier等[74]为了减少样品的收缩,更好地保护组织形态,进一步改进了iDISCO方法的步骤,发展了iDISCO+方法。Wang等[75]针对骨组织提出了BoneClear方法,在预处理过程中引入EDTA-Na对组织进行脱钙处理,调整透明处理过程中每步的时间,对iDISCO+方法进行了优化改进,实现了对成年小鼠完整骨组织中多种细胞结构的免疫标记与三维成像。最近,Ertürk团队[76]发展了一种基于纳米抗体的全身免疫标记方法vDISCO,该方法显著增强了荧光蛋白信号的强度,构建了成年小鼠全身的神经投射图谱,揭示了颅骨骨髓与脑膜之间的血管连接,并在中枢神经损伤模型上观察了神经投射的变化与免疫反应的过程,为全身的全景成像提供了技术支撑。
上述方法为鼠脑组织的标记提供了有效的手段,但对人脑组织,特别是福尔马林固定过的成年脑组织而言,标记仍然较难。Hildebrand等[77]发展了一种简单、快速且低成本的人脑组织细胞构筑标记方法——MASH(Multiscale Architectonic Staining of Human Cortex),也是基于甲醇预处理及过氧化氢漂白,利用吖啶橙、亚甲基蓝、甲基绿或中性红等有机小分子染料对细胞质或细胞核进行标记,可应用于厚达5 cm的福尔马林固定的成人脑样本。
3.2 基于水溶性试剂的方法
针对完整/成年组织器官的透明方法,除了上述基于有机溶剂的方法,还有很多基于水溶性试剂的方法。
Miyawaki团队于2011年报道的Scale(一种基于尿素的水溶性光透明剂)方法[78]源于一个偶然发现:尿素具有透明能力。Scale方法利用尿素与甘油的混合试剂,在小鼠大脑与完整胚胎上获得了良好的透明效果,同时保留了组织内源荧光信号。将双光子显微镜与Scale结合,能够实现全脑成像,并能以亚细胞分辨率重建神经投射。但其透明时间长达数月,组织会发生明显的膨胀且较为脆弱。因此,他们继续对该方法进行了改进,在2015年提出了ScaleS(一种基于尿素及山梨醇的水溶性光透明剂)方法,其关键的技术进步是引入一种糖醇——山梨醇,该方法不仅加速了透明过程,且对样本的形态与结构表现出了极好的保存能力,透明后的组织能够用于切削与电镜检测[79]。
同时,Ueda课题组[39]基于Scale的试剂组成,进行了复杂的化学试剂筛选,提出了一种基于尿素的高效透明方法——CUBIC(Clear, Unobstructed Brain Imaging Cocktails)。该方法能够在约2周的时间内实现全脑的透明,适用于多种荧光蛋白标记的脑和免疫染色的组织,还被用于狨猴脑组织的细胞构筑的研究[49]。同时,CB-Perfusion(CUBIC Based on Transcardial Perfusion)方法利用心脏灌注将CUBIC相关的透明试剂灌到小鼠体内,再结合后续的浸泡,实现了成年小鼠的全身透明[40]。该团队接着针对癌症研究发展了基于CUBIC-cancer的透明方法,其相比CUBIC使用了更有效的脱脂试剂及更高折射率的透明试剂,在无需灌注的条件下,即可实现全身的透明,满足单细胞水平的全身肿瘤转移的检测[50]。近年,Ueda团队[80]又提出了一种水溶性光透明方法CUBIC-X,该方法能够基于膨胀实现整体器官的高效透明,被用于单细胞水平的三维脑图谱的获取研究中。还有一些基于尿素及尿素类似物的水溶性方法,例如:UbasM(Urea-Based Amino-Sugar Mixture),相比于CUBIC方法,能够较快地实现小鼠多种器官的透明,并能保存脂溶性染料的荧光信号[58]; Ce3D(Clearing-Enhanced 3D)方法利用甲基乙酰胺与碘海醇实现了多种完整器官的高度透明[61]。
与上述方法均不同,CLARITY(Clear Lipid-Exchanged Acrylamide-Hybridized Rigid Imaging / Immunostaining / In situ Hybridization-Compatible Tissue-Hydrogel)是基于水凝胶包埋实施光透明的一种全新方法[35,81]。首先通过水凝胶单体与组织的交联来稳定组织结构,然后利用组织电泳装置(ETC)与强离子型去污剂(SDS)主动去除脂质,最后将组织浸泡到折射率匹配溶液中,达到最终的透明效果。该方法不仅能使组织变得光学透明,也使得组织对大分子的通透性增强,这使得完整组织的多轮分子表征变为可能。
CLARITY虽然是一种主动的去脂方法,但仍受透明速率慢及样本破坏风险的限制。目前,有很多针对CLARITY的优化方法。Epp等[82]通过交替设置37 ℃与55 ℃来加速透明过程;Bastrup等[83]通过优化ETC装置来避免透明过程中组织的损坏。Chung课题组[41]提出了一种利用随机电场(SE)对完整组织进行无损、快速透明和染色的方法,能够在1~3天实现小鼠器官的透明,但它需要复杂的定制装置。同时,Chung课题组[52]提出了一种SWITCH(System-Wide Control of Interaction Time and Kinetics of Chemicals)方法,该方法不需要特殊的装置,选择戊二醛作交联剂来保存组织结构与分子成分,并通过温升来加速透明过程,被应用于小鼠脑、大鼠脑、狨猴乃至人体组织样本的透明与标记[84]。它能够实现人体组织的22轮分子标记,但不能保存内源荧光。同时,他们[85]提出了一种MAP(Magnified Analysis of the Proteome)方法,该方法通过凝胶包埋、组织变性以及膨胀处理,也能实现小鼠全脑、心、肺、肝、肾等器官的透明。因为吸水效应,该方法处理样本获得的透明度极高,但组织膨胀数倍,这无疑增加了整体成像的难度与挑战。近年,Chung课题组[86]提出了一种SHIELD(Stabilization Under Harsh Conditions via Intramolecular Epoxide Linkages to Prevent Degradation)方法,该方法利用聚环氧化物在生物分子内或分子间形成交联,稳定了组织结构,进而利用SDS浸泡或SE进行脱脂,最后利用折射率匹配试剂进行透明处理。其优势是能够在多种苛刻条件下保存蛋白质荧光和抗原性、转录产物和组织结构。最近,研究人员提出了一种利用电场驱动及亲和力控制来加速标记的方法——eFLASH方法,首先利用SHIELD固定组织结构,再利用SE进行去脂透明和标记,通过调节标记溶液的pH和脱氧胆酸钠溶液的浓度来控制探针-靶标的结合亲和力,能快速实现多种组织完整、均匀的标记[87]。
PACT(Passive CLARITY Technique)是一种基于CLARITY的被动透明方法,仅采用试剂浸泡的方式对样本进行透明,已被用于多种组织器官的成像,包括脑、脊髓、肾、心、肺、小肠[48,59]。后来也有许多基于PACT的优化方法。PARS(Perfusion Assisted Agent Release in situ)方法基于CLARITY的基本原理,利用动物自身的循环系统将光透明相关的试剂运输到全身,实现全身的快速透明与标记[48,59]。该方法对水凝胶包埋试剂、透明试剂以及成像试剂均作了优化,可以实现原位透明,并能够较好地保存样本荧光和组织结构。Bone-CLARITY(Bone Tissue Clearing Method)则是针对骨组织的优化方法,它通过结合EDTA与氨基醇进一步提高了骨组织的透明能力[43]。Lee等[42]提出一种基于压力诱导的高效稳定传递大分子的主动透明方法(ACT-PRESTO),通过优化水凝胶聚合以及组织电泳的条件,该方法能在几小时到几天的时间内实现多种生物体的全身透明,包括小鼠、斑马鱼、蟾蜍等。并且,在ACT-PRESTO方法实施标记的过程中,大分子向深层组织的渗透能力也因为压力的引入而有所提升。该方法还适用于大鼠脑、兔脑、人体脊髓节段组织的透明。
上述大多数的光透明方法主要关注蛋白结构,而 用1-(3-二甲基氨基丙基)-3-乙基碳二亚胺作为RNA固定剂的透明方法EDC-CLARITY则尝试挖掘大组织样本中的RNA信息,利用基于碳化二亚胺的化学试剂稳定组织中的RNA结构,实施组织透明,以观察完整组织中多种RNA的分布[88]。
综上所述,目前的光透明方法种类非常多,每种方法都有其适用性和特点。
随着组织光透明方法的不断涌现,基于组织光透明的应用研究也得以飞速发展。从早期的果蝇、斑马鱼的三维成像[90-93],以及啮齿类动物(如大鼠、小鼠)的脑、脊髓与其他器官三维结构的获取[68,72,94],发展到人体组织器官[36,57,73,77,79,86,95],乃至人体器官芯片的成像研究[96-99]。研究的问题也从观察正常组织的三维结构(如鼠脑单细胞水平脑图谱绘制[80]),发展到研究病理过程相应结构的变化(如阿尔茨海默病脑的病理变化[79,100]、肿瘤转移过程全器官成像[50]),疾病介入治疗效果的评估(如干细胞移植疗效评估等[72]),乃至人类胚胎早期发育[73]等,涉及面十分广泛,极大地推动了生命科学的研究进展。
![不同类型光透明方法的透明效果 [34-35,39,50,52,59,76,80,89]](/richHtml/zgjg/2020/47/2/0207007/img_5.jpg)
图 5. 不同类型光透明方法的透明效果 [34-35,39,50,52,59,76,80,89]
Fig. 5. Clearing performance of different optical clearing methods[34-35,39,50,52,59,76,80,89]
表 1. 几种典型光透明方法的性能比较
Table 1. Comparison of several typical optical clearing methods
|
3.3 商业化的光透明试剂及仪器
目前,商用的光透明试剂与产品包括早年Olympus的ScaleView-A2,FUJIFILM Wako的ScaleView-S、CUBIC等系列试剂,CelExplorer的FocusClearTM和DeepClearTM试剂,Visikol公司的Visikol HISTOTM等产品,SUNJin Lab的RapiClear系列试剂产品,国内礼智生物科技有限公司代理的PEGASOS试剂等,Logos Biosystems开发的组织透明化处理系统X-CLARITYTM与相关试剂,LifeCanvas Technologies的EasyClear与SmartClear II Pro透明系统及配套的透明试剂。
4 基于光透明的大组织标记策略
在整体器官成像的研究中,除了第3节讨论的组织透明与成像方法,细胞结构或分子的特异性标记也十分重要。目前,细胞标记的主要方法包括:1)转基因标记;2)在体病毒标记或化学染料标记;3)离体免疫组化标记或化学染料标记。这些标记技术在生命科学的研究中发挥了极为重要的作用。从样本获取的角度,前两者可归类为取材前(在体)标记,第三类可归为取材后(离体)标记。本文将着重介绍针对大组织或器官的整体免疫组化(染料)标记方法。
离体组织的标记通常依赖于分子探针的被动扩散,因而免疫组化标记技术通常被用于组织薄片或小样本的标记。目前,为了实现大组织或器官的整体免疫标记,主要采用以下方法(
4.1 增加大组织样本渗透性
为了加速分子的渗透,常用的方法包括减轻样本固定程度、反复冻融、反复脱水-复水、温升、引入促渗试剂(如二甲基亚砜DMSO)等[101-103]。Renier等[46]提出一种iDISCO方法,通过结合脱水-复水、增加孵育温度及DMSO的浓度来增加组织的通透性,能对多种组织器官(如小鼠脑、胚胎、肾等)进行整体标记与成像。
其他一些具有高效透明能力、适用于整体器官的光透明方法[37,39,79,81],例如CLARITY、CUBIC、AbScale等,不仅能够增加组织对光的通透性,也能够增加组织对大分子物质的通透性。透明过程中,脂质的去除、胶原结构的疏松及多孔水凝胶的形成均有利于抗体分子的渗透,能够加速标记过程,但基于分子被动扩散的染色过程仍需要较长的时间。例如,CUBIC浸泡处理的成年小鼠下丘脑组织的染色仍需要1周,CLARITY处理的完整小鼠脑的免疫标记需要1个多月,AbScale标记成年小鼠半脑大约需要9天。
4.2 施加外力促进分子渗透
在染色过程中施加外力,例如流体压力、电场力,无疑会加速分子的渗透。Lee等[42]提出了增加流体压力的标记方法PRESTO,其中又分为利用离心力的c-PRESTO和利用注射泵增加对流的s-PRESTO,该方法能在3 h左右完成30~120 μm厚的组织的标记。此外,Li等[105]在染色过程中施加电场,有效地增大了标记速率,且并未引起细胞结构的改变或变形。同时,Kim等[41]提出的随机电场方法利用组织与分子探针之间的电迁移率的差异,基于电场力在1天内完成了小鼠器官的完整标记,该方法适用于多种标记探针,例如核染料以及抗体分子等。
4.3 选择小尺寸探针
大组织或器官的标记的难度主要在于分子的渗透。除了4.1和4.2节的增加组织渗透性或者施加外力促进分子渗透的方法以外,从分子本身的角度出发,例如在染色过程中选择尺寸较小的探针类型[如亲合体(affibody)、F(ab')2片段、Fab片段、纳米抗体],分子越小,扩散系数越大,标记的速率会得到很大的提高。
Ertürk团队[76]提出了基于纳米抗体的vDISCO方法,利用纳米抗体结合高压循环灌注实现了小鼠全身的标记,显著增强了组织中内源性荧光蛋白信号的强度。
4.4 调节探针-靶标结合的反应动力学
在免疫染色中,抗体分子与抗原的特异性结合受很多因素的影响,包括pH、温度、电解质。Chung团队[52]提出的SWITCH方法通过控制组织内探针-靶标结合的反应动力学来增强探针在组织中的渗透。具体而言,他们采用了一组缓冲液:SWITCH-off和SWITCH-on, SWITCH-off缓冲液可抑制标记探针与其靶标之间的结合反应,SWITCH-on缓冲液可促进反应,从而实现对探针-靶标之间反应动力学的可逆调节。在SWITCH-off缓冲液中将分子探针(例如抗体)分散在整个组织中,然后将缓冲液“切换”到SWITCH-on缓冲液,使结合反应发生。这样,既可以促进探针在组织中的穿透,又可以提高标记的均匀性。利用SWITCH方法,整个小鼠半脑的髓鞘纤维可在9天之内被均匀染色。
最近,Chung团队[86]基于前期的工作优化发展了一种超快标记方法eFLASH,声称可以在一天内实现多种组织(如鼠脑、肠、狨猴脑块等)的完整、均匀标记。该方法首先基于SHIELD固定组织结构,然后利用SE对交联的组织进行去脂,再基于SE进行标记。其中标记过程采用与SWITCH类似的机制,通过调节标记溶液的pH和脱氧胆酸钠溶液的浓度来影响探针-靶标的结合亲和力。具体来说,基于SE,利用D-山梨醇电催化氧化反应产生酸性产物以逐步改变缓冲液pH,同时利用纳米多孔膜维持标记液中脱氧胆酸钠溶液的浓度,进而逐步将探针-靶标结合条件由不利条件转变为有利条件。
5 整体组织器官成像技术
随着光学显微成像技术的飞速发展,目前已有许多商用的光学层析成像技术被用于组织的三维成像。其中,最常用的是共聚焦(单光子)显微镜和双光子显微镜。它们都以点扫描或线扫描的方式对样品成像,成像速度较慢。因此,这两种成像技术仅适用于小区域的高分辨率成像。如果将其用于较大的透明样本的成像,成像时间将会很长,此时不仅需要成像系统能够长时间稳定的成像,也对透明样本自身的形态与荧光信号保存提出了高要求。
近年发展的光片照明显微成像技术通过产生较薄的光片,激发一个薄平面,用相机(例如sCMOS)同时检测整个平面的发射荧光,再利用样本或光片的移动来实现轴向的分辨,从而获取组织三维结构信息[9,63,106]。不同于共聚焦/双光子的点扫描或线扫描,它采用面扫描成像的方式,极大地减少了大体积样本成像所需的时间。在光片照明显微镜中,光片的宽度决定了轴向分辨率,在光片宽度和成像视野之间也存在平衡。那些追求最薄的光片和最佳轴向分辨率的设计,例如晶格光片显微镜(lattice light-sheet microscopy)[10,107],只能在很小的区域内对几百微米厚的样本进行成像,而其他具有较厚光片的系统,例如DSLM[108]、Ultramicroscopy[63],则可以在更深、更大范围内实现对透明组织的成像,适用于那些不需要极高分辨率的成像。
在结合组织透明和光片照明实施三维结构成像的研究中,除了利用上述商用的光片系统外,为了满足实际应用中的透明样本的特定成像需求,研究人员也针对性地设计了一些特制的光片成像系统,例如COLM(CLARITY-Optimized Light-Sheet Microscopy),其利用同步照明-检测来提高图像质量[81]。Tomer等[109]设计了一种SPED (SPherical-Aberration-Assisted Extended Depth-of-Field)光片显微镜,通过扩展景深,并结合光片的光学层析能力,在三维成像时无需进行物镜的扫描,从而进一步提高了成像速度。最近,Dodt课题组[110]设计了一种新的光片显微系统——Aspheric Ultramicroscope,他们通过减少光片的半峰全宽,也实现了各向同性的成像分辨率。Voigt等[12]提出的mesoSPIM利用扫描振镜在垂直平面快速扫描高斯光束,生成薄的光片,并将轴向扫描光片显微镜整合进来,能在几分钟内对厘米尺度的样本进行成像,且各向的分辨率基本相同,其样本夹持器与样品池的更换是利用磁力进行的,操作极为快速、简便。
经典的光片系统设计采用的是侧面照明,还有一些光片系统的照明路径与样本表面成一定角度(例如45°),通常称这种系统为斜扫光片系统。将该类系统用于大组织成像时,需与光透明方法和机械切片结合,才能进行整体组织的成像。Osten课题组[111]提出了一种斜扫光片层析技术(OLST),并对CUBIC方法进行优化提出了mCUBIC方法,将其用于组织透明,能使组织保持一定硬度适合于切片制作。Bi课题组[112]也利用搭建的VISoR斜扫光片系统对CLARITY方法透明的切片样本进行了成像,并对切片的成像结果进行重构,获取了全脑的结构信息。
目前,商业的光片成像系统主要包括德国Lavision Biotec的 Ultramicroscope、德国Carl Zeiss的 Lightsheet Z.1、美国3i公司的Cleared Tissue/Lattice/Marianas LightSheet、德国Luxendo的MuVi/InVi/QuVi SPIM,及美国LifeCanvas Technologies的SmartSPIM等。
此外,近年发展的自动连续切片及成像技术,如序列双光子成像(STP)[24],也为大组织的成像提供了重要的手段。在利用STP进行大样本成像时,引入组织光透明方法,能够减少组织切片的次数,进而减少数据采集时间,并一定程度地减少切割伪影。此时,对透明方法的透明度要求降低,但需要其能使组织维持良好的结构稳定性和适当的硬度,以保证透明后的样本可用于后续的连续切削。2015年,Costantini等[113]利用STP,结合passive CLARITY方法与TDE(2,2'-thiodiethanol)试剂,以亚细胞分辨率重建了小鼠全脑。接着,Economo等[55]发展了一种基于DMSO/D-Sorbitol的透明方法,透明后的样本性能较稳定,利用该方法能在数天内对其进行稳定的切片与成像。除STP外,WVT(Wide-Field Large-Volume Tomography)[26]是另一种连续切片成像的技术,有望与组织光透明技术结合,从而达到加快成像速度的目的。采用ScaleS方法[79]得到的样品具有良好的透明度并能用于切削,且具有可连续切片成像的潜力。
除了光学层析成像外,另一种用于组织三维成像的方法是光学投影层析成像(OPT)[114-116],最早由James Sharpe提出,是一种基于可见光而非X射线的光学计算机断层扫描成像方法。基于透射和发射模式,采用OPT可以分别获得组织的结构和荧光信息。目前,已将其与组织光透明方法结合,用于3D成像。OPT能够以高分辨率(~5 μm)对完整的大样本(~1 cm3)进行成像,图像采集速度较快(一个小鼠脑15 min)。大多数OPT的成像研究采用BABB透明大体积样品。OPT已被应用到多种组织成像中,包括小鼠胚胎、小鼠器官及肿瘤组织。
![几种用于三维成像的典型光学显微成像技术[81,117]](/richHtml/zgjg/2020/47/2/0207007/img_7.jpg)
图 7. 几种用于三维成像的典型光学显微成像技术[81,117]
Fig. 7. Several typical optical microimaging technologies for 3D imaging of large samples[81,117]
![光透明结合光学成像技术获取整体器官三维结构信息[35,55,72,118]](/richHtml/zgjg/2020/47/2/0207007/img_8.jpg)
图 8. 光透明结合光学成像技术获取整体器官三维结构信息[35,55,72,118]
Fig. 8. 3D reconstruction of whole organs by combining tissue clearing and optical imaging techniques[35,55,72,118]
6 总结与展望
作为一门新兴的热门技术,组织光透明方法的发展十分迅速,其与多种标记及成像技术的结合,为高分辨地获取生物组织三维结构提供了重要的手段,在生物、医学相关的研究领域中发挥了重要的作用。本文从标记、透明与成像三个方面对整体器官的光透明成像方法进行了综述,期望为利用组织光透明开展相关工作的研究人员提供一定的参考。
可以预测,在未来,随着组织光透明方法的不断发展,以及标记、成像、数据处理方法的同步发展,有望将组织光透明成像应用到非人灵长类组织的研究中,更希望其在人体组织器官的基础研究、临床诊断中发挥重要作用,真正极大地推动生命科学的发展与进步。
[1] Kim S Y, Chung K, Deisseroth K. Light microscopy mapping of connections in the intact brain[J]. Trends in Cognitive Sciences, 2013, 17(12): 596-599.
[2] Richardson D S, Lichtman J W. Clarifying tissue clearing[J]. Cell, 2015, 162(2): 246-257.
[3] Susaki E A, Ueda H R. Whole-body and whole-organ clearing and imaging techniques with single-cell resolution: toward organism-level systems biology in mammals[J]. Cell Chemical Biology, 2016, 23(1): 137-157.
[4] 骆清铭. 脑空间信息学——连接脑科学与类脑人工智能的桥梁[J]. 中国科学: 生命科学, 2017, 47(10): 1015-1024.
Luo Q M. Brainsmatics-bridging the brain science and brain-inspired artificial intelligence[J]. Scientia Sinica(Vitae), 2017, 47(10): 1015-1024.
[5] Zipfel W R, Williams R M, Webb W W. Nonlinear magic: multiphoton microscopy in the biosciences[J]. Nature Biotechnology, 2003, 21(11): 1369-1377.
[6] Conchello J A, Lichtman J W. Optical sectioning microscopy[J]. Nature Methods, 2005, 2(12): 920-931.
[7] Helmchen F, Denk W. Deep tissue two-photon microscopy[J]. Nature Methods, 2005, 2(12): 932-940.
[8] Huisken J, Swoger J, Del Bene F, et al. Optical sectioning deep inside live embryos by selective plane illumination microscopy[J]. Science, 2004, 305(5686): 1007-1009.
[9] Verveer P J, Swoger J, Pampaloni F, et al. High-resolution three-dimensional imaging of large specimens with light sheet-based microscopy[J]. Nature Methods, 2007, 4(4): 311-313.
[10] Chen B C, Legant W R, Wang K, et al. Lattice light-sheet microscopy: imaging molecules to embryos at high spatiotemporal resolution[J]. Science, 2014, 346(6208): 1257998.
[11] Stefaniuk M, Gualda E J, Pawlowska M, et al. Light-sheet microscopy imaging of a whole cleared rat brain with Thy1-GFP transgene[J]. Scientific Reports, 2016, 6: 28209.
[12] Voigt F F, Kirschenbaum D, Platonova E, et al. The mesoSPIM initiative: open-source light-sheet microscopes for imaging cleared tissue[J]. Nature Methods, 2019, 16(11): 1105-1108.
[13] Tuchin V V. Light propagation in tissues with controlled optical properties[J]. Journal of Biomedical Optics, 1997, 2(4): 401-417.
[14] Tuchin V V. Optical clearing of tissues and blood using the immersion method[J]. Journal of Physics D: Applied Physics, 2005, 38(15): 2497-2518.
[15] Tuchin VV. Optical clearing of tissues and blood[M]. Bellingham: SPIE, 2005.
[16] Genina E A, Bashkatov A N, Tuchin V V. Tissue optical immersion clearing[J]. Expert Review of Medical Devices, 2010, 7(6): 825-842.
[18] Wang T, Ouzounov D G, Wu C Y, et al. Three-photon imaging of mouse brain structure and function through the intact skull[J]. Nature Methods, 2018, 15(10): 789-792.
[19] Li J B, Liu H W, Fu T, et al. Recent progress in small-molecule near-IR probes for bioimaging[J]. Trends in Chemistry, 2019, 1(2): 224-234.
[22] Miyamichi K, Amat F, Moussavi F, et al. Cortical representations of olfactory input by trans-synaptic tracing[J]. Nature, 2011, 472(7342): 191-196.
[23] Ertürk A, Bradke F. High-resolution imaging of entire organs by 3-dimensional imaging of solvent cleared organs (3DISCO)[J]. Experimental Neurology, 2013, 242: 57-64.
[25] Li A, Gong H, Zhang B, et al. Micro-optical sectioning tomography to obtain a high-resolution atlas of the mouse brain[J]. Science, 2010, 330(6009): 1404-1408.
[26] Gong H, Xu D L, Yuan J, et al. High-throughput dual-colour precision imaging for brain-wide connectome with cytoarchitectonic landmarks at the cellular level[J]. Nature Communications, 2016, 7: 12142.
[27] Luo Y L, Wang A L, Liu M M, et al. Label-free brainwide visualization of senile plaque using cryo-micro-optical sectioning tomography[J]. Optics Letters, 2017, 42(21): 4247-4250.
[28] Zhu D, Larin K V, Luo Q M, et al. Recent progress in tissue optical clearing[J]. Laser & Photonics Reviews, 2013, 7(5): 732-757.
[29] Yu T T, Qi Y S, Gong H, et al. Optical clearing for multiscale biological tissues[J]. Journal of Biophotonics, 2018, 11(2): e201700187.
[30] Vigouroux R J, Belle M, Chédotal A. Neuroscience in the third dimension: shedding new light on the brain with tissue clearing[J]. Molecular Brain, 2017, 10: 33.
[31] Miyawaki A. Brain clearing for connectomics[J]. Microscopy, 2015, 64(1): 5-8.
[32] Tainaka K, Kuno A, Kubota S I, et al. Chemical principles in tissue clearing and staining protocols for whole-body cell profiling[J]. Annual Review of Cell and Developmental Biology, 2016, 32(1): 713-741.
[33] Silvestri L, Costantini I, Sacconi L, et al. Clearing of fixed tissue: a review from a microscopist's perspective[J]. Journal of Biomedical Optics, 2016, 21(8): 081205.
[35] Chung K, Wallace J. KimS Y, et al. Structural and molecular interrogation of intact biological systems[J]. Nature, 2013, 497(7449): 332-337.
[36] Jing D, Zhang S W, Luo W J, et al. Tissue clearing of both hard and soft tissue organs with the PEGASOS method[J]. Cell Research, 2018, 28(8): 803-818.
[37] TainakaK, Murakami TC, Susaki EA, et al., 2018, 24(8): 2196-2210. e9.
[38] Moffitt J R, Hao J J, Bambah-Mukku D, et al. High-performance multiplexed fluorescence in situ hybridization in culture and tissue with matrix imprinting and clearing[J]. Proceedings of the National Academy of Sciences, 2016, 113(50): 14456-14461.
[41] Kim S Y, Cho J H, Murray E, et al. Stochastic electrotransport selectively enhances the transport of highly electromobile molecules[J]. Proceedings of the National Academy of Sciences, 2015, 112(46): E6274-E6283.
[42] Lee E, Choi J, Jo Y, et al. ACT-PRESTO: rapid and consistent tissue clearing and labeling method for 3-dimensional (3D) imaging[J]. Scientific Reports, 2016, 6: 18631.
[43] Greenbaum A, ChanK Y, Dobreva T, et al. 9(387): eaah6518[J]. imaging, computational analysis of osteoprogenitors within intact bone marrow. Science Translational Medicine, 2017.
[44] Zhao Y J, Yu T T, Zhang C, et al. Skull optical clearing window for in vivo imaging of the mouse cortex at synaptic resolution[J]. Light: Science & Applications, 2018, 7(2): 17153.
[45] Zhang C, Feng W, Zhao Y J, et al. A large, switchable optical clearing skull window for cerebrovascular imaging[J]. Theranostics, 2018, 8(10): 2696-2708.
[47] Kristinsson H G, Hultin H O. Changes in trout hemoglobin conformations and solubility after exposure to acid and alkali pH[J]. Journal of Agricultural and Food Chemistry, 2004, 52(11): 3633-3643.
[48] Treweek J B, Chan K Y, Flytzanis N C, et al. Whole-body tissue stabilization and selective extractions via tissue-hydrogel hybrids for high-resolution intact circuit mapping and phenotyping[J]. Nature Protocols, 2015, 10(11): 1860-1896.
[49] Susaki E A, Tainaka K, Perrin D, et al. Advanced CUBIC protocols for whole-brain and whole-body clearing and imaging[J]. Nature Protocols, 2015, 10(11): 1709-1727.
[50] Kubota S I, Takahashi K, Nishida J, et al. Whole-body profiling of cancer metastasis with single-cell resolution[J]. Cell Reports, 2017, 20(1): 236-250.
[51] Ke M T, Fujimoto S, Imai T. See DB: a simple and morphology-preserving optical clearing agent for neuronal circuit reconstruction[J]. Nature Neuroscience, 2013, 16(8): 1154-1161.
[52] Murray E. ChoJ H, Goodwin D, et al. Simple, scalable proteomic imaging for high-dimensional profiling of intact systems[J]. Cell, 2015, 163(6): 1500-1514.
[53] Tsai P S, Kaufhold J P, Blinder P, et al. Correlations of neuronal and microvascular densities in murine cortex revealed by direct counting and colocalization of nuclei and vessels[J]. The Journal of Neuroscience, 2009, 29(46): 14553-14570.
[54] Hou B, Zhang D, Zhao S, et al. Scalable and DiI-compatible optical clearance of the mammalian brain[J]. Frontiers in Neuroanatomy, 2015, 9: 19.
[55] Economo M N, Clack N G, Lavis L D, et al. A platform for brain-wide imaging and reconstruction of individual neurons[J]. eLife, 2016, 5: e10566.
[57] Lai H M. Liu A K L, Ng H H M, et al. Next generation histology methods for three-dimensional imaging of fresh and archival human brain tissues[J]. Nature Communications, 2018, 9: 1066.
[58] Chen L L, Li G Y, Li Y M, et al. Ubas M: an effective balanced optical clearing method for intact biomedical imaging[J]. Scientific Reports, 2017, 7: 12218.
[60] Ke M T, Nakai Y, Fujimoto S, et al. Super-resolution mapping of neuronal circuitry with an index-optimized clearing agent[J]. Cell Reports, 2016, 14(11): 2718-2732.
[61] Li W Z, Germain R N, Gerner M Y. Multiplex, quantitative cellular analysis in large tissue volumes with clearing-enhanced 3D microscopy (Ce3D)[J]. Proceedings of the National Academy of Sciences, 2017, 114(35): E7321-E7330.
[62] Ariel P. A beginner's guide to tissue clearing[J]. The International Journal of Biochemistry & Cell Biology, 2017, 84: 35-39.
[63] Becker K, Jährling N, Kramer E R, et al. Ultramicroscopy: 3D reconstruction of large microscopical specimens[J]. Journal of Biophotonics, 2008, 1(1): 36-42.
[64] Dodt H U, Leischner U, Schierloh A, et al. Ultramicroscopy: three-dimensional visualization of neuronal networks in the whole mouse brain[J]. Nature Methods, 2007, 4(4): 331-336.
[65] Jährling N, Becker K, Dodt H U. 3D-reconstruction of blood vessels by ultramicroscopy[J]. Organogenesis, 2009, 5(4): 227-230.
[66] Jährling N. Three-dimensional reconstruction and segmentation of intact Drosophila by ultramicroscopy[J]. Frontiers in System Neuroscience, 2010, 4: 1.
[67] Becker K, Jährling N, Saghafi S, et al. Chemical clearing and dehydration of GFP expressing mouse brains[J]. PLoS One, 2012, 7(3): e33916.
[69] Schwarz M K, Scherbarth A, Sprengel R, et al. Fluorescent-protein stabilization and high-resolution imaging of cleared, intact mouse brains[J]. PLoS One, 2015, 10(5): e0124650.
[70] Klingberg A, Hasenberg A, Ludwig-Portugall I, et al. Fully automated evaluation of total glomerular number and capillary tuft size in nephritic kidneys using lightsheet microscopy[J]. Journal of the American Society of Nephrology, 2017, 28(2): 452-459.
[71] MasselinkW, ReumannD, MurawalaP, et al., 2019, 146(3): dev166884.
[72] Pan C C, Cai R Y, Quacquarelli F P, et al. Shrinkage-mediated imaging of entire organs and organisms using uDISCO[J]. Nature Methods, 2016, 13(10): 859-867.
[73] Belle M, Godefroy D, Couly G, et al. 169(1): 161-[J]. analysis of early human development. Cell, 2017, 173: e12.
[74] Renier N, Adams E L, Kirst C, et al. Mapping of brain activity by automated volume analysis of immediate early genes[J]. Cell, 2016, 165(7): 1789-1802.
[75] Wang Q, Liu K L, Yang L, et al. BoneClear: whole-tissue immunolabeling of the intact mouse bones for 3D imaging of neural anatomy and pathology[J]. Cell Research, 2019, 29(10): 870-872.
[76] Cai R Y, Pan C C, Ghasemigharagoz A, et al. Panoptic imaging of transparent mice reveals whole-body neuronal projections and skull-meninges connections[J]. Nature Neuroscience, 2019, 22(2): 317-327.
[77] Hildebrand S, Schueth A, Herrler A, et al. Scalable labeling for cytoarchitectonic characterization of large optically cleared human neocortex samples[J]. Scientific Reports, 2019, 9: 10880.
[78] Hama H, Kurokawa H, Kawano H, et al. Scale: a chemical approach for fluorescence imaging and reconstruction of transparent mouse brain[J]. Nature Neuroscience, 2011, 14(11): 1481-1488.
[79] Hama H, Hioki H, Namiki K, et al. ScaleS: an optical clearing palette for biological imaging[J]. Nature Neuroscience, 2015, 18(10): 1518-1529.
[80] Murakami T C, Mano T, Saikawa S, et al. A three-dimensional single-cell-resolution whole-brain atlas using CUBIC-X expansion microscopy and tissue clearing[J]. Nature Neuroscience, 2018, 21(4): 625-637.
[82] Epp JR, NiiboriY, ( Liz) Hsiang HL, et al. and other intact organs[J].Eneuro, 2015, 2(3): ENEURO. 0022-15. 2015.
[83] Bastrup J, Larsen P H. Optimized CLARITY technique detects reduced parvalbumin density in a genetic model of schizophrenia[J]. Journal of Neuroscience Methods, 2017, 283: 23-32.
[84] Ren J, Choi H, Chung K, et al. Label-free volumetric optical imaging of intact murine brains[J]. Scientific Reports, 2017, 7: 46306.
[85] Ku T, Swaney J, Park J Y, et al. Multiplexed and scalable super-resolution imaging of three-dimensional protein localization in size-adjustable tissues[J]. Nature Biotechnology, 2016, 34(9): 973-981.
[86] Park Y G, Sohn C H, Chen R, et al. Protection of tissue physicochemical properties using polyfunctional crosslinkers[J]. Nature Biotechnology, 2019, 37(1): 73-83.
[88] Sylwestrak E L, Rajasethupathy P, Wright M A, et al. Multiplexed intact-tissue transcriptional analysis at cellular resolution[J]. Cell, 2016, 164(4): 792-804.
[89] Qi YS, Yu TT, Xu JY, et al., 2019, 5(1): eaau8355.
[91] Diekmann H, Kalbhen P, Fischer D. Characterization of optic nerve regeneration using transgenic zebrafish[J]. Frontiers in Cellular Neuroscience, 2015, 9: 118.
[92] Frétaud M, Rivière L, De Job É, et al. High-resolution 3D imaging of whole organ after clearing: taking a new look at the zebrafish testis[J]. Scientific Reports, 2017, 7: 43012.
[93] Hsiao P Y, Tsai C L, Chen M C, et al. Non-invasive manipulation of Drosophila behavior by two-photon excited red-activatable channelrhodopsin[J]. Biomedical Optics Express, 2015, 6(11): 4344-4352.
[94] Žygelyté E, Bernard M E, Tomlinson J E, et al. RetroDISCO: clearing technique to improve quantification of retrograde labeled motor neurons of intact mouse spinal cords[J]. Journal of Neuroscience Methods, 2016, 271: 34-42.
[95] Liu A K L, Hurry M E D, Ng O T W, et al. Bringing CLARITY to the human brain: visualization of Lewy pathology in three dimensions[J]. Neuropathology and Applied Neurobiology, 2016, 42(6): 573-587.
[96] Grist S M, Nasseri S S, Poon T, et al. On-chip clearing of arrays of 3-D cell cultures and micro-tissues[J]. Biomicrofluidics, 2016, 10(4): 044107.
[97] Silva Santisteban T, Rabajania O, Kalinina I, et al. Rapid spheroid clearing on a microfluidic chip[J]. Lab on a Chip, 2018, 18(1): 153-161.
[98] Chen Y Y, Silva P N, Syed A M, et al. Clarifying intact 3D tissues on a microfluidic chip for high-throughput structural analysis[J]. Proceedings of the National Academy of Sciences, 2016, 113(52): 14915-14920.
[99] Zhang Y B, Shin Y, Sung K, et al. 3D imaging of optically cleared tissue using a simplified CLARITY method and on-chip microscopy[J]. Science Advances, 2017, 3(8): e1700553.
[100] Liebmann T, Renier N, Bettayeb K, et al. Three-dimensional study of alzheimer's disease hallmarks using the iDISCO clearing method[J]. Cell Reports, 2016, 16(4): 1138-1152.
[101] Kim SH, CheP, Chung SH, et al., 2006, 36(1): 2. 10.1-2.10. 9.
[103] Gleave J A, Lerch J P, Henkelman R M, et al. A method for 3D immunostaining and optical imaging of the mouse brain demonstrated in neural progenitor cells[J]. PLoS One, 2013, 8(8): e72039.
[104] Seo J, Choe M, Kim S Y. Clearing and labeling techniques for large-scale biological tissues[J]. Molecules and Cells, 2016, 39(6): 439-446.
[105] Li J, Czajkowsky D M, Li X W, et al. Fast immuno-labeling by electrophoretically driven infiltration for intact tissue imaging[J]. Scientific Reports, 2015, 5: 10640.
[107] GaoR, Asano SM, UpadhyayulaS, et al. and whole-brain imaging with molecular contrast and nanoscale resolution[J].Science, 2019, 363(6424): eaau8302.
[108] Keller P J, Schmidt A D, Wittbrodt J, et al. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy[J]. Science, 2008, 322(5904): 1065-1069.
[109] Tomer R, Lovett-Barron M, Kauvar I, et al. SPED light sheet microscopy: fast mapping of biological system structure and function[J]. Cell, 2015, 163(7): 1796-1806.
[110] Pende M, Becker K, Wanis M, et al. High-resolution ultramicroscopy of the developing and adult nervous system in optically cleared Drosophila melanogaster[J]. Nature Communications, 2018, 9: 4731.
[112] Wang H, Zhu Q Y, Ding L F, et al. Scalable volumetric imaging for ultrahigh-speed brain mapping at synaptic resolution[J]. National Science Review, 2019, 6(5): 982-992.
[113] Costantini I. Ghobril J P, di Giovanna A P, et al. A versatile clearing agent for multi-modal brain imaging[J]. Scientific Reports, 2015, 5: 9808.
[116] Sharpe J, Ahlgren U, Perry P, et al. Optical projection tomography as a tool for 3D microscopy and gene expression studies[J]. Science, 2002, 296(5567): 541-545.
[117] Osten P, Margrie T W. Mapping brain circuitry with a light microscope[J]. Nature Methods, 2013, 10(6): 515-523.
[118] Oldham M, Sakhalkar H, Oliver T, et al. Optical clearing of unsectioned specimens for three-dimensional imaging via optical transmission and emission tomography[J]. Journal of Biomedical Optics, 2008, 13(2): 021113.
Article Outline
俞婷婷, 朱丹. 整体器官的光透明成像方法综述[J]. 中国激光, 2020, 47(2): 0207007. Yu Tingting, Zhu Dan. Review of Tissue Optical Clearing Methods for Imaging Whole Organs[J]. Chinese Journal of Lasers, 2020, 47(2): 0207007.

![大组织的整体免疫标记策略[52,104]](/richHtml/zgjg/2020/47/2/0207007/img_6.jpg)





