基于IABC-SVR算法的拉曼光谱定量分析山羊血清蛋白含量
1 引言
血清总蛋白(total protein, TP)是血清中各种蛋白质的总称, 是各种蛋白的复杂混合物[1], 包含白蛋白、 α1球蛋白、 α2球蛋白、 β球蛋白等, 是人体保持健康的重要组成部分。 人体生理机能的变化会引起血浆中蛋白质发生质和量的变化, 检测血清生化成分, 可作为协助诊断肿瘤、 肝病、 肾病综合征等疾病的方法[2]。 例如, 郭丽等通过对比健康人和患者的血清拉曼光谱, 为乳腺癌的诊测提供了新的方法。 王玉等[3]利用不同批次人血白蛋白的拉曼光谱, 结合拉曼光谱峰位移和峰强度的相似度计算结果, 来判断血液白蛋白样品的真假伪劣。 本文提出了一种基于拉曼光谱和改进人工蜂群算法优化支持向量机回归(IABC-SVR)算法快速定量检测山羊血清蛋白含量的方法。
血清总蛋白测定方法有很多, 最主要的有凯氏定氮法、 双缩脲法、 酚试剂法等, 目前检测血清蛋白总量的常规方法为双缩脲法[4], 其原理是蛋白质分子中的肽键(—CO—NH—)与双缩脲试剂反应, 形成紫色化合物, 其紫色深浅程度与样品中蛋白质浓度成正比, 根据所测样品吸光度值, 在标准曲线上查出相应蛋白质含量, 从而计算出样品溶液蛋白质含量。 该方法需消耗多种试剂, 步骤较繁琐, 存在污染试剂、 精度差、 容易交叉污染等缺点。 而拉曼光谱技术是一种非破坏性测试技术, 几乎无需试样制备, 用很少量的试样就能获得足够的信号, 所表征的是分子振动的信息, 水的拉曼散射极微弱, 因而水溶液样品可直接进行测量, 这对生物大分子的研究非常有利。 从拉曼光谱的谱图中可以得到频率、 强度、 偏振特性、 峰形等信息, 在生物、 材料、 环保、 地质等领域得到应用。 这种生化法拉曼光谱技术具有灵敏度高、 无需样品预处理、 操作简便、 信息含量丰富、 能够获取分子的结构信息等特点。 本文将拉曼光谱技术与IABC-SVR算法相结合, 对山羊血清蛋白总量进行快速定量分析。
1 模型原理和方法
1.1 支持向量机回归
支持向量机分类(support vector machine, SVM)于1995年由Vapnik提出, 支持向量机回归(support vector regression, SVR)是在SVM的基础上发展起来的, 是将低维空间的非线性问题通过核函数映射到高维空间, 使其在高维空间内成为一个线性问题, 从而使问题的求解得到了简化[5]。 设原有空间训练样本(x1, y1), (x2, y2), …, (xi, yi), (i=1, 2, …, l), xi(xi∈Rd)是第i个训练集样本的输入列向量, xi=[ , , …, ]T, yi∈R为对应的输出值。 利用非线性映射函数Φ(x)变换将l个训练样本映射到高维特征空间, 基于结构风险最小化的原则建立高维空间线性回归函数为
(1)
式(1)中, b为阈值; αi和 为拉格朗日乘子; K(xi, xj)为核函数, 本文选用径向基函数作为核函数。
(2)
约束条件为 为惩罚参数。
1.2 改进的人工蜂群算法
标准人工蜂群算法(artificial bee colony, ABC)是由土耳其学者Karaboga于2005年提出模拟蜜蜂寻觅花蜜过程的一种群体智能算法[6]。 在人工蜂群的框架下, 蜜源的位置表示要优化问题的可能候选解, 蜜源中花蜜的数量与候选解的适应度(目标值函数)相对应, 而蜂群的规模等于待优化解的数量。 ABC算法更新种群采用随机选取的方法, 在ABC算法搜索过程中存在收敛速度较慢、 不能实现全局收敛的问题, 针对此问题, 提出改进的人工蜂群算法(improved artificial bee colony, IABC), 在对蜜源更新公式中引入柯西变异算子[7]。 同时改进随机解生成方法, 随着迭代次数增加, 步长从一个较小的数逐渐增长到一个较大数值。 改进后的算法保证模型前期能够进行全面的局部搜索, 且后期保持全局搜索能力, 并加快收敛速度。
设初始种群含有NP个解, 每个解xi(i=1, 2, …, NP)为一个d维向量, 对应的解的适应度值fit, 个体最大更新次数为G, 最大迭代次数为T。 具体步骤为
第1步: 初始阶段, 随机产生蜜源的初始位置, 即
(3)
第2步: 初始蜜源周围搜索产生一个新的蜜源, 即
(4)
其中, Cij为对应xij服从柯西分布C(0, 1)的随机数, k∈{i=1, 2, …, NP}, j∈{1, 2, …, d}且k≠i。 φij为[-1, 1]内的随机数。
第3步: 评价蜜源的适用度, 如果新的蜜源适应度高于原先的蜜源, 则新的蜜源代替原先的蜜源, 否则保留原先的蜜源, 即
(5)
其中fi为解的函数值。 此外, 基于轮盘赌原则, 找到新蜜源被跟随的概率Pi为
(6)
某蜜源经过G次循环后没有得到优化, 则放弃该蜜源寻找下一个。 若运算迭代次数达到T或者最优适应度, 运算结束, 否则返回式(4), 迭代出最优解。
2 实验部分
2.1 仪器与样品
本次实验使用的山羊血清为经过无菌过滤处理的山羊血清, 血清蛋白初始浓度为0.042 34 g·mL-1。 实验中用于稀释血清浓度的试剂为生理盐水, 生理盐水对实验结果影响极小且不影响生物样本的活性。 按体积比配置不同浓度的血清蛋白样本, 配置的单个液体样本体积为3 mL, 共计35组, 随机选取8组实验样本作为模型测试集, 剩余27组作为模型训练集。 配置好的样本在4 ℃恒温箱中静置24 h, 确保样本混合均匀。 光谱测量过程中易受外界光干扰, 所以在实验中进行避光处理, 防止外界干扰。
本次实验使用的是BWS465-785S型拉曼光谱仪, 由美国必达泰克公司生产, 光谱测量范围0~3 500 cm-1, 激发光源波长为785 nm, 设置拉曼光谱激光功率为300 mW, 每个样本连续扫描取光谱稳定后的数值, 积分时间10 000 ms, 光谱仪的采集分辨率为5 cm-1。
2.2 光谱采集与数据处理
使用拉曼光谱仪采集样本拉曼光谱, 通过BWRam4软件进行光谱读取。 测量光谱前, 将拉曼光谱仪打开预热20 min。 用超纯水清洗样品池, 控干池内水分, 并使用擦镜纸清理样品池表面(测量各组样品前重复此步骤)。 将试剂样本缓慢移入样品池, 避免气泡进入, 放入遮光罩中进行避光处理, 待样品静置2 min后, 进行光谱采集。 拉曼光谱处理过程如图1所示。
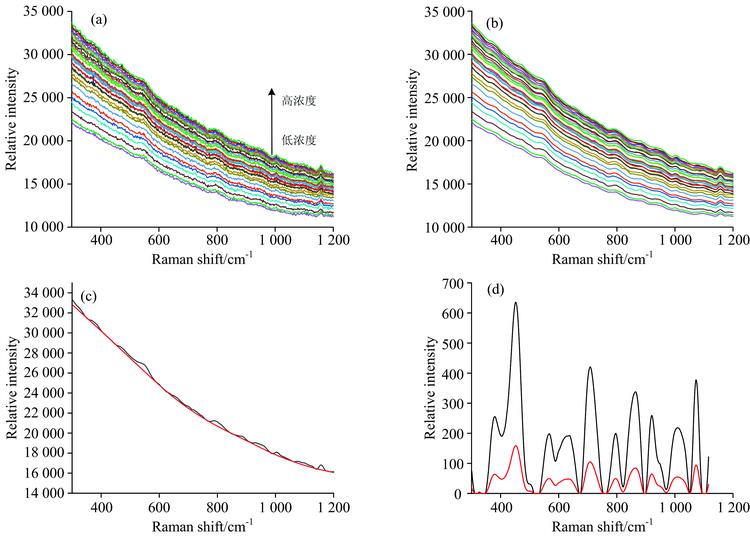
图 1. 拉曼光谱处理过程
(a): 原始拉曼光谱; (b): 平滑处理; (c): 背景基线扣除; (d): 归一化处理
Fig. 1. Raman processing
(a): 26 groups of original Raman spectra; (b): Smoothing processing; (c): Background baseline deduction; (d): Normalization
通过对图1分析可知, 随着样品中血清蛋白浓度变化, 拉曼光谱原始数据强度发生明显变化。 原始光谱信噪比较高, 影响了对特征峰的观察和数据处理。 在建模之前对数据进行预处理, 随机选取一条拉曼光谱为例进行处理。
每组样品采集前进行暗电流采集并扣除。 采集的原始拉曼光谱如图1(a)所示(选取光谱范围300~1 300 cm-1)。 为了消除CCD噪声、 采集电路的噪声以及激光功率抖动等对拉曼信号强度的影响, 同时简化计算量, 采用平滑处理、 背景扣除、 归一化三种预处理方法对测得的原始拉曼光谱进行数据预处理。 平滑采用Savitzky-Golay平滑法进行处理, 该方法在滤除噪声的同时可保持原信号谱宽、 强度及形状不发生改变, 其滤波原理是局部多项式时域最小二乘法拟合[8], 处理结果如图1(b)所示; 背景扣除采用扣除基线的方法, 采用非对称最小二乘平滑建立基线, 可将有效拉曼信号保存下来, 去除掉背景荧光信号如图1(c)所示, 黑色线代表拉曼光谱数据, 红色线代表需要扣除的基线; 数据归一化处理的作用是简化计算, 缩小量值, 提高数学预测模型的准确度, 如图1(d)所示, 黑色线代表拉曼光谱扣除基线后的数据, 红色线代表执行归一化处理后的数据。
2.3 特征峰归属
经过处理的山羊血清样本拉曼光谱, 在379.05, 451.87, 568.07, 636.12, 709.73, 797.08, 864.21, 920.15, 1 009.95和1 072.24 cm-1处表现出明显的特征峰, 如图1(d)所示。 通过比对, 对山羊血清蛋白特征峰进行归属[9,10], 峰值标记如图2所示, 特征峰归属如表1所示。
表 1. 血清蛋白特征峰及其归属
Table 1. Characteristic peak of serum protein and its attribution
|
3 结果与讨论
在35组实验样品中随机选取27组样本作训练集, 通过拉曼光谱的数据采集及预处理, 提取的特征峰强度值和训练集已知浓度信息作为输入, 余下8组样本作为测试集, 同训练集一样, 将特征峰强度作为模型的输入, 通过模型运算的浓度信息作为输出。 分别建立基于改进人工蜂群算法优化支持向量机回归算法(IABC-SVR)的预测模型和人工蜂群算法优化支持向量机回归算法(ABC-SVR)模型, 并与经典BP(back propagation)神经网络算法建模结果比较。 IABC属于迭代优化, 可优化SVR中的惩罚因子C和函数参数σ, 以提升该模型的泛化能力及预测精度。 建立IABC-SVR模型流程如图3所示。
将测试集已知血清蛋白浓度与预测结果进行均方差运算来评价模型精准度, 相关系数来评价模型预测关联度。 样本为M的均方误差定义为
(7)
式(7)中, M为样本个数; yi为实际值; 为预测值。
均方根误差可以显示出预测结果与真实结果平方和的平均数, 由于对误差进行了平方处理, 就避免了误差出现正负值而相抵消的情况, 从而提高了对误差分析的准确性。 相关系数r定义为
(8)
r值越接近 1, 则反映定量预测模型精度越高, 预测集建模相关度曲线如图4所示, 相关系数为0.990 27, 显示出极高的相关性, 表明模型结果较好。 预测结果残差如图5所示, 残差均小于0.001 g·mL-1, 预测准确率为99.8%。

图 4. 模型测试集中真实值与预测值相关度拟合
Fig. 4. Correlation curve between actual value and predicted value in model test set
将数据分别导入ABC-SVR 算法和BP神经网路算法进行建模, 并将预测模型进行对比, 建模结果如图6所示, 得到的各模型均方差(MSE), 相关系数(r)与建模时间(Time)如表2所示。

图 6. 三种模型预测结果中真实值与预测值相关度曲线
Fig. 6. Correlation curve between actual value and predicted value in three models’ prediction results
表 2. 三种建模方法模型评价结果
Table 2. Evaluation results of three modeling methods
|
由表2可知, 经典BP神经网络算法的预测精度低于ABC-SVR算法建模精度, IABC-SVR算法预测模型在ABC-SVR算法基础上有明显提升, 说明IABC-SVR算法能较好的避免局部最优解, 有较好的全局搜索能力。 建模时间为1.9 s, 具有更好的工作效率。 由实验结果可以看出, 文中提出的基于IABC-SVR算法预测模型定量分析山羊血清蛋白总量的方法能有效实现预期功能, 并在ABC-SVR算法基础上提高了精度, 加快了算法收敛运算速度, 达到了更好的效果。
4 结论
提出了一种基于拉曼光谱和IABC-SVR算法对山羊血清蛋白含量快速、 无损的定量检测方法。 拉曼光谱仪采集实验样本光谱, 结合三种光谱预处理的方法对原光谱进行处理, 标定的光谱特征峰强度作为模型输入; 针对传统人工蜂群算法在区域规模较大时收敛速度逐渐减慢, 会出现效率低、 精准度下降、 局部最优解概率高等弊端, 改进了蜂群的初始化和位置更新方法, 旨在提高预测模型的全局搜索能力和准确性; 建立了IABC-SVR山羊血清蛋白定量检测模型, 对山羊血清蛋白总含量进行定量分析。 结果表明, 该方法能快速、 准确、 无损的预测山羊血清蛋白总量, 具有较高的可行性。 建立模型的相关系数为0.990 27, 均方误差为0.244 3, 建模时间为1.9 s, 预测准确率为99.8%。 说明该方法能较好的完成山羊血清蛋白总量的快速定量分析。 在下一步的实验中, 将样品的浓度梯度进一步细化, 增加模型的训练集和测试集, 提升模型的精度和预测能力。
Article Outline
付兴虎, 赵飞, 王振兴, 芦鑫, 付广伟, 金娃, 毕卫红. 基于IABC-SVR算法的拉曼光谱定量分析山羊血清蛋白含量[J]. 光谱学与光谱分析, 2021, 41(2): 540. Xing-hu FU, Fei ZHAO, Zhen-xing WANG, Xin LU, Guang-wei FU, Wa JIN, Wei-hong BI. Quantitative Analysis of Goat Serum Protein Content by Raman Spectroscopy Based on IABC-SVR[J]. Spectroscopy and Spectral Analysis, 2021, 41(2): 540.






