基于改进ResNeXt的乳腺癌组织病理学图像分类  下载: 1048次
下载: 1048次
1 引言
钼靶成像、核磁共振成像、X射线(X-ray)等技术,可对大量病理学图像做出准确的分期、分级,从而对乳腺癌进行诊断。相比人工诊断方法,计算机辅助诊断不仅可以提高诊断效率,还增加了客观性的判断,具有重要的临床应用价值。对组织病理图像的分类难点一方面在于细胞核重叠会影响单独细胞核的特征,导致有些核特征不明显,不便于用肉眼观察到,若染色不均匀,还会增加分类难度;另一方面,1张病理图像中可能包含多种类型的细胞核,这对判断整张图像处于哪一类有很大的影响。此外,目前公开的数据集都是小型数据集,对基于深度学习的分类方法具有很大的挑战。
Spanhol等[1]在BreaKHis数据集上,采用支持向量机(SVM)、随机森林等分类算法进行乳腺癌组织病理图像的分类,准确率为80%~85%;随后Spanhol等[2]在相同数据集上,用一种改进的AlexNet对组织病理图像进行分类,在图像水平级四分类任务中的准确率达到89.6%;Araújo等[3]提出了卷积神经网络(CNN)结合SVM(CNN+SVM)的方法并在2015乳腺癌分类挑战数据集上进行实验,得到四分类任务的准确率为77.8%;Golatkar等[4]采用Inception-v3网络的迁移学习,对400张均匀划分的四种类别图像进行预处理和补丁提取,采用多数表决决策在四分类任务中的准确率达到85%;Rakhlin等[5]采用多种深层神经网络结构和梯度提升树(LightGBM)分类器,在文献[ 4]使用的数据集上得到的四分类任务准确率为87.2%;Koné等[6]采用二叉树思想,首先对病理图像进行二分类,再对每一类进行更精细的二分类以达到四分类的目的,用提出的分层深度残差网络(ResNeXt)对三个不同的网络进行训练,得到四分类任务的准确率为99%;Nazeri等[7]提出了一种基于补丁的技术,由两个连续的CNN组成,在四类病理图像上的分类准确率为94%;Wang等[8]用SVM算法对68张乳腺癌组织病理图像的分类准确率为96.19%;Krizhevsky等[9]采用谷歌网络(GoogLeNet)进行迁移学习,在BreakHis数据集上的分类识别率为91%。上述部分方法的最终准确率因数据集和评价决策标准而不同,因此不具有可比性。
采用传统方法进行特征提取时,由于相近类间的细胞特征形态相似性较大,导致特征提取难度大、算法的准确率较低;传统的CNN虽然准确率较高,但也会带来较高的特征冗余,增加模型的计算量;迁移学习方法中卷积核的数量会影响对图像中特征的学习效果,且大多数训练网络比较单一,学到的特征权重不能很好地适用于组织病理图像特征的提取,导致识别率较低。针对上述问题,本文提出了一种更深、更有效的CNN,同时采用一种有效的数据增强方法,避免了数据量过少造成的过拟合现象,从而提高模型的分类准确率。
2 本文方法
CNN作为图像处理领域中一种高效的识别方法[9-12],有很多主流的CNN框架得到了广泛应用。相比传统的残差网络,实验采用的ResNeXt[13]模型计算量更少,训练精度更高;同时用八度卷积(OctConv)[14]代替传统卷积层,可以有效提取组织病理图像中的高频特征,降低低频特征的信息权重,有利于对相近类别的图像进行识别;此外,还引入异构卷积(HetConv)[15]降低训练参数并提高模型的分类准确率。
2.1 网络架构的组成
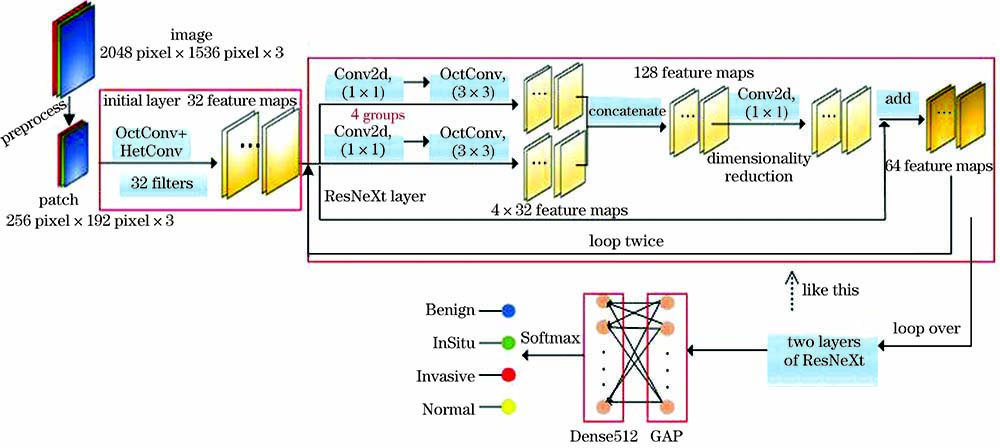
用OctConv替换传统卷积层,由于卷积核为1×1的卷积层具有升维或降维的作用,用OctConv替换后只会增加训练参数和训练时间,因此,只替换卷积核为3×3的卷积层。初始层OctConv中基础卷积层的卷积核采用HetConv,以减少训练参数,提高分类准确率。本网络的整体流程结构如
2.1.1 初始层
初始层为32个卷积核大小为3×3、步长为1的卷积层,输入尺寸为256pixel×192pixel×3的张量,其中,3为通道数。然后进行批标准化(BN)[16],用线性整流函数(ReLU)[17]进行激活,实现初步的特征提取。
2.1.2 OctConv模块
图像中的高频部分含有大量的细节信息,低频部分则体现图像的大体信息,决定性信息较少。Chen等[14]用OctConv替代传统CNN中的卷积层,将特征图分为高频通道和低频通道,再将低频通道的特征图按比例缩减一半,即将特征图分为高频和低频两个部分,如
相近种类的组织病理图像之间具有很多相同的特征,且很难提取各自的细节特征,因此,引入OctConv网络模块,以提高对高频信息的提取效率,降低冗余的低频信息,进而提高对相近种类的识别准确率。设计的OctConv结构包括初始层、过渡层和输出层。其中,初始层是单输入双输出,负责接收输入特征图。原始图像经过一层卷积核大小为3×3的卷积层输出高频特征图(XH),对原始图像进行平均池化后经过相同的卷积层输出低频特征图(XL)。低频通道数为Ffilters×α,高频通道数为1-Ffilters×α,其中,Ffilters为输入通道数,实验取64。为降低低频特征冗余,参数α的取值范围为0~0.5,且α为0.125的整数倍。经过对比实验,最终将α取为0.25;过渡层是双输入双输出,以XH、XL为输入,高频特征XH和低频特征XL再经过一层卷积层后分别进行下采样和上采样操作输出YH、YL;输出层是双输入单输出,输入为YH、YL,将过渡层的输出YL经一层卷积层后进行上采样,然后与YH经过一层卷积层后得到的特征图进行相加操作,得到该模块的输出特征图。
2.1.3 HetConv模块
与传统卷积结构不同,HetConv以一种新的滤波器思想作用在卷积层中。同构卷积包含卷积核大小相同的卷积层,如一层二维(2D)卷积包含256个大小为3×3的过滤器,如

图 3. 不同结构的卷积。(a)传统卷积;(b) HetConv
Fig. 3. Convolution with different structures. (a) Traditional convolution; (b) HetConv
设卷积输出特征图的尺寸为
将初始层的卷积核设计为异构核形式,用根据HetConv原理设计的算法代替传统卷积核,以达到卷积核异构的目的。算法的流程如
2.1.4 残差层
ResNeXt[18]是一个高度模块化的网络结构,实验设计的ResNeXt模块将卷积核为1×1和3×3的卷积层进行堆叠,由四个相同的结构并行输入共同输出,用卷积核为1×1的卷积层作为ResNeXt模块的最后一层,再用最后一层的输出与ResNeXt模块的输入进行add操作,最终形成一个完整的ResNeXt模块,如
2.1.5 全局平均池化层
用全局平均池化 (GAP)层[19]替代传统的全连接层,GAP层后是一个包含512个节点的Dense层。Dense层可将上一层的输出特征转化为N×1的1维向量综合起来,获取的图像特征具有的高层含义。为避免过拟合,在Dense层后再加入一个Dropout层[20],经Softmax分类器输出。选用RAdam(Rectified Adam)优化器进行优化[21],一方面,实验数据集较小,相比Adam优化器,RAdam优化器可以省去预热的步骤[22];另一方面,RAdam优化器对学习率的鲁棒性较好,同时具有与Adam优化器相同的收敛速度,可以避免陷入局部最优解。
2.2 数据预处理
2.2.1 数据集
在Grand Chanllenge的BACH公开数据集[23]上进行实验,该数据集包括四类正常组织(Normal)、良性病变(Benign)、原位癌(InSitu)、浸润性癌(Invasive)经过H&E (Hematoxylin-Eosin)染色的乳房组织病理学显微镜图像,图像的尺寸为2048pixel×1536pixel,均为RGB(Red,Green,Blue)图像,每个像素覆盖0.42μm×0.42μm的组织。该数据集包含两位病理学家共同给出的注释,存在分歧的图像已经被丢弃。为使数据均匀,从每类图像中选取100张图像进行实验。
2.2.2 预处理
数据预处理[24-26]是图像处理中必不可少的步骤,由于选取的数据集样本数量有限,为防止模型训练过拟合,从每张图像中提取小斑块(patch),以增加数据集的样本数量,同时只保留具有细胞核的patch,丢弃无细胞核或细胞核数较少的patch。
由于细胞核的大小和形状及其周围的组织结构是分类的主要特征[27-31]。其中,正常组织经过H&E染色后细胞质区域较大,且有密集的成核簇;良性病变由多个相邻的核簇组成;原位癌表现为细胞核增大、核仁突出,但均在一个圆形的团簇中;浸润性癌打破了原位癌的团簇形式,癌细胞核扩散到附近区域且核密度较高,细胞核间排列杂乱。因此,实验提取尺寸为256 pixel×192 pixel的区域,可以很好的包含团簇的轮廓、细胞核及其周围结构。为了得到更全面的图像特征信息,每个patch需要覆盖提取图像50%的区域,即宽度上的步长Sweight为128pixel,高度上的步长Shight为96pixel,在宽度上能够提取的patch数为WT,在高度上能提取的patch数为HT,可表示为
式中,Tall为从每张完整图像提取的patch。实验不使用所有的patch,仅保留核密度较高的patch,丢弃核密度稀疏的patch,保留与丢弃patch的标准参考文献[
4],提取得到所有patch的标签与原始图像的标签保持一致。经H&E染色的病理学图像中细胞核的边缘特征不突出,因此,对所有patch进行对比度拉伸处理,使细胞核及其周围特征更明显,如

图 6. 验证集中Benign类的图像。(a)整张图像;(b)小斑块
Fig. 6. Image of the Benign class in the verification set. (a) Whole image; (b) small patches
2.2.2 多数投票原则
每张图像的任意patch经过Softmax分类器都会得到一个类别的输出,将对应类别数量加1。当该图像提取的所有patch中属于某一类的patch样本数相对较多时,将该图像判定为这一类,分类原理如
3 实验结果与分析
3.1 实验环境
实验使用Python语言基于Keras框架进行编程,实验平台为DGX的Ubuntu系统NVIDIA v100。在CPU环境下进行数据预处理,在GPU上训练CNN模型以加快数据的并行计算,提高实验效率。
3.2 训练策略
将数据集每一类均匀地分为训练集(60%)、验证集(20%)和测试集(20%),将提取的patch作为最终的训练集、验证集和测试集。其中,训练集用于模型训练和参数学习;验证集用于验证模型,通过训练不断验证模型的泛化能力并自动微调参数,随时保存最佳模型;测试集用于测试模型的识别率以及泛化能力,训练时将所有训练数据打乱后再进行处理。
训练在四分类任务下进行,即正常、良性、原位癌和浸润性癌四类。训练策略:首先在原始ResNeXt中训练模型;再用OctConv替换ResNeXt中的3×3卷积层重新训练;最后用HetConv将初始层的传统卷积核替换后重新训练。前一模型训练得到的数据可为后一模型的优化提供有效的判断依据,并作为对比实验。
3.3 评价标准
实验结果的评价指标为patch的识别率和整张图像的识别率,patch的识别率
式中,
式中,
3.4 实验结果与分析
用训练patch的准确率和对整张图像的分类准确率(测试集中每类图像的数量为20张)评价本网络模型的性能,且前者的准确率很大程度地影响了后者的准确率。在三种改进的网络中依次进行实验,达到依次递增的实验效果。分别以训练集的准确率(acc)和验证集的准确率(val_acc)、测试集整张图像的混淆矩阵分析实验结果。
3.4.1 ResNeXt
ResNeXt由包含32个滤波器的卷积层(输入层)、三个ResNeXt模块、GAP层、一个Dense层、一个Dropout层及Softmax分类器组成,整体结构如

图 8. ResNeXt模型的训练准确率和验证准确率
Fig. 8. Training accuracy and verification accuracy of the ResNeXt model
由于实验采用多数决策原则,因此验证集的准确率并不能表示整张图像的判别准确率,验证集的准确率基于patch级别,而图像级别的准确率由该张图像中所有patch的最多数类别决定,最终结果如
表 1. ResNeXt模型的图像级混淆矩阵
Table 1. Image-level confusion matrix of ResNeXt
|

图 9. 部分判断错误的图像。 (a) Invasive; (b) InSitu1; (c) InSitu2
Fig. 9. Image of partially judged wrong. (a) Invasive; (b) InSitu1; (c) InSitu2
3.4.2 ResNeXt+OctConv模型
ResNeXt+OctConv模型以ResNeXt为基础,将其中的传统卷积替换为OctConv模块。OctConv模块能有效提取高频信息,适当弱化低频信息,该网络在验证集和训练集中的准确率如

图 10. ResNeXt +OctConv模型的训练准确率和验证准确率
Fig. 10. Training accuracy and verification accuracy of the ResNeXt+OctConv model
ResNeXt+OctConv模型的图像级别混淆矩阵如
表 2. ResNeXt+OctConv模型的图像级别混淆矩阵
Table 2. Image-level confusion matrix of ResNeXt+OctConv model
|
Normal类识别错误的图像如
3.4.3 ResNeXt+OctConv+HetConv模型
由于OctConv模块的特殊结构,用其替换原始的传统卷积,会大大增加训练参数的数量,使一个轮次的训练时间是ResNeXt模型的两倍左右。为了降低训练时间,引入HetConv模块,同时能够提高模型性能。该网络在ResNeXt+OctConv网络的基础上引入了HetConv(P=2)结构,替换初始层OctConv模块中的传统卷积层。该网络在验证集和训练集中的准确率如

图 12. ResNeXt+OctConv+HetConv模型的训练准确率和验证准确率
Fig. 12. Training accuracy and verification accuracy of the ResNeXt+OctConv+HetConv model
表 3. ResNeXt+OctConv+HetConv模型的图像级别混淆矩阵
Table 3. Image-level confusion matrix of ResNeXt +OctConv+HetConv model
|
从
表 4. 文献[ 4]的图像级别混淆矩阵
Table 4. Image level confusion matrix of Ref. [4]
|
召回率指在实际正样本中被预测为正样本的概率,可表示为
精准率指在所有被预测正样本中实际为正样本的概率,可表示为
准确率指预测正确的结果占总样本数的概率,可表示为
表 5. 两种方法的召回率、精准率和准确率
Table 5. Recall,precision and accuracy of two methods unit: %
| |||||||||||||||||||||||||||||||||
召回率可以反映被正确判定正例占总正例的比重,从
HetConv结构中的参数P经过实验得出,当P=4时,是一个3×3和三个1×1的卷积核组成的结构,模型的泛化能力较弱。因此,选择P=2的HetConv结构。
3.4.4 实验结果对比
为了验证本方法的有效性,用相同数据集对比不同模型的四分类任务识别率,结果如
表 6. 不同模型的识别率
Table 6. Recognition rate of different models unit: %
|
表 7. 不同方法得到的实验结果
Table 7. Experimental results obtained by different methods unit: %
|
4 结论
利用CNN对乳腺癌组织病理学图像进行自动分类,通过改进的深度CNN模型使网络具有更深的网络结构,在减少训练参数的同时提高分类准确率。在数据预处理部分采用对比度拉伸方法增加了图像中细胞核的可识别度,同时克服了因为数据量小导致的过拟合问题。实验结果表明,相比其他方法,本方法对四种类别的识别度均有提高,同时降低了提取特征的冗余情况,减少了冗余对识别的影响及计算消耗。此外,本方法对识别相近类别的细节特征更敏感,且具有较好的鲁棒性和实时性,在一定程度上可满足临床应用的需求。
[2] Spanhol FA, Oliveira LS, PetitjeanC, et al. Breast cancer histopathological image classification using convolutional neural networks[C]//2016 International Joint Conference on Neural Networks (IJCNN), July 24-29, 2016, Vancouver, BC, Canada. New York: IEEE, 2016: 2560- 2567.
[4] GolatkarA, AnandD, SethiA. Classification of breast cancer histology using deep learning[EB/OL]. [2020-03-23].https://arxiv.org/abs/1802. 08080.
[5] RakhlinA, ShvetsA, IglovikovV, et al. Deep convolutional neural networks for breast cancer histology image analysis[M] //Rakhlin A, Shvets A, Iglovikov V, et al. Image Analysis and Recognition. ICIAR 2018. Lecture Notes in Computer Science. Cham: Springer, 2018, 10882: 737- 744.
[6] KonéI, BoulmaneL. Hierarchical ResNeXt models for breast cancer histology image classification[M] //Campilho A, Karray F, ter Haar Romeny B. et al. Image Analysis and Recognition. ICIAR 2018. Lecture Notes in Computer Science. Cham: Springer, 2018, 10882: 796- 803.
[7] NazeriK, AminpourA, EbrahimiM. Two-stage convolutional neural network for breast cancer histology image classification[EB/OL]. [2020-03-21].https://arxiv.org/abs/1803. 04054.
[8] Wang Z H, You K Y, Xu J J, et al. Consensus design for continuous-time multi-agent systems with communication delay[J]. Journal of Systems Science and Complexity, 2014, 27(4): 701-711.
[10] Gu Y, Lu X Q, Yang L D, et al. Automatic lung nodule detection using a 3D deep convolutional neural network combined with a multi-scale prediction strategy in chest CTs[J]. Computers in Biology and Medicine, 2018, 103: 220-231.
[11] 孟婷, 刘宇航, 张凯昱. 一种基于增强卷积神经网络的病理图像诊断算法[J]. 激光与光电子学进展, 2019, 56(8): 081001.
[12] 李素梅, 雷国庆, 范如. 基于双通道卷积神经网络的深度图超分辨研究[J]. 光学学报, 2018, 38(10): 1010002.
[13] Xie SN, GirshickR, DollárP, et al. Aggregated residual transformations for deep neural networks[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), July 21-26, 2017, Honolulu, HI, USA. New York: IEEE, 2017: 5987- 5995.
[14] Chen YP, Fan HQ, XuB, et al. Drop an octave: reducing spatial redundancy in convolutional neural networks with octave convolution[C]//2019 IEEE/CVF International Conference on Computer Vision (ICCV), October 27-November 2, 2019, Seoul, Korea (South). New York: IEEE, 2019: 3434- 3443.
[15] SinghP, Verma VK, RaiP, et al. HetConv: heterogeneous kernel-based convolutions for deep CNNs[C]//2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR), June 15-20, 2019, Long Beach, CA, USA. New York: IEEE, 2019: 4830- 4839.
[16] IoffeS, SzegedyC. Batch normalization: accelerating deep network training by reducing internal covariate shift[EB/OL]. [2020-03-23].https://arxiv.org/abs/1502. 03167.
[18] He KM, Zhang XY, Ren SQ, et al. Deep residual learning for image recognition[C]//2016 IEEE Conference on Computer Vision and Pattern Recognition (CVPR), June 27-30, 2016, Las Vegas, NV, USA. New York: IEEE, 2016: 770- 778.
[19] LinM, ChenQ, Yan SC, et al. Network in network[EB/OL]. [2020-03-25].https://arxiv.org/abs/1312. 4400.
[20] Srivastava N. Improving neural networks with dropout[J]. University of Toronto, 2013, 53(9): 1689-1699.
[23] Aresta G, Araújo T, Kwok S, et al. BACH: grand challenge on breast cancer histology images[J]. Medical Image Analysis, 2019, 56: 122-139.
[24] Gu Y, Lu X Q, Zhang B H, et al. Automatic lung nodule detection using multi-scale dot nodule-enhancement filter and weighted support vector machines in chest computed tomography[J]. PLOS One, 2019, 14(1): e0210551.
[25] 郭琳琳, 李岳楠. 基于专家乘积系统的组织病理图像分类算法[J]. 激光与光电子学进展, 2018, 55(2): 021008.
[26] Khan S, Islam N, Jan Z, et al. A novel deep learning based framework for the detection and classification of breast cancer using transfer learning[J]. Pattern Recognition Letters, 2019, 125: 1-6.
[27] 谷宇, 吕晓琪, 吴凉, 等. 基于NSCT和CLAHE的乳腺钼靶X线图像微钙化点增强方法[J]. 光学技术, 2018, 44(1): 6-12.
[28] GuptaV, BhavsarA. Breast cancer histopathological image classification: is magnification important?[C]//2017 IEEE Conference on Computer Vision and Pattern Recognition Workshops (CVPRW), July 21-26, 2017, Honolulu, HI, USA. New York: IEEE, 2017: 769- 776.
[29] Bardou D, Zhang K, Ahmad S M. Classification of breast cancer based on histology images using convolutional neural networks[J]. IEEE Access, 2018, 6: 24680-24693.
[30] 谷宇, 吕晓琪, 赵瑛, 等. 基于PSO-SVM的乳腺肿瘤辅助诊断研究[J]. 计算机仿真, 2015, 32(5): 344-349.
Gu Y, Lü X Q, Zhao Y, et al. Research on computer-aided diagnosis of breast tumors based on PSO-SVM[J]. Computer Simulation, 2015, 32(5): 344-349.
[31] 何雪英, 韩忠义, 魏本征. 基于深度学习的乳腺癌病理图像自动分类[J]. 计算机工程与应用, 2018, 54(12): 121-125.
He X Y, Han Z Y, Wei B Z. Breast cancer histopathological image auto-classification using deep learning[J]. Computer Engineering and Applications, 2018, 54(12): 121-125.
牛学猛, 吕晓琪, 谷宇, 张宝华, 张明, 任国印, 李菁. 基于改进ResNeXt的乳腺癌组织病理学图像分类[J]. 激光与光电子学进展, 2020, 57(22): 221021. Xuemeng Niu, Xiaoqi Lü, Yu Gu, Baohua Zhang, Ming Zhang, Guoyin Ren, Jing Li. Breast Cancer Histopathological Image Classification Based on Improved ResNeXt[J]. Laser & Optoelectronics Progress, 2020, 57(22): 221021.