基于密集连接网络模型的致病菌拉曼光谱分类  下载: 636次
下载: 636次
1 引言
细菌感染严重危害人类身体健康,但目前临床上常用的检测手段需要进行细菌培养,操作繁琐且耗时较长,往往会耽误最佳治疗时机[1]。因此,开发一种快速准确的细菌识别方法对及时有效治疗细菌感染具有重要意义。
拉曼光谱可以提供细菌所含不同生物分子的指纹图谱信息,如蛋白质、DNA和脂质[2]。每种细菌都有其特定的指纹图谱信息,因此,可将拉曼光谱用于细菌种类鉴定[1]。但细菌的拉曼信号弱、易被噪声干扰且同一菌种不同亚型的拉曼光谱差异极小、难以区分[3]。细菌拉曼光谱分类中应用广泛的是传统机器学习方法[4],如:Kloβ等[5]用支持向量机(SVM)成功实现了11种会造成尿路感染细菌的分类;史如晋等[6]构建的集成学习模型实现了对大肠杆菌和布鲁氏菌的快速鉴定;Yan等[7]结合核主成分分析和决策树方法实现了23种食源性致病菌的分类。传统机器学习方法对细菌的分类效果主要取决于光谱预处理策略,且只能采用尝试法寻找最佳预处理策略,效率不高[8]。卷积神经网络(CNN)模型具有强大的特征自学习能力,可根据数据集自动提取最有利于任务的特征[9]。如:Liu等[10]将CNN模型用于矿物的拉曼光谱分类,结果表明,CNN模型的分类效果优于常见的传统机器学习算法;Huang等[11]使用一维卷积网络模型成功实现了20种动物血液拉曼光谱的分类;Ho等[12]设计出一个基于残差的一维卷积网络模型,实现了30种常见致病菌拉曼光谱的分类。
本文构建了一种基于密集连接的一维卷积网络模型Raman-net,无需额外的光谱预处理就能有效对细菌拉曼光谱进行分类。在Bacteria-ID数据集上的实验结果表明,Raman-net的分类准确率远高于传统机器学习方法且优于Ho等[12]提出的残差网络模型。此外,对细菌表面增强拉曼光谱Raman-net也能得到较好的分类效果。
2 算法原理
2.1 Raman-net模型
2.1.1 Raman-net原理
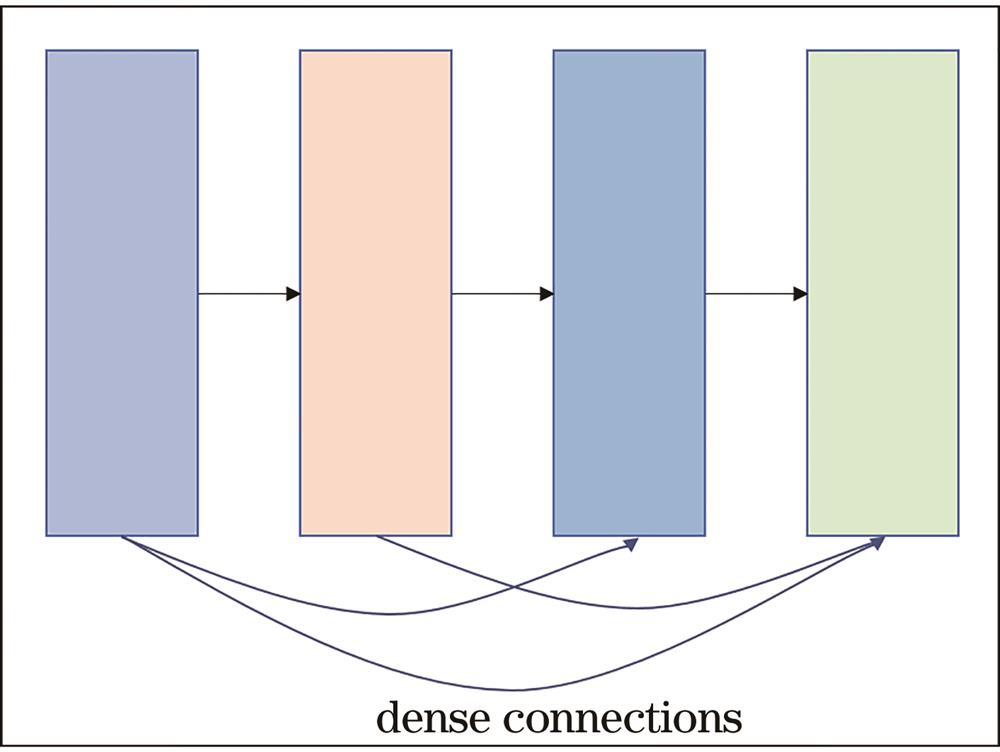
何凯明等[13]提出的残差网络(ResNet)在普通CNN模型中引入快捷连接(Shortcut connection),缓解了训练深层神经网络模型过程中出现的梯度消失和退化问题(Degradation problem),使深层的网络模型更易优化,从而通过增加网络模型的层数增强模型的表达能力,改善模型性能。黄高等[14]受ResNet的启发,在普通卷积网络模型中引入更密集的快捷连接,提出了密集连接网络(DenseNet)。该模型一经提出就在MNIST、CIFAR-10和CIFAR-100等图片数据集上取得了良好的分类性能。DenseNet密集连接块内每个卷积层的特征图都和前面所有层的特征图在通道维度上拼接在一起,作为下一网络层的输入,如
式中:
受DenseNet启发,Raman-net将密集连接引入一维CNN,提高了模型对拉曼光谱特征图的利用率以及模型的准确率和鲁棒性。密集连接块内每一层仅向后层传递少量特征图,使Raman-net的待学习参数量少于普通CNN,给模型的训练和部署带来便利。
2.1.2 Raman-net模型的结构
Raman-net是一个密集连接的一维CNN模型,由一个初始卷积层(Conv)、4个密集连接块以及完全连接层等组成。Raman-net密集连接块的主体结构如
使用预激活机制,即在模型所有卷积层之前依次设置批归一化(BN)层和线性整流单元(ReLU)激活函数。ReLU激活函数可表示为
BN的主要作用是加快模型收敛速度,同时具有一定的正则化效果。模型训练过程中,采用交叉熵作为损失函数,可表示为
式中:
2.2 对比算法
SVM是一种线性二分类模型,其目标是在样本线性可分情况下从样本空间中寻找一个最大间隔分离超平面完成数据二分类[15]。通过引入核函数将非线性可分的数据映射到高维空间中,进而实现对非线性可分数据的分类,再通过一对一或一对多策略实现SVM的多分类模型。
随机森林(RF)是一种优秀的并行集成学习方法,是许多决策树的集成。对集成模型而言,基学习器间的差异性越大,模型的效果就越好。由于引入了随机抽样和随机选取划分属性两个随机步骤,增大了RF模型中各决策树间的差异性,进而提升了模型的性能。RF模型不过度依赖特定样本和属性,具有较强的抗干扰能力和泛化能力[16]。
K近邻(KNN)算法的思想是先计算待分类样本到其余各样本的距离(一般采用Minkowsky距离或Euclidean距离),然后选出距离待分类样本最近的K个样本,最后统计K个样本的类别,将待分类样本划分为K个样本中数量最多的类别。KNN算法一般适用于样本容量较大的数据集,对于样本容量较小的数据集分类效果不佳[17]。
3 研究对象
3.1 普通拉曼光谱数据集
Ho等[12]公开的Bacteria-ID细菌拉曼光谱数据集包含30种常见致病菌的78500条拉曼光谱,光谱范围为381.98~1792.4 cm-1,共1000个波段,实验中使用了该数据集的reference、finetune和test子集。其中:reference子集包含了60000条拉曼光谱数据,30种致病菌各2000条;finetune和test子集各包括3000条拉曼光谱数据,30种致病菌各100条。所有拉曼光谱数据均经过归一化处理并去除基线。
3.2 表面增强拉曼光谱数据集
3.2.1 实验样本
细菌收集自徐州医科大学附属医院检验科,包含耐碳青霉烯类肺炎克雷伯菌(CRKP)和对碳青霉烯类敏感的肺炎克雷伯菌(CSKP)各10株。分离株在哥伦比亚血平板培养基上37 ℃ 过夜培养,然后用无菌去离子水制备浓度约为107 mL-1的细菌溶液。
3.2.2 实验器材及过程
根据Lee等[18]报道的柠檬酸三钠还原法制备纳米银颗粒(质量浓度为0.1 mg/mL,颗粒粒径为40~60 nm)作为增强基底,将10 μL菌液样品与10 μL银纳米颗粒均匀混合在EP管中,孵育10 min后取4 μL混合物滴在单晶硅片上作为测量样品。测量前以硅片在520.7 cm-1的峰为基准,对便携式拉曼光谱仪(B &W TEK,i-Raman Plus BWS465-785H)进行校准。仪器设置:用785 nm氦氖激光作为激发光源,激光功率为14 mW;20倍长聚焦物镜,积分时间为4 s,积分3次,测量范围为400~1800 cm-1。测量中每个菌株制备10份样品,每份样品随机选择6个位点进行检测,共获得2种肺炎克雷伯菌各600条表面增强拉曼光谱。含有碳青霉烯酶是CRKP具备耐药性的最主要原因[19]。
4 实验结果与分析
4.1 普通拉曼光谱数据集实验结果与分析
首先,用Raman-net在reference数据集上进行训练得到预训练模型。然后,将预训练模型在finetune数据集上进行微调整。最后,在test数据集上测试模型的泛化效果,得到Raman-net的分类准确率。反复尝试后设置的模型参数:1)用非线性函数ReLU作为激活函数;2)用dropout技术及早停策略降低模型的过拟合程度,drop rate为0.1;3)批处理大小为16;4)用Adam作为优化器,学习速率设置为0.001,betas设置为(0.5,0.999);5)采用5折交叉验证方式进行模型训练。
SVM、RF和KNN模型均使用finetune数据集作为训练集,test数据集作为测试集,使用网格搜索策略寻找最佳参数组合,采用5折交叉验证方式训练模型。为避免偶然性干扰,所有实验均重复5次,取5次分类结果的平均值作为最终分类准确率,结果如
表 1. 不同模型对普通拉曼光谱的分类准确率
Table 1. Classification accuracy of ordinary Raman spectra by different models
|
4.2 表面增强拉曼光谱数据集实验结果与分析
为研究数据集大小对各模型分类准确率的影响,依次从肺炎克雷伯菌表面增强拉曼光谱数据集中取每种细菌各100、200、300、400、500、600条光谱作为数据集,所有实验均将数据集随机划分80%作为训练集,20%作为测试集。无需任何光谱预处理流程,将Raman-net的预训练模型在训练集上进行训练,在测试集上测试模型的分类效果。SVM、RF和KNN模型均在训练集上训练,在测试集上得出分类准确率,使用网格搜索策略寻找最佳参数组合,采用5折交叉验证方式训练模型。为避免偶然性干扰,所有实验均重复5次,取5次分类结果的平均值作为最终分类准确率,结果如
表 2. 不同模型对表面增强拉曼光谱的分类准确率
Table 2. Classification accuracy of surface-enhanced Raman spectra by different models
|
5 结论
Raman-net无需光谱预处理就可有效分类细菌的拉曼光谱,对30种致病菌的拉曼光谱分类的准确率为84.26%,优于SVM(79.19%)、RF(63.80%)和KNN(36.63%)以及一维CNN模型。对于两种极其相似的肺炎克雷伯菌的表面增强拉曼光谱,Raman-net的识别准确率可达99.16%,显著高于传统机器学习方法。综上所述,Raman-net对细菌的普通拉曼光谱和表面增强拉曼光谱均具有优秀的分类性能且无需光谱预处理,为细菌的拉曼光谱分类提供了一种快速有效的手段。
[1] 杨凯, 李弘哲, 朱永官, 等. 基于拉曼光谱的快速细菌诊断[J]. 光散射学报, 2019, 31(4): 336-345.
Yang K, Li H Z, Zhu Y G, et al. Rapid bacterial diagnosis by Raman spectroscopy[J]. The Journal of Light Scattering, 2019, 31(4): 336-345.
[2] 依丽努尔·木合塔尔, 沈爱国, 胡继明. 细菌的拉曼光谱研究进展[J]. 光散射学报, 2015, 27(2): 110-118.
[3] 高鹏亚, 苏英会, 孙晖, 等. 显微共聚焦拉曼技术在细菌分类鉴定中的应用[J]. 疾病监测, 2021, 36(1): 74-81.
Gao P Y, Su Y H, Sun H, et al. Application of micro confocal Raman technique in classification and identification of bacteria[J]. Disease Surveillance, 2021, 36(1): 74-81.
[4] Lussier F, Thibault V, Charron B, et al. Deep learning and artificial intelligence methods for Raman and surface-enhanced Raman scattering[J]. TrAC Trends in Analytical Chemistry, 2020, 124: 115796.
[5] Kloβ S, Kampe B, Sachse S, et al. Culture independent Raman spectroscopic identification of urinary tract infection pathogens: a proof of principle study[J]. Analytical Chemistry, 2013, 85(20): 9610-9616.
[6] 史如晋, 夏钒曾, 曾万聃, 等. 基于PCA-Stacking模型的食源性致病菌拉曼光谱识别[J]. 激光与光电子学进展, 2019, 56(4): 043003.
[7] Yan S S, Wang S Y, Qiu J X, et al. Raman spectroscopy combined with machine learning for rapid detection of food-borne pathogens at the single-cell level[J]. Talanta, 2021, 226: 122195.
[8] Engel J, Gerretzen J, Szymańska E, et al. Breaking with trends in pre-processing?[J]. TrAC Trends in Analytical Chemistry, 2013, 50: 96-106.
[9] SzegedyC, LiuW, JiaY, et al. Going deeper with convolutions[C]∥2015 IEEE Conference on Computer Vision and Pattern Recognition, June 7-12, 2015, Boston, MA. New York: IEEE Press, 2015: 1-9.
[10] Liu J C, Osadchy M, Ashton L, et al. Deep convolutional neural networks for Raman spectrum recognition: a unified solution[J]. The Analyst, 2017, 142(21): 4067-4074.
[11] Huang S, Wang P, Tian Y B, et al. Blood species identification based on deep learning analysis of Raman spectra[J]. Biomedical Optics Express, 2019, 10(12): 6129-6144.
[12] Ho C S, Jean N, Hogan C A, et al. Rapid identification of pathogenic bacteria using Raman spectroscopy and deep learning[J]. Nature Communications, 2019, 10(1): 4927.
[13] HeK, ZhangX, RenS, et al. Deep residual learning for image recognition[C]∥2016 IEEE Conference on Computer Vision and Pattern Recognition, June 27-30, 2016, Las Vegas, NV, USA. New York: IEEE Press, 2016: 770-778.
[14] HuangG, LiuZ, Van der MaatenL, et al. Densely connected convolutional networks[C]∥2017 IEEE Conference on Computer Vision and Pattern Recognition, July 21-26, 2017, Honolulu, HI, USA. New York: IEEE Press, 2017: 2261-2269.
[15] Xu Y, Zomer S, Brereton R G. Support vector machines: a recent method for classification in chemometrics[J]. Critical Reviews in Analytical Chemistry, 2006, 36(3/4): 177-188.
[16] Gromski P S, Muhamadali H, Ellis D I, et al. A tutorial review: Metabolomics and partial least squares-discriminant analysis-a marriage of convenience or a shotgun wedding[J]. Analytica Chimica Acta, 2015, 879: 10-23.
[17] Morais C L M, Lima K M G, Singh M, et al. Tutorial: multivariate classification for vibrational spectroscopy in biological samples[J]. Nature Protocols, 2020, 15(7): 2143-2162.
[18] Lee P C, Meisel D. Adsorption and surface-enhanced Raman of dyes on silver and gold sols[J]. The Journal of Physical Chemistry, 1982, 86(17): 3391-3395.
[19] 崔娟, 王佳, 姚慧生, 等. 2006—2010年儿童肺炎支原体感染流行病学分析[J]. 中国实用儿科杂志, 2013, 28(6): 446-448.
Cui J, Wang J, Yao H S, et al. Epidemiologic analysis of MP infection in children from 2006 to 2010[J]. Chinese Journal of Practical Pediatrics, 2013, 28(6): 446-448.
[20] Maquelin K, Kirschner C, Choo-Smith L P, et al. Identification of medically relevant microorganisms by vibrational spectroscopy[J]. Journal of Microbiological Methods, 2002, 51(3): 255-271.
[21] Notingher I, Hench L L. Raman microspectroscopy: a noninvasive tool for studies of individual living cells in vitro[J]. Expert Review of Medical Devices, 2006, 3(2): 215-234.
[22] Fan C, Hu Z Q, Mustapha A, et al. Rapid detection of food- and waterborne bacteria using surface-enhanced Raman spectroscopy coupled with silver nanosubstrates[J]. Applied Microbiology and Biotechnology, 2011, 92(5): 1053-1061.
[23] Devitt G, Howard K, Mudher A, et al. Raman spectroscopy: an emerging tool in neurodegenerative disease research and diagnosis[J]. ACS Chemical Neuroscience, 2018, 9(3): 404-420.
Article Outline
杨勇, 董浩, 桑瑶烁, 李志刚, 张龙, 王玲, 王澍. 基于密集连接网络模型的致病菌拉曼光谱分类[J]. 激光与光电子学进展, 2023, 60(1): 0130003. Yong Yang, Hao Dong, Yaoshuo Sang, Zhigang Li, Long Zhang, Ling Wang, Shu Wang. Raman Spectral Classification of Pathogenic Bacteria Based on Dense Connection Network Model[J]. Laser & Optoelectronics Progress, 2023, 60(1): 0130003.