超分辨荧光显微成像的若干研究进展  下载: 646次特邀综述
下载: 646次特邀综述
1 引言
对生命本质的探索和生理活动的调控是人类的终极目标之一,而实现该目标的重要手段是在细胞和亚细胞水平观测生命活动的精细过程,目前这种观测主要依赖于可视化的显微技术。在各种显微技术中,荧光显微成像是应用最广泛的技术之一,它具有诸多优点:通过荧光发色团特异性标记可以观测特定的细胞成分及其相互作用[1],比如细胞核、细胞质、线粒体等;其次荧光显微成像可以观测活体样品的内部结构[2]及其动力学过程[3-5];此外,相比于X射线等放射性成像技术,荧光显微成像基本不会对生命体造成任何伤害。然而,相比于电子显微成像等技术,荧光显微成像在空间分辨率上有先天的不足,这主要是光学衍射极限导致的:即点光源经过光学系统到达探测器后,所成的像不是一个无限小的点,而是有一定大小的光斑(可通过点扩散函数来描述);当两个点光源相聚太近时,两个成像光斑就会相互叠加而无法分辨,这就使得荧光显微成像在空间分辨率上受到了明显的限制。德国物理学家Abbe[6]指出光学显微镜分辨率的极限遵循公式d=λ/2nsin α,其中,d为分辨率,λ为波长,n为折射率,α为孔径角。根据此公式可知,荧光显微镜的横向(即xy方向)分辨率在200~300 nm,纵向(即z方向)分辨率在500~700 nm。此空间分辨率与许多亚细胞结构相当,甚至比亚细胞结构要大得多,因此难以观测亚细胞结构的生命活动过程,更遑论观测蛋白质等结构(通常在10 nm尺度)的相互作用。
为了打破该限制,荧光超分辨显微成像技术应运而生,科学家通过不同的技术路径分别打破了光学衍射极限,实现了100 nm量级甚至10 nm以下的空间分辨率。2014年诺贝尔化学奖颁发给Eric Betzig、William E. Moerner、Stefan W. Hell等三位从事荧光超分辨显微成像的科学家,更是在全球范围内掀起了荧光超分辨研究的热潮。Moerner等[7-8]首先实现了单分子荧光的检测,为标记单个荧光蛋白开启了大门,随后Betzig等[9]使用不同颜色的激光照射荧光分子,使荧光分子处于激活的开态或者淬灭的关态,通过对不同分子分别进行成像,结合质心定位突破了光学衍射极限,这种技术被称为光活化定位显微成像,即PALM(Photoactived Localization Microscopy)。华人科学家庄小威教授通过使用荧光共振能量转移对获得荧光开关,用633 nm激光照射Cy5使其进入关态,再用532 nm激光照射Cy3,通过能量转移使 Cy5 进入开态,接着再用633 nm照射使其进入关态,多次往复,也实现了类似的超分辨成像,将空间分辨率提升了10倍,其基本原理如
![超分辨荧光显微技术的主要发展历史[10]](/richHtml/lop/2023/60/11/1106012/img_01.jpg)
图 1. 超分辨荧光显微技术的主要发展历史[10]
Fig. 1. Development of super-resolution photoluminescence imaging techniques[10]
上述几种成像方法虽然突破了光学衍射极限,获得了极高的空间分辨率,但也存在一定的缺陷。如:受激辐射损耗成像技术需要用较强的擦除光来照射样品,光漂白和光毒性较强,且样本需特殊荧光染料处理;受激辐射损耗成像技术使用点扫描技术,单分子定位显微成像通常需要采集上千帧,通过对图像进行重构以获得超分辨效果,二者均会消耗大量时间,因此牺牲了时间分辨率,难以实现细胞的动态超分辨成像。与其他超分辨率方法相比,SIM通常只能提供标准宽场显微镜大约两倍的分辨率增强,使用非线性饱和 SIM可以实现更高的空间分辨率(~50 nm),但由于显微成像中的背景噪声和结构光照明显微成像技术的照明光本身也受到衍射极限的限制,对显微系统分辨能力的提升也很有限,且需要增加图像采集次数和复杂的重建过程,依赖计算机算法处理,易导致图像出现伪影。近年来,科研工作者仍在不断努力,来克服这些缺陷,以期获得更佳的成像效果。本文介绍了若干种新的超分辨荧光显微成像方法,包括最低光子数显微成像、波动超分辨显微成像、基于单分子反聚束效应的超分辨成像以及基于深度学习的超分辨显微成像技术,其发展历程如
2 最低光子数荧光超分辨显微成像
2.1 最低光子数荧光超分辨显微成像的基本原理
理论上,受激辐射损耗显微成像和光激活定位显微成像(或随机光学重构显微成像)的空间分辨率可以达到单个荧光发色团的物理尺度(在1 nm量级);然而这两类成像方法或需要较长积分时间,或需要较高激发功率,在取得高空间分辨率的同时可能会严重损害细胞的活性。为了保障细胞活性,在实际应用中,只能采用低功率、短时间的方法限制光通量,以获得最佳的成像效率,综合考虑系统噪声和荧光探针特性的影响,这两类成像技术的空间分辨率通常被限制在 20 nm 左右。2017年,Hell教授团队结合这两类荧光超分辨显微技术的优势,提出了一种新的超分辨方案,被命名为最低光子数显微成像[16],即MINFLUX(Minimal Photon Fluxes),其基本原理如
![最低光子数显微成像技术的原理及装置示意图。(a)(b)常规纳米显微技术和最低光子数显微成像技术定位荧光分子中心的原理示意图[17];(c)实现最低光子数超分辨显微成像的实验装置示意图[18];(d)~(f)一维、二维、三维最低光子数显微成像技术的定位原理示意图[16,18];(g)相同光子数下,单分子定位显微成像与最低光子数显微成像分辨率的对比效果(比例尺:10 nm)[16]](/richHtml/lop/2023/60/11/1106012/img_02.jpg)
图 2. 最低光子数显微成像技术的原理及装置示意图。(a)(b)常规纳米显微技术和最低光子数显微成像技术定位荧光分子中心的原理示意图[17];(c)实现最低光子数超分辨显微成像的实验装置示意图[18];(d)~(f)一维、二维、三维最低光子数显微成像技术的定位原理示意图[16,18];(g)相同光子数下,单分子定位显微成像与最低光子数显微成像分辨率的对比效果(比例尺:10 nm)[16]
Fig. 2. Schematic diagrams of the principle and experimental setup of MINFLUX. (a) (b) Schematic diagrams of the principle to localize the center of fluorescent molecules for common nanoscopy and MINFLUX, respectively[17]; (c) schematic diagram of the setup of MINFLUX[18]; (d)-(f) principles of MINFLUX illustrated in one, two, and three dimensions, respectively[16,18]; (g) comparison of the spatial resolution between SMLM and MINFLUX under same photon counts (scale is 10 nm)[16]
最低光子数显微成像的实验方法一般如下:首先使用一束特定波长的激光激活发色团,使得在每次定位时,激光照射范围内只有一个荧光发色团处于开启状态。同时使用另一束光强分布为I(x,y)的激光来定位发色团,其特点是I(x,y)=0。最终结合超快速光学扫描设备和精密的可编程逻辑门阵列实现分子的精确定位,如
由于I0、f0、I1和f1均符合泊松分布,所以求解获得的位置xm也符合一定的概率分布,因此相对于真实值有一定偏差。数学上,该方法的定位精度可由克拉美-罗界公式(CRB)计算而得,即为
式中:σ为定位精度;N为采集到的光子数;x为荧光发射团位置。由
相比于常规的单分子定位显微成像技术,最低光子数显微成像所需要的荧光光子数减少了22倍,定位精度达到了1 nm,可分辨发色团的间距达6 nm。以单个荧光蛋白的最低光子数显微成像为例,其时间分辨率和单位时间跟踪的定位数量都增加了100倍。在相同荧光光子数的情况下,相比于受激发射损耗显微成像,最低光子数荧光超分辨显微成像的空间分辨率得到了显著的提高,如
2.2 最低光子数超分辨显微成像的优化与改进
2018年,Hell教授等在综合考虑信号背景比(SBR)和感兴趣区域(ROI)两个条件下,对最初的最低光子数显微成像技术作了进一步的改进与提升,从而确定了探测单个发色团位置坐标集的最佳空间范围L。通过优化最佳范围L,可以在1 ms的时间分辨率下使空间定位精度达到1.7 nm(当时间分辨率提升到0.1 ms时,空间定位精度仍可达到11 nm)。而在相同的时间尺度上,最初的最低光子数显微成像定位精度通常在40~60 nm,可见改进后的性能得到了大大提升。此外,从光子效率的角度考虑,改进后的最低光子数显微成像也得到了5~20倍的提升[19]。需要说明的是,以上实验主要是在二维尺度(即xy方向)上开展的,尽管如此,最低光子数显微成像在定位精度与成像速度上的优势已然得到了充分肯定,然而生物成像的最终目标是在三维尺度(即xyz方向)上实现超分辨显微。为此,2020年,德国马克斯-普朗克研究所的Balzarotti等在二维最低光子数显微成像的基础上作了两方面的改进:1)将二维成像中用于确定荧光发色团定位的4个成像位置提升到7个成像位置,如
![最低光子数显微成像在分辨率、三维多色成像方面的优化与改进。(a)用AF647和CF680染色后的U-2 OS细胞的双色三维最低光子数显微成像(比例尺:500 nm)[19];(b)双色最低光子数显微成像技术在三维空间的定位精度[20];(c)脉冲交错式最低光子数显微成像的实验装置图[21];(d)(e)基于脉冲交错式最低光子数显微成像技术获得的光子数分布及其寿命分布[21];(f)(g)基于光栅扫描单分子定位的测量方法以及该方法在不同扫描距离下的成像模拟结果[23]](/richHtml/lop/2023/60/11/1106012/img_03.jpg)
图 3. 最低光子数显微成像在分辨率、三维多色成像方面的优化与改进。(a)用AF647和CF680染色后的U-2 OS细胞的双色三维最低光子数显微成像(比例尺:500 nm)[19];(b)双色最低光子数显微成像技术在三维空间的定位精度[20];(c)脉冲交错式最低光子数显微成像的实验装置图[21];(d)(e)基于脉冲交错式最低光子数显微成像技术获得的光子数分布及其寿命分布[21];(f)(g)基于光栅扫描单分子定位的测量方法以及该方法在不同扫描距离下的成像模拟结果[23]
Fig. 3. Development in terms of resolution and 3D multicolor imaging of MINFLUX. (a) Two-color MINFLUX nanoscopy in 3D of U-2 OS cell with AF647 and CF680(scale: 500 nm)[19]; (b) three-dimensional localization accuracy of MINFLUX[20]; (c) schematic of the p-MINFLUX setup[21]; (d)(e) 2D localization image and fluorescence lifetime image for one DNA origami structure through p-MINFLUX[21]; (f)(g) measurement method based on grating scanning single molecule localization and imaging simulation results of this method at different scanning distances[23]
在实验系统方面,可以想象,实现如此高定位精度和时间分辨率的方法,必然需要较为复杂的仪器相互配合,比如超快速光学扫描设备和精密的可编程逻辑门阵列电路,如
在与常规显微系统结合方面,Hell教授团队通过同步电光光束偏转器与扫描振镜,结合样品位置锁定装置,即可在标准的荧光显微系统上实现了纳米分辨率,极大地拓展了最低光子数显微成像的应用范围[22]。在该系统中,既可以选择高斯光束,使用常规的共聚焦扫描模式激发样品,实现荧光显微成像,也可以选择环状甜甜圈光束,使用最低光子数显微成像方案,实现超分辨荧光显微成像。此外,还可以采用可变形镜改变聚焦光的散焦效果,使焦点零强度点沿z轴移动,通过层析的方式获得三维超分辨荧光显微成像。在该方案中,样品位置的稳定是极其重要的。为此,一方面可以将荧光发色团与反射基准(比如金纳米棒)绑定在一起,利用反射基准所发射的背向散射信号结合反馈系统,来维持生物样品的稳定性。另一方面,则可以预先采集轴向设定点上方和下方的图片作参考,将相机实时采集的图片与参考图片进行交叉关联,使用多基准组合信号的图像关联分析,实现更精准的位置锁定。基于该系统可以对细胞等样品进行最低光子数显微成像,相关结果表明:对于二维成像,当检测光子数达到800时,单个发色团的定位精度可达到2.2 nm;当光子数增加到2500时,定位精度可小于1 nm;在0.1 ms的积分时间下可以实现小于20 nm的定位精度。对于三维成像,当光子数为1200时,横向定位精度(焦平面定位精度)可达2.4 nm,沿光轴的定位精度可达1.9 nm。此外,Masullo等于[23-24]近期提出了一种基于光栅扫描的单分子定位方法,即RASTMIN(single-molecule localization by RASTer scanning a MINimum of light),如
2.3 最低光子数显微成像技术与其他技术的结合
最低光子数显微成像拥有优异的定位精度和时间分辨能力,将该技术与其他超分辨技术相结合,有望获得更加优越的性能。具体而言:最低光子数显微成像技术的定位精度可以达到1 nm量级,已经与单个发色团的尺寸相当;然而定位精度不等于光学分辨率,定位精度为多次测量后的误差,而分辨率为能够分辨的最小尺寸,该技术的光学分辨率仍有很大的提升空间;此外,该方法在时间分辨率与成像信背比方面还有很大的挖掘空间。
多光子显微成像的重要特征是荧光信号与激发强度呈非线性关系,其空间分辨率可通过非线性过程来提升,因此利用多光子激发,有望进一步提高最低光子数显微成像的空间分辨率。北京大学的席鹏教授团队首先发展了双光子最低光子数显微成像技术[25],即2p-MINFLUX(Two Photon MINFLUX),并从理论上证明通过设计和利用点扩散函数的特性,可以使其定位精度和空间分辨率提高1倍,获得小于1 nm的定位精度,结果如
![最低光子数显微成像与其他显微成像技术相结合的成像效果。(a)(b)通过模拟获得的最低光子数显微成像的z轴定位效果,其中图a为单光子激发,图b为双光子激发,(比例尺:1 nm)[25];(c)图像扫描最低光子数显微成像的原理示意图,利用阵列探测器代替点探测器来扩大成像视野[27];(d)(e)单分子定位显微技术与结构光照明最低光子数显微技术对相同发色团进行成像的效果比较(比例尺:50 nm)[28];(f)共聚焦显微技术、(g)受激辐射损耗显微技术以及(h)最小光子数受激辐射损耗技术对相似线粒体进行成像的效果对比(比例尺:200 nm)[29];(i)利用Vimentin-reEGFP2荧光蛋白实现的共聚焦荧光成像(左图)与利用DNA-PAINT技术获得的最低光子数显微成像效果的对比(比例尺:200 nm)[31]](/richHtml/lop/2023/60/11/1106012/img_04.jpg)
图 4. 最低光子数显微成像与其他显微成像技术相结合的成像效果。(a)(b)通过模拟获得的最低光子数显微成像的z轴定位效果,其中图a为单光子激发,图b为双光子激发,(比例尺:1 nm)[25];(c)图像扫描最低光子数显微成像的原理示意图,利用阵列探测器代替点探测器来扩大成像视野[27];(d)(e)单分子定位显微技术与结构光照明最低光子数显微技术对相同发色团进行成像的效果比较(比例尺:50 nm)[28];(f)共聚焦显微技术、(g)受激辐射损耗显微技术以及(h)最小光子数受激辐射损耗技术对相似线粒体进行成像的效果对比(比例尺:200 nm)[29];(i)利用Vimentin-reEGFP2荧光蛋白实现的共聚焦荧光成像(左图)与利用DNA-PAINT技术获得的最低光子数显微成像效果的对比(比例尺:200 nm)[31]
Fig. 4. Super-resolution imaging by combining the MINFLUX with other techniques. Simulation of z-axis localization for (a) single-photon MINFLUX and (b) two-photon MINFLUX, respectively(scale: 1 nm)[25];(c) concept of ISM-FLUX, an activation laser beam activates a single fluorophore in the sample (yellow star) which is sequentially excited by a series of spatially displaced doughnut beams[27]; (d) SMLM and (e) SIMFLUX image for the same nano-rulers(scale: 50 nm)[28]; (f) comparison of imaging effects of confocal microscopy, (g) stimulated radiation loss microscopy, and (h) minimum photon number stimulated radiation loss technique on similar mitochondria (scale: 200 nm)[29]; (i) comparison of confocal fluorescence imaging using Vimentin-reEGFP2 fluorescent protein (left image) and minimum photon number microscopy imaging using DNA-PAINT technology (scale: 200 nm)[31]
尽管最低光子数显微成像在定位精度方面具有突出优势,但该技术使用单点扫描技术结合单点探测器进行探测,视场一般为百纳米量级,通过将所有光子与单点探测器进行空间积分,一些关于分子位置的信息就会丢失了,当分子在甜甜圈状激光所在区域外时情况尤为严重。为了实现对单个发色团的高精度定位,通常需要预定位步骤来大致确定分子的位置。为了突破这一限制,2022年,意大利理工学院的Slenders与Vicidomini结合最低光子数显微成像与图像扫描显微成像[26]两种技术优势,发展了基于图像扫描的最低光子数显微成像,即ISM-FLUX(Image Scanning Microscopy MINFLUX)[27],其实验原理如
2021年德国马克斯-普朗克研究所的Weber等[29]则将最低光子数原理与激辐射损耗荧光显微技术相结合,构建了最低光子数受激辐射损耗荧光显微成像,即MINSTED,能够在更少光子数的情况下(相应地,对光漂白事件需求也减少)将空间分辨率从衍射极限提升到荧光发色团本身尺度。
如上所述,最低光子数显微成像依赖于可切换或可激活的荧光发色团,为了实现多色荧光成像,就要求不同发色团的亮度和开关行为在一个狭窄的范围内进行适当的匹配;而且多色成像还需要使用特性的缓冲溶液来实现发色团的开关切换,这显然对多色显微成像提出了极大的挑战。2017年哈佛大学Schnitzbauer等[30]提出通过DNA折纸的点累积成像技术(DNA-PAINT)来实现荧光切换。若将DNA双链中的一条与目标分子相连作为对接链,而互补的另一条与荧光发色团相连作为成像链,在显微成像系统的焦平面,成像链和对接链会自发结合和脱离,实现开关状态切换。通过设计DNA双链,可以控制目标分子与荧光发色团的结合时间,从而控制状态切换速率,而不需要专门的缓冲液,其成像效果如
通过以上综述可以发现,将最低光子数显微成像技术,与结构光照明、受激辐射损耗、图像扫描等技术结合后,增大了成像视场,简化了实验装置,提高了信背比,在保证发色团定位精度、空间分辨率和时间分辨率的同时,可以获取荧光发色团的寿命、光谱、偏振等信息,为研究生物大分子之间的相互作用及其动力学过程供了强有力的工具。但这些技术仍有一定的缺陷,无法同时在视场、所需光子数、信背比、系统简易性及分辨能力等方面均达到最佳,他们的优缺点如
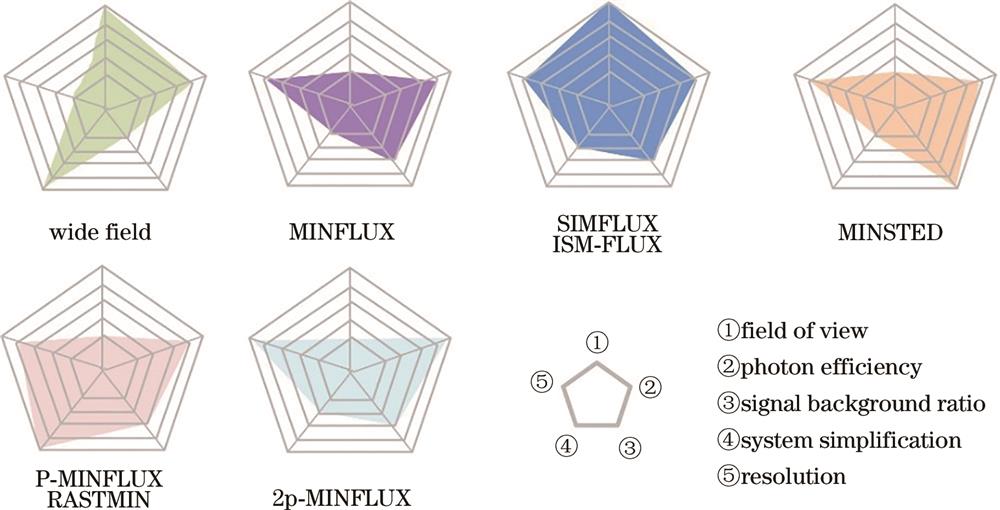
图 5. 宽场成像、最低光子数显微成像技术及其与其他超分辨方法结合技术在视场、光子效率、信背比、系统简易性及分辨能力等方面的对比
Fig. 5. Comparison of wide field imaging, minimum photon number microscopic imaging technology, and their combination with other super-resolution methods in field of view, photon efficiency, signal to back ratio, system simplicity, and resolution ability
3 波动超分辨荧光显微成像
常规宽场荧光显微成像通常使用像素较多的高灵敏相机进行拍照(比如像素为512×512的电子倍增电感耦合器件,即EMCCD),这些像素不仅给出了发色团荧光强度的空间分布,而且记录了发色团的荧光闪烁等特性,蕴含着丰富的光学信息。2009年,加州大学Dertinger教授[32]小组基于每个像素所记录的光子序列的波动性(即由荧光发色团间歇性闪烁导致的光子数起伏)以及相邻像素的关联性,提出了光学波动超分辨荧光显微成像技术,即SOFI(Super-resolution Optical Fluctuation Imaging)。基于该技术可以在三个维度上获得突破光学衍射极限的空间分辨率。与单分子定位超分辨显微成像等技术相比,波动超分辨显微成像只需要满足三个条件:1)荧光发色团保持至少两种不同的发射状态(比如开态和关态);2)不同发色团的不同发射状态可以随机、独立、反复的切换;3)成像系统的像素尺寸小于光学系统的衍射极限。当满足以上条件时,即可对原始像素的时间序列进行自相关计算,获得n阶累积量,实现超分辨荧光显微成像,而不需要构建复杂的成像系统。此外,波动超分辨荧光显微成像不需要进行长时间采样,有效降低了光漂白的概率,非常适用于生物成像。在本小节中我们将简要介绍波动超分辨荧光显微成像的基本原理和最新研究进展。
3.1 波动超分辨显微成像的基本原理
在由N个单一且独立发射的荧光发色团所组成的样品中(
式中:εk为荧光发色团的开态亮度;sk(t)表征了荧光强度随时间的波动特性。在位置r和时间t处的荧光信号F(r,t),可通过系统的点扩散函数h(r)与发色团荧光分布的卷积得出:
假设发色团在采集过程中未发生位置漂移,荧光的波动性可表示为
式中:
为了减小光学系统的点扩散函数,生成高阶的波动超分辨显微图像,需要将n阶相关函数转换为n阶累积函数Cn,即
式中:wk为点扩散函数的半峰全宽。用上述算法对图像进行处理后,得到的点扩散函数在xy和z方向上缩小了N1/2倍,因此将图像的空间分辨率提高了N1/2倍,如
![波动超分辨荧光成像的原理示意图[32]。(a)像平面中发射器的空间分布;(b)图(a)中虚线框的放大细节;(c)每个像素的时间轨迹;(d)根据信号波动计算的每个像素的二阶相关函数;(e)每个像素二阶相关函数积分结果](/richHtml/lop/2023/60/11/1106012/img_06.jpg)
图 6. 波动超分辨荧光成像的原理示意图[32]。(a)像平面中发射器的空间分布;(b)图(a)中虚线框的放大细节;(c)每个像素的时间轨迹;(d)根据信号波动计算的每个像素的二阶相关函数;(e)每个像素二阶相关函数积分结果
Fig. 6. Principle of SOFI[32]. (a) Emitter distribution in the object plane; (b) magnified detail of the dotted box in Fig. (a); (c) time trajectory of each pixel; (d) second-order correlation function is calculated from the fluctuations for each pixel; (e) result of integrating the second-order correlation function per pixel
在以上基础上,2010年,Dertinger等[33-34]进一步优化了波动超分辨显微成像的算法,通过使用时空互积累量的方法消除了波动超分辨显微成像对相机像素尺寸的限制,即不再要求像素尺寸小于系统的光学衍射极限。结合使用重新加权光传递函数,可以使空间分辨率的增强与累积量阶数呈线性关系,这意味着n阶累积量即可实现n倍的分辨率增强。
具有荧光闪烁的标记物是实现波动成像的基本要求,常见的标记物有量子点、可切换染料、荧光蛋白及DNA-PAINT标记技术。量子点凭借其高荧光量子产率、较强的抗光损伤能力(与荧光染料相比)和高吸收横截面等优势在生物显微成像中得以广泛使用[35]。此外,也可以通过改变其尺寸、形状和组成成分来调整量子点的光谱,结合量子点的宽带吸收特性,使它们在多色成像中具有很高的吸引力[36]。北京大学席鹏团队通过用联合标记的方法[Joint-Tagging,如
![不同荧光标记物的波动超分辨成像图。(a)联合标记波动超分辨显微成像示意图[37];(b)超分辨波动光学成像的横截面示意图[37];(c)多色波动超分辨显微成像的光谱交叉累积分析,以及线性分解模拟的工作流程[38];(d)(e)用Lyn-Dronpa(绿色)和Kras-rs TagRFP(红色)标记的HeLa细胞共聚焦荧光成像和光致变色波动超分辨成像图[41](比例尺:10 μm)](/richHtml/lop/2023/60/11/1106012/img_07.jpg)
图 7. 不同荧光标记物的波动超分辨成像图。(a)联合标记波动超分辨显微成像示意图[37];(b)超分辨波动光学成像的横截面示意图[37];(c)多色波动超分辨显微成像的光谱交叉累积分析,以及线性分解模拟的工作流程[38];(d)(e)用Lyn-Dronpa(绿色)和Kras-rs TagRFP(红色)标记的HeLa细胞共聚焦荧光成像和光致变色波动超分辨成像图[41](比例尺:10 μm)
Fig. 7. SOFI images with different fluorescent labels. (a) Schematic diagram of joint labeled wave super-resolution microscopy imaging[37]; (b) schematic cross-section of the joint-tagging SOFI imaging[37]; (c) workflow of multicolor SOFI imaging by spectral cross-cumulant analysis followed by linear unmixing using simulations[38]; (d) conventional and (e) pcSOFI image of HeLa cell labeled with Lyn-Dronpa (green) and Kras-rsTagRFP (red), respectively[41] (scale: 10 μm)
3.2 波动超分辨显微成像的研究进展
尽管从波动超分辨的理论角度出发,通过更高阶的累积量,可以重构出具有更高空间分辨率的超分辨图像;然而在实验中,累积量阶数的增加会使超分辨图像产生伪影,进而使得图像失真,因此实际的空间分辨率提升效果受到了一定的限制。一种解决方案是使用局部动态范围压缩,即以局部的方式将高阶波动超分辨图像像素强度的动态范围,压缩到低分辨率参考图像的动态范围(例如,压缩到2阶波动超分辨图像的动态范围)[44]。为此,可以定义一个小的移动窗口,并将窗口内的像素强度重新调整到参考图像中相同区域的强度范围;窗口在视场上移动,由所有重新缩放的窗口平均值形成具有压缩动态范围的输出图像,具体计算流程如
![解决高阶波动超分辨成像伪影的算法流程图。(a)局部动态范围压缩算法流程图[44];(b)平衡波动超分辨荧光成像算法流程图[45]](/richHtml/lop/2023/60/11/1106012/img_08.jpg)
图 8. 解决高阶波动超分辨成像伪影的算法流程图。(a)局部动态范围压缩算法流程图[44];(b)平衡波动超分辨荧光成像算法流程图[45]
Fig. 8. Flowchart of algorithm to solve artifacts in the high-order SOFI. (a) Flowchart of local dynamic range compression algorithm[44]; (b) flowchart to illustrate the different steps of the bSOFI algorithm[45]
低阶自相关累积量在任何编程语言中都很容易实现,然而高阶累积量,特别是交叉累积量的计算,大大增加了计算中所包含的像素组合和时间延迟组合的数量,为波动超分辨成像的重建带来了一定的困难,因此需要使用高效的专用库编写特定的代码。为此,2012年,鲁汶大学的Dedecker教授和同事创建了超分辨率显微图像分析包——定位器,其框架和流程如
![波动超分辨成像数据分析的流程图以及波动超分辨成像技术与其他成像技术的对比。(a)超分辨定位器或者超分辨波动显微成像数据分析的一般流程[46];(b)单个发色团的空间分布以及这些发色团的宽场成像、平衡超分辨波动显微成像和随机光学重构超分辨成像的比较[48];(c)商业量子点QD625的共聚焦荧光显微成像、图像扫描显微成像,以及2阶和4阶超分辨光学波动图像扫描显微成像的对比[51]](/richHtml/lop/2023/60/11/1106012/img_09.jpg)
图 9. 波动超分辨成像数据分析的流程图以及波动超分辨成像技术与其他成像技术的对比。(a)超分辨定位器或者超分辨波动显微成像数据分析的一般流程[46];(b)单个发色团的空间分布以及这些发色团的宽场成像、平衡超分辨波动显微成像和随机光学重构超分辨成像的比较[48];(c)商业量子点QD625的共聚焦荧光显微成像、图像扫描显微成像,以及2阶和4阶超分辨光学波动图像扫描显微成像的对比[51]
Fig. 9. Flowchart of the analysis of SOFI data and the contrast between SOFI and other imaging techniques. (a) Schematic overview of a super-resolution localization or SOFI analysis[46]; (b) comparison of the spatial distribution of individual chromophores and their wide field imaging, balanced super-resolution wave microscopy imaging, and random optical reconstruction super-resolution imaging[48]; (c) comparison of confocal fluorescence microscopy, image scanning microscopy, and second-order and fourth-order super-resolution optical wave image scanning microscopy for commercial quantum dot QD625[51]
虽然波动超分辨显微成像具有易使用、低背景和高信噪比等优势,但由于其主要使用宽场显微技术,因此难以消除离焦平面信号的影响,导致最终z轴切片效果差且信噪比低,难以对厚度超过几微米的样品进行成像。将波动超分辨显微成像与其他显微成像技术相结合,恰恰可以克服该缺点。比如将波动超分辨显微成像与全内反射显微成像、共聚焦显微成像、光片荧光显微成像结合,可提供光学切片,实现对厚样品的超分辨成像。比如,与全内反射显微成像相结合时,有限深度的倏波照明提供了光学切片,同时降低了光毒性[49]。然而,这种方法只能对倏逝波界面很薄的细胞样品(百纳米量级)进行成像,另一种适用于厚样品的光学切片的方法是光片荧光显微成像。2020年,冷泉港实验室的Mizrachi教授团队[50]结合了切片荧光显微镜和超分辨光学波动成像,在原生三维组织的大视场上实现了快速纳米级的超分辨成像;并且通过这种方法获得了小鼠大脑厚切片的超分辨图像。尽管光片荧光显微镜成像能够在样品的任何深度进行成像,但该技术会受散射的限制。为了通过光学切片方法,获取厚样品良好的成像效果,还可以将变形图像扫描显微技术(即ISM)与波动超分辨显微成像技术相结合,形成超分辨光学波动图像扫描显微成像,即SOFISM(Super-resolution Optical Fluctuation Image Scanning Microscopy)[51],此时共焦针孔被包含几十个像素的探测器阵列所替换,相比图像扫描显微成像技术,该技术的分辨率得到了4倍的提升,同时z切片成像效果也得到了改善,如
相比于其他显微技术,波动超分辨显微成像可以将图像扫描显微成像和结构光照明显微成像的横向分辨率提高2倍,与非线性结构光照明显微技术[52]的空间分辨率相当。相比于受激发射损耗显微成像和单分子定位显微成像等技术,虽然波动超分辨显微成像未能达到其优越的分辨能力,但在时间分辨率上有一定的优势,更适用于动态过程和实时样本成像。相比于结构光照明成像和受激辐射损耗显微成像,波动超分辨显微成像装置更加简单。除了分辨率的提升,波动超分辨成像还可以作为荧光生物传感器,通过将荧光团附着在两个分子上来检测两个分子之间距离的变化,如键的结合或裂解[53]。
4 反聚束超分辨荧光显微成像
传统的光学显微成像技术会受到经典光场自身性质的限制:经典光场中光量子随机涨落产生的散粒噪声,会限制成像的灵敏度、分辨率和成像速率。量子光学理论和技术的快速发展已经被证明可以显著提高干涉测量的分辨率,而且具有增强成像灵敏度的效果,因此也为超分辨率显微成像提供了一些新的手段[54-55]。一种方案是直接将激发光由传统激光光源转换为量子光源(例如纠缠光)。当使用一束具有动量纠缠特性的光束探测物体时,通过符合计数测量就可以获得高分辨率的图像[56-57]。此方案利用了照明光的量子特性,需要非经典光源或者可产生非经典光的量子发射器。然而,非经典光源的产生需要通过非线性过程产生,光学系统相对复杂,且产生光子速率低,目前还难以用于显微成像。另一方面,由于目前缺乏高效的量子发射器,尚未有利用荧光标记物产生纠缠光实现超分辨成像的报道;但随着量子光学的快速发展,有望在不久的将来实现基于纠缠光的超分辨显微成像。另一种较为成熟的方法是:将待成像的细胞或样品,使用具有高效多光子吸收的荧光发色团进行染色标记,通过多光子吸收及发射实现超分辨显微,其原理类似于拥有超分辨率效果的量子光刻技术,通过获取高频量子干涉图案,克服衍射极限以实现成像的超分辨效果[58]。然而,常规标记物多光子吸收效率低,需要极高的光子通量,限制了其使用场景。
从另一个角度,即量子光学的本质-光场分布和光子数统计性质的角度来看,当光场呈现亚泊松分布,光子数的量子涨落将被压缩,此时可以降低散粒噪声,进而突破衍射极限,实现超分辨显微成像[59-60]。该方案在实验上具有可操作性,这主要是因为单个荧光发色团的荧光光子是逐个、分离的辐射出来的,而不是集中“爆炸式”的辐射,因此单个荧光发色团的辐射过程普遍具有亚泊松分布。具体的实现方法是在探测荧光光子时进行HBT(Hanbury Brown-Twiss)检测,即通过分束器将荧光光子均分为2束,并通过两个独立的单光子探测器分别探测这两路荧光光子的时间序列,并进行关联计算,如
![反聚束超分辨荧光成像的基本原理。(a)HBT测量的原理示意图[61];(b)连续光激发下单个荧光发色团发射光子的反聚束现象[62];(c)~(e)同一区域量子点的一阶、二阶和三阶反聚束荧光图像[64]](/richHtml/lop/2023/60/11/1106012/img_10.jpg)
图 10. 反聚束超分辨荧光成像的基本原理。(a)HBT测量的原理示意图[61];(b)连续光激发下单个荧光发色团发射光子的反聚束现象[62];(c)~(e)同一区域量子点的一阶、二阶和三阶反聚束荧光图像[64]
Fig. 10. Principle of super-resolution image based on anti-bunching effect. (a) Schematic diagram of the HBT measurement[61]; (b) anti-bunching signal of single molecule excited by continuous laser[62]; (c)-(e) first order, second order, and third order anti-bunching images of quantum dots in the same region[64]
式中:
2012年,以色列魏茨曼科学研究所Schwartz等[63]首次提出,通过检测单个激发脉冲后的荧光光子数分布A(x),可以在任意数量的激发周期内累积,进而实现超分辨成像。其中A(x)表示为
式中:Q(x)为Mandel Q参数。可见,A(x)对应的有效点扩散函数可表示为
式中:h(x)为有效点扩散函数。通过简单的数学转换可知,在傅里叶域中,hA跨越的空间频率间隔是h的2倍。换言之,反聚束显微成像的空间分辨率是经典成像的两倍,类似于双光子显微成像可实现分辨率2倍的提高(相对于其所用激光波长的分辨率)。反聚束信号由独立探测器光信号的自相关,或一对探测器中检测到的光子数N(x1)和N(x2)之间的互相关来确定。自相关所获得的2阶反聚束结果作为量化双光子符合事件的标准,其数越低则双光子事件越少。同样地,n阶反聚束结果量化了n光子符合事件,n阶反聚束信号对应的点扩散函数可表示为
与2阶类似,n阶反聚束可使三维成像的空间分辨率比经典光衍射极限下的成像要好n倍。基于该方案,该团队于2013年利用单量子点产生的具有反聚束特性的光子流,结合电子倍增相机探测进行成像,通过计算相邻像素间的光子关联特性,计算了2阶和3阶强度关联,在标准宽场荧光显微镜下实现了超越衍射极限的量子成像[64],如
通过以上分析,我们可以发现反聚束超分辨荧光显微技术与荧光波动超分辨显微成像技术关系紧密。两者的相似之处是均在荧光显微镜的成像平面上检测光子,而主要区别是信号波动的来源:荧光波动超分辨成像技术分析发色团亮度波动所导致的超泊松光子统计,而反聚束超分辨显微技术分析的是量子发射器的荧光反聚束效应所导致的亚泊松统计。由此可以看出,荧光波动超分辨成像技术信号高度依赖于荧光发色团的辐射波动,而反聚束成像信号的统计特性来源于量子发射器的稳定辐射。
原则上具有单光子发射特性的荧光发色团都适用于反聚束荧光超分辨成像,比如金刚石NV色心。2013年,中国科学技术大学郭光灿团队提出了一种基于反聚束光子发射的量子测量方法[65],在共聚焦系统中计算单光子和双光子信号,从中获得NV色心的荧光强度,实现了相距仅为(8.5±2.4)nm的两个NV色心的区分,如
![反聚束超分辨荧光成像的效果。(a)在共聚焦系统中测量2阶反聚束实现超分辨成像的装置示意图[65];(b)基于图(a)系统采集获得的NV色心的单双光子信号重建图像[65];(c)用单光子光纤束相机测量高阶反聚束实现超分辨成像装置示意图[67];(d)图(c)系统测量单个量子点的二维实验定位精度(实线)及理论预测精度(虚线)[67];(e)基于结构光照明成像与纠缠显微镜实现结构光照明的量子关联显微成像的原理示意图[68];(f)结构光照明的量子关联的显微成像结果,其中CM2指2阶关联显微成像[68];(g)(h)在共聚焦显微系统和量子图像扫描显微系统中QD625量子点标记的微管细胞样本成像结果[69]](/richHtml/lop/2023/60/11/1106012/img_11.jpg)
图 11. 反聚束超分辨荧光成像的效果。(a)在共聚焦系统中测量2阶反聚束实现超分辨成像的装置示意图[65];(b)基于图(a)系统采集获得的NV色心的单双光子信号重建图像[65];(c)用单光子光纤束相机测量高阶反聚束实现超分辨成像装置示意图[67];(d)图(c)系统测量单个量子点的二维实验定位精度(实线)及理论预测精度(虚线)[67];(e)基于结构光照明成像与纠缠显微镜实现结构光照明的量子关联显微成像的原理示意图[68];(f)结构光照明的量子关联的显微成像结果,其中CM2指2阶关联显微成像[68];(g)(h)在共聚焦显微系统和量子图像扫描显微系统中QD625量子点标记的微管细胞样本成像结果[69]
Fig. 11. Imaging results of super-resolution image based on anti-bunching effect. (a) Schematic diagram of a super-resolution imaging device for measuring second-order anti-bunching in a confocal system[65]; (b) reconstructed image according to the single and double photon signals of NVs collected from the system in a[65]; (c) schematic diagram of a super-resolution imaging device for measuring high-order anti-bunching with a single-photon fiber beam camera[67]; (d) two-dimensional localization accuracy measured for a single QD (solid blue) and theoretical accuracy by using the system in c[67]; (e) schematic of SIQCM by combining SIM and quantum correlation microscopy[68]; (f) simulation results illustrated the resolution of SIQCM[68]; (g) (h) imaging results of QD625-labeled microtubule cell samples in confocal microscopy and Q-ISM[69]
反聚束超分辨技术也可以和其他超分辨技术进行结合,以进一步提高超分辨成像的性能。2017年,德国埃尔朗根-纽伦堡大学的Classen等[68]将反聚束超分辨显微技术与结构光照明显微成像技术相结合后,在m阶自关联条件下,可以实现分辨率m+m1/2倍的提升,他们称该技术为结构光照明的量子关联显微成像,即SIQCM(Structured Illumination Quantum Correlation Microscopy),成像原理与结果如
除了荧光的反聚束效应可用于实现超分辨成像外,合理利用光的其他量子特性也使发展新的超分辨率荧光成像技术成为可能。2022年中国科学院上海高等研究院的王中阳课题组[70]从荧光分子的发光机制出发,获取并分析了荧光染料通用的单光子波函数及其在显微系统中随时间和空间的变化行为,计算了双光子干涉的探测概率,构建了荧光量子相干统计模型。在此模型基础上,提出了一种基于荧光量子相干特性的超分辨荧光显微成像方法,利用具有时间相关单光子计数技术的单光子雪崩探测器阵列进行荧光相干性的时空光子统计,从确定的相干性可以得到超衍射极限的空间分辨率。仿真结果表明,当探测到的光子速率达到104 Hz时,分辨率可以达到50 nm。通过充分利用发射光子的量子时空波动和量子相干性,即使在荧光信号仅为103 Hz情况下,该方法仍可突破经典光源的衍射极限。
5 基于深度学习的荧光超分辨显微成像
荧光超分辨显微成像的重要目标是突破光学衍射极限,提升空间分辨能力,然而在实际应用中空间分辨率并不是唯一的性能指标,时间分辨率、成像速度、成像视场、光毒性和光漂白等指标也十分重要。这些指标在超分辨显微技术中相互限制,难以同时优化,例如在提高成像速度时必然会减少曝光时间,降低信噪比从而影响空间分辨率。这使得显微成像在亚细胞结构观测、活体生物超精密成像和分子结构研究领域的应用受到了极大阻碍。数据驱动的深度学习为提高超分辨显微技术的性能提供了一种变革性的解决方案,利用深度学习等方法对原始数据进行重建,以提高空间分辨率,不仅可以避免构建复杂的光学成像系统,而且能够在空间分辨率和时间分辨率等参数上取得良好的协同。因此,本节将重点介绍深度学习在超分辨显微图像计算重建中的基本概念和研究进展。
5.1 深度学习的基本概念和架构
自从20世纪50年代各种人工智能(AI)的概念被相继提出以来,机器学习(ML)作为人工智能的一部分,在过去几十年里取得了显著的进步,对许多应用领域进行了革命性的变革。如
![神经网络架构发展。(a)人工智能的分类[72];(b)卷积神经网络的基本架构,包括一个输入层、多个交替卷积层和最大池化层、一个全连接层和一个分类层[72];(c)生成对抗网络的基本架构[75];(d)U-Net网络的基本架构[76]](/richHtml/lop/2023/60/11/1106012/img_12.jpg)
图 12. 神经网络架构发展。(a)人工智能的分类[72];(b)卷积神经网络的基本架构,包括一个输入层、多个交替卷积层和最大池化层、一个全连接层和一个分类层[72];(c)生成对抗网络的基本架构[75];(d)U-Net网络的基本架构[76]
Fig. 12. Development of neural network architecture. (a) Taxonomy of AI[72]; (b) overall architecture of the CNN includes an input layer, multiple alternating convolution, and max-pooling layers, one fully-connected layer, and one classification layer[72]; (c) GAN model frame diagram[75]; (d) U-net architecture[76]
5.2 基于深度学习的荧光超分辨显微成像研究进展
根据以上论述可知,基于深度学习的图像处理方法不仅可以提高显微成像的空间分辨率,而且可以提高成像的时间分辨率。因此我们将分别从空间分辨率和时间分辨率两个角度介绍基于深度学习荧光超分辨显微成像的最新进展。
传统光学显微镜的成像能力通常受到像素数量的限制,即使使用不同的放大倍率和数值孔径的物镜,其图像的分辨率也难以大幅提高。这就导致了在追求高分辨率的同时,需要在视场范围上做出取舍。然而,生物医学成像领域往往需要实现大视场的高分辨率成像。2017年,美国加州大学洛杉矶分校的Ozcan课题组[77]首次将卷积神经网络引入宽场显微成像领域,训练后的网络能够显著提高在大视场、大景深下所获图像的空间分辨率。这一突破标志着深度学习技术与显微成像的完美结合,开启了全新的超分辨显微成像时代。2019年,该课题组又基于生成对抗网络实现了显微图像跨模态的转换,利用生成对抗网络提高低数值孔径物镜获取的宽视场图像分辨率,与高数值孔径物镜获得的分辨率相匹配,结果如
![基于深度学习荧光超分辨显微成像的最新进展。(a)通过深度学习对牛肺动脉内皮细胞宽场图像的重建结果[78];(b)基于深度学习的全内反射荧光显微效果,及其与基于全内反射的结构光超分辨显微成像的对比,所用细胞是SUM159[78];(c)利用生成对抗网络提高成像分辨率的效果[79];(d)基于无监督的内容保留变换显微成像可将宽场图像转换为超分辨图像,从而能够从宽场图像中分辨亚衍射结构,如微管和分泌颗粒[80];(e)对121组肌动蛋白图像分别使用scU-net、DFCAN和DFGAN网络重建后在归一化均方根误差、多尺度结构相似性指数和分辨率的统计比较,其中黑色的十字为异常值[82]](/richHtml/lop/2023/60/11/1106012/img_13.jpg)
图 13. 基于深度学习荧光超分辨显微成像的最新进展。(a)通过深度学习对牛肺动脉内皮细胞宽场图像的重建结果[78];(b)基于深度学习的全内反射荧光显微效果,及其与基于全内反射的结构光超分辨显微成像的对比,所用细胞是SUM159[78];(c)利用生成对抗网络提高成像分辨率的效果[79];(d)基于无监督的内容保留变换显微成像可将宽场图像转换为超分辨图像,从而能够从宽场图像中分辨亚衍射结构,如微管和分泌颗粒[80];(e)对121组肌动蛋白图像分别使用scU-net、DFCAN和DFGAN网络重建后在归一化均方根误差、多尺度结构相似性指数和分辨率的统计比较,其中黑色的十字为异常值[82]
Fig. 13. Latest progress in fluorescence super-resolution microscopy imaging based on deep learning. (a) Reconstruction results of wide-field images of bovine pulmonary artery endothelial cells through deep learning[78]; (b) total internal reflection fluorescence microscopic effect based on depth learning, and its comparison with structured light super-resolution microscopic imaging based on total internal reflection, the cell used is SUM159[78]; (c) resolution characterization of GAN net [79]; (d) based on unsupervised content retention transformation, microscopic imaging can convert wide-field images into super-resolution images, enabling the resolution of sub diffractive structures such as microtubules and secretory particles from the wide-field images[80]; (e) statistical comparison of normalized root mean square error, multi-scale structure similarity index, and resolution of 121 groups of actin images reconstructed by scU-net, DFCAN, and DFGAN networks respectively, black cross is the outlier[82]
鉴于监督学习方法依赖于注释数据集,而手工注释耗时且容易出错,2021年,清华大学的戴琼海课题组[80]提出了无监督的内容保留变换模型,即UTOM(Unsupervised content-preserving Transformation for Optical Microscopy),消除了对配对训练数据集的依赖,能够在无需配对训练数据集的情况下学习两个图像域之间的转换,结果如
受到成像原理的限制,使用超分辨显微技术获得超分辨图像往往需要大量的高质量原始数据,成像速度缓慢,这严重阻碍了超分辨显微技术在活细胞成像中的广泛应用。下面对目前使用深度学习方法提高超分辨显微成像时间分辨率的相关研究进行简要介绍。以结构光照明成像为例,当使用多个不同相位的结构光图案照明样品时,每个相位下的条纹形状和间距都不同,从而提供了更多的高频信息。因此,为了获取超分辨图像,结构光照明显微成像需要获取多张具有不同相位的原始帧,导致成像速度慢,不适用于长期的活细胞成像。2020年,深圳大学的Ling等[83]基于深度学习实现了快速结构光照明超分辨成像,他们在工作中使用由3张原始图像重建获得的单方向超分辨图像(原文中称其为1d_SIM),和由9张原始图像根据传统结构光照明显微成像重建算法获得的超分辨图像(原文中称其为9_SIM)作为训练数据集,训练后的循环生成对抗网络获得了光谱各个方向的高频信息之间的相关性,从而能够通过单向相移得到单方向超分辨图像的重建结果,与使用9张原始图像获得的超分辨重建结果具有相当的空间分辨率(原文中称其为3_SIM),结果如
![使用深度学习方法将线数据集中的图像从1d_SIM转换为9_SIM[83]。(a)宽场图像;(b)1d_SIM图像为网络输入;(c)3_SIM图像为网络输出;(d)9_SIM图像为真实图像。基于U-Net网络对微管成像的重构效果[81]。(e)15个SIM原始数据图像的平均投影;(f)传统结构光照明显微成像的重建算法的重建结果;(g)U-Net-SIM15网络的输出效果;(h)U-Net-SIM3网络的输出效果。ANNA-PALM重建免疫染色微管显微图像的结果对比[85]。(i)宽场图像;(j)从采集的前9 s获得的稀疏PALM图像(k=300 frame,n=11740个定位);(k)通过15 min的采集获得的密集PALM图像(K=30000 frame,N=409364个定位);(l)仅从宽场图像(i)重建的结果;(m)仅从稀疏的PALM图(j)重建的结果;(n)从宽场图(i)和稀疏PALM图(j)的组合中重建的结果(比例尺:1 μm)。COS-7细胞的双色sSMLM图像[86]。(o)3000 frame的低密度图像;(p)深度卷积神经网络的重建结果;(q)19997 frame的高密度图像(像素大小为16 nm,比例尺为1.5 μm)](/richHtml/lop/2023/60/11/1106012/img_14.jpg)
图 14. 使用深度学习方法将线数据集中的图像从1d_SIM转换为9_SIM[83]。(a)宽场图像;(b)1d_SIM图像为网络输入;(c)3_SIM图像为网络输出;(d)9_SIM图像为真实图像。基于U-Net网络对微管成像的重构效果[81]。(e)15个SIM原始数据图像的平均投影;(f)传统结构光照明显微成像的重建算法的重建结果;(g)U-Net-SIM15网络的输出效果;(h)U-Net-SIM3网络的输出效果。ANNA-PALM重建免疫染色微管显微图像的结果对比[85]。(i)宽场图像;(j)从采集的前9 s获得的稀疏PALM图像(k=300 frame,n=11740个定位);(k)通过15 min的采集获得的密集PALM图像(K=30000 frame,N=409364个定位);(l)仅从宽场图像(i)重建的结果;(m)仅从稀疏的PALM图(j)重建的结果;(n)从宽场图(i)和稀疏PALM图(j)的组合中重建的结果(比例尺:1 μm)。COS-7细胞的双色sSMLM图像[86]。(o)3000 frame的低密度图像;(p)深度卷积神经网络的重建结果;(q)19997 frame的高密度图像(像素大小为16 nm,比例尺为1.5 μm)
Fig. 14. Deep learning-enabled transformation of images of curves from 1d_SIM to 9_SIM[83]. (a) WF curve image; (b) 1d_SIM image is network input; (c) 3_SIM image is network output; (d) 9_SIM image is real images. Reconstruction effect of microtubule imaging based on U-Net network[81]. (e) Average projection of 15 SIM raw data images; (f) reconstruction results of reconstruction algorithm of traditional structured light illumination microscopic imaging; (g) output effect of U-Net-SIM15 network; (h) output effect of U-Net-SIM3 network. Comparison of results of ANNA-PALM reconstruction of immunostaining microtubule microscopy images[85]. (i) Widefield image; (j) sparse PALM image obtained from the first 9 s of acquisition (k=300 frame, n=11740 localizations); (k) dense PALM image obtained from a 15 min-long acquisition (K=30000 frame, N=409364 localizations); (l) ANNA-PALM reconstruction from the widefield Fig. (i) only; (m) ANNA-PALM reconstruction from the sparse PALM Fig. (j) only; (n) ANNA-PALM reconstruction from the widefield Fig. (i) and sparse PALM Fig. (j) combined (scale: 1 µm). Two-color sSMLM images in COS-7 cell[86]. (o) Low-density image with 3000 frame; (p) deep CNN reconstruction; (q) high-density image with 19997 frame (pixel size:16 nm. scale: 1.5 µm)
单分子定位显微成像包括随机光学重建显微成像和光激活定位显微成像,已突破传统光学荧光显微成像的分辨率。该技术首先获取大量连续衍射极限帧,利用每帧中对样品的稀疏荧光团成像,然后精确定位单个荧光团的位置,最后,组合这些帧的所有定位位置以生成超分辨率图像,这导致了较长的采集时间。2018年,法国巴黎巴斯德研究所的Ouyang等[85]提出了人工神经网络加速的光激活定位显微成像,即ANNA-PALM(Artificial Neural Network Accelerated PALM),其目标是从远小于标准光激发定位显微镜采集(具有K帧和N个定位点)的原始帧数(
基于深度学习的超分辨显微技术的出现,丰富了现有的显微成像技术,
表 1. 深度学习超分辨率成像的参数
Table 1. Parameters of the deep-learning super-resolution imaging
|
6 结束语
毫无疑问,超分辨荧光显微成像技术的发展为生命科学带来了前所未有的发展,为生命起源、重大疾病诊断、健康长寿解密等人们重点关注的问题带来了新的解决方案。目前有众多科研工作者及相关从业人员都集中在该领域,以期发展出具有更高空间分辨率和时间分辨率的超分辨显微技术,并努力降低相关仪器设备的使用门槛,使更多感兴趣者能够方便使用超分辨显微技术开展相关研究。因此,近年来超分辨荧光显微技术发展日新月异,受限于文章篇幅及作者的研究视野与写作能力,仅对该领域近期的部分研究成果进行了简单的说明,以期给该领域的研究人员提供一些参考。
本文主要对4种超分辨技术或方法的研究情况进行了简要说明,包括最低光子数超分辨荧光显微成像、波动超分辨荧光显微成像、基于荧光发色团反聚束效应的超分辨显微成像以及基于深度学习的超分辨显微成像方法。本文分别从这些超分辨荧光显微技术的基本原理、实验实现方法及相关要求、成像性能及其与相关技术的对比,以及与其他超分辨技术的结合等角度,对近期的研究进展和应用情况进行了说明。重点聚焦在最低光子数超分辨显微成像与基于深度学习的超分辨显微成像两方面,前者提出将发色团置于光场强度最低处,有效抑制了泊松噪声的影响,使超分辨成像对光子数的要求大大降低,不仅将荧光发色团的定位精度提高到单个发色团的物理尺寸(1 nm量级),而且大大提高了超分辨显微成像的时间分辨率,打破了超分辨显微成像空间分辨率与时间分辨率不可得兼的困境。因此该技术自被发明以来,得到了众多超分辨显微成像研究人员的青睐与关注,该技术也很快与其他超分辨显微技术相结合,比如双光子显微、结构光照明、图像扫描显微、受激辐射损耗等,使其空间分辨率、三维成像能力、成像视野等性能得到进一步提升。此外,也有很多科研工作者致力于研发最低光子数超分辨显微成像的设备,使其能够在常规的显微系统中实现超分辨成像效果,这极大了拓展了该技术的应用范围。而基于深度学习的超分辨显微成像技术则从计算重构的角度为超分辨显微提供了新的思路,特别是借助近年来人工智能领域的快速发展,大大加快了基于深度学习的超分辨显微成像技术的发展速度。该技术不需要构建复杂的超分辨成像系统,仅需要从数据挖掘的角度来思考和提升成像分辨能力,在一定程度上降低了超分辨成像的门槛。另一方面,波动超分辨荧光显微成像与反聚束超分辨显微成像都是利用光子的时空分布特性,其中波动超分辨利用的是荧光发色团在较长时间(ms量级)的闪烁特性,而反聚束超分辨利用的是荧光光子在较短时间(ns量级)的亚泊松分布特性。相比其他显微成像技术,波动超分辨显微成像对光束的操控以及光学系统的要求都比较低,便于使用,然而其缺陷也较为明显,即对空间分辨率的提升有限。而反聚束超分辨显微成像作为量子超分辨显微成像的代表,目前仍处于发展阶段,尚未展现出巨大的优势,但在不久的将来,随着量子科技的快速发展,量子超分辨显微成像很可能为超分辨荧光显微成像带来新的机遇。
对于超分辨荧光显微成像的未来,限于作者水平,我们很难给出定言,但很可能会在以下几方面取得新的发展。其一仍然是进一步提高超分辨显微成像的性能,包括空间分辨率、时间分辨率、成像视野、光毒性等。目前部分超分辨显微成像的空间分辨率已经达到了令人满意的程度,然而时间分辨率还有待进一步提高,以此来研究具有较快动力学行为的生理反应。未来可能要特别关注成像视野的问题:一个普遍的观点是成像分辨率和成像视野不可得兼,然而过小的成像视野会限制对很多关联体系之间相互作用的研究,不利于超分辨显微成像的实际应用。光毒性是活体超分辨成像必须关注的问题,特别是在未来将超分辨显微成像应用于临床医学中,降低激光功率是不可避免的要求。其二是发展更易使用的超分辨成像算法。尽管目前已经发展了很多超分辨显微成像技术,但无疑都要求对原始成像数据进行特定的处理,这些处理方法目前主要处于实验室阶段,还没有完全实现普及化和自动化,要求使用者具有相当的技术背景才能实现较好的成像效果,在一定程度上限制了超分辨显微成像的使用。另一方面,基于深度学习的超分辨显微成像技术缺乏泛化能力,难以处理不同方法和不同环境下获得的原始成像数据,这可能也是未来数据处理需要解决的问题。其三是推广超分辨荧光显微成像的实际应用。目前发展的诸多超分辨荧光显微成像技术主要还是应用在科研院所,需要由科研人员或受过相当训练的工作人员才能使用,极大限制了这些技术的应用范围。瓶颈主要体现在两方面:在硬件方面,超分辨荧光显微成像往往涉及较为复杂的光学系统,模块化和集成化可能是简化光学系统,降低使用门槛的重要手段;在软件方面,简化数据处理流程,减少操作步骤,设计客户友好的访问界面,可能是推广超分辨显微成像应用必须解决的问题。总而言之,在不久的将来,超分辨荧光显微成像很可能像目前常规的光学显微镜一样,进入到各个领域,为探索生命活动,揭示生命起源带来无穷的想象。
[1] Lim C S, Kim E S, Kim J Y, et al. Measurement of the nucleus area and nucleus/cytoplasm and mitochondria/nucleus ratios in human colon tissues by dual-colour two-photon microscopy imaging[J]. Scientific Reports, 2015, 5(1): 1-11.
[2] Kampmann M, Atkinson C E, Mattheyses A L, et al. Mapping the orientation of nuclear pore proteins in living cells with polarized fluorescence microscopy[J]. Nature Structural & Molecular Biology, 2011, 18(6): 643-649.
[3] Okabe K, Inada N, Gota C, et al. Intracellular temperature mapping with a fluorescent polymeric thermometer and fluorescence lifetime imaging microscopy[J]. Nature Communications, 2012, 3(1): 1-9.
[4] Summers P A, Lewis B W, Gonzalez-Garcia J, et al. Visualising G-quadruplex DNA dynamics in live cells by fluorescence lifetime imaging microscopy[J]. Nature Communications, 2021, 12(1): 1-11.
[5] Schermelleh L, Ferrand A, Huser T, et al. Super-resolution microscopy demystified[J]. Nature Cell Biology, 2019, 21(1): 72-84.
[6] Abbe E. Beiträge zur theorie des mikroskops und der mikroskopischen wahrnehmung[J]. Archiv Für Mikroskopische Anatomie, 1873, 9(1): 413-418.
[7] Moerner W E, Kador L. Optical detection and spectroscopy of single molecules in a solid[J]. Physical Review Letters, 1989, 62(21): 2535-2538.
[8] Dickson R M, Cubitt A B, Tsien R Y, et al. On/off blinking and switching behaviour of single molecules of green fluorescent protein[J]. Nature, 1997, 388(6640): 355-358.
[9] Betzig E, Patterson G H, Sougrat R, et al. Imaging intracellular fluorescent proteins at nanometer resolution[J]. Science, 2006, 313(5793): 1642-1645.
[10] Rust M J, Bates M, Zhuang X W. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM)[J]. Nature Methods, 2006, 3(10): 793-796.
[11] Hell S W, Wichmann J. Breaking the diffraction resolution limit by stimulated emission: stimulated-emission-depletion fluorescence microscopy[J]. Optics Letters, 1994, 19(11): 780-782.
[12] Gustafsson M G. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy[J]. Journal of Microscopy, 2000, 198(2): 82-87.
[13] Sigal Y M, Zhou R B, Zhuang X W. Visualizing and discovering cellular structures with super-resolution microscopy[J]. Science, 2018, 361(6405): 880-887.
[14] Qiao C, Chen X Y, Zhang S W, et al. 3D structured illumination microscopy via channel attention generative adversarial network[J]. IEEE Journal of Selected Topics in Quantum Electronics, 2021, 27(4): 6801711.
[15] Vicidomini G, Bianchini P, Diaspro A. STED super-resolved microscopy[J]. Nature Methods, 2018, 15(3): 173-182.
[16] Balzarotti F, Eilers Y, Gwosch K C, et al. Nanometer resolution imaging and tracking of fluorescent molecules with minimal photon fluxes[J]. Science, 2017, 355(6325): 606-612.
[17] Xiao J, Ha T. Flipping nanoscopy on its head[J]. Science, 2017, 355(6325): 582-584.
[18] Eilers Y, Ta H S, Gwosch K C, et al. MINFLUX monitors rapid molecular jumps with superior spatiotemporal resolution[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(24): 6117-6122.
[19] Gwosch K C, Pape J K, Balzarotti F, et al. MINFLUX nanoscopy delivers 3D multicolor nanometer resolution in cells[J]. Nature Methods, 2020, 17(2): 217-224.
[20] Pape J K, Stephan T, Balzarotti F, et al. Multicolor 3D MINFLUX nanoscopy of mitochondrial MICOS proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(34): 20607-20614.
[21] Masullo L A, Steiner F, Zähringer J, et al. Pulsed interleaved MINFLUX[J]. Nano Letters, 2021, 21(1): 840-846.
[22] Schmidt R, Weihs T, Wurm C A, et al. MINFLUX nanometer-scale 3D imaging and microsecond-range tracking on a common fluorescence microscope[J]. Nature Communications, 2021, 12(1): 1478.
[23] Masullo L A, Szalai A M, Lopez L F, et al. An alternative to MINFLUX that enables nanometer resolution in a confocal microscope[J]. Light: Science & Applications, 2022, 11(1): 1-9.
[24] Masullo L A, Stefani F D. Multiphoton single-molecule localization by sequential excitation with light minima[J]. Light: Science & Applications, 2022, 11(1): 1-4.
[25] Zhao K, Xu X Z, Ren W, et al. Two-photon MINFLUX with doubled localization precision[J]. eLight, 2022, 2(1): 1-10.
[26] Müller C B, Enderlein J. Image scanning microscopy[J]. Physical Review Letters, 2010, 104(19): 198101.
[28] Cnossen J, Hinsdale T, Thorsen R Ø, et al. Localization microscopy at doubled precision with patterned illumination[J]. Nature Methods, 2020, 17(1): 59-63.
[29] Weber M, Leutenegger M, Stoldt S, et al. MINSTED fluorescence localization and nanoscopy[J]. Nature Photonics, 2021, 15(5): 361-366.
[30] Schnitzbauer J, Strauss M T, Schlichthaerle T, et al. Super-resolution microscopy with DNA-PAINT[J]. Nature Protocols, 2017, 12(6): 1198-1228.
[31] Ostersehlt L M, Jans D C, Wittek A, et al. DNA-PAINT MINFLUX nanoscopy[J]. Nature Methods, 2022, 19(9): 1072-1075.
[32] Dertinger T, Colyer R, Iyer G, et al. Fast, background-free, 3D super-resolution optical fluctuation imaging (SOFI)[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52): 22287-22292.
[33] Dertinger T, Colyer R, Vogel R, et al. Achieving increased resolution and more pixels with superresolution optical fluctuation imaging (SOFI)[J]. Optics Express, 2010, 18(18): 18875-18885.
[34] Geissbuehler S, Dellagiacoma C, Lasser T. Comparison between sofi and storm[J]. Biomedical Optics Express, 2011, 2(3): 408-420.
[35] Michalet X, Pinaud F F, Bentolila L A, et al. Quantum dots for live cells, in vivo imaging, and diagnostics[J]. Science, 2005, 307(5709): 538-544.
[36] Murray C B, Kagan C R, Bawendi M G. Synthesis and characterization of monodisperse nanocrystals and close-packed nanocrystal assemblies[J]. Annual Review of Materials Science, 2000, 30(1): 545-610.
[37] Zeng Z P, Chen X Z, Wang H N, et al. Fast super-resolution imaging with ultra-high labeling density achieved by joint tagging super-resolution optical fluctuation imaging[J]. Scientific Reports, 2015, 5(1): 1-7.
[38] Grußmayer K S, Geissbuehler S, Descloux A, et al. Spectral cross-cumulants for multicolor super-resolved SOFI imaging[J]. Nature Communications, 2020, 11(1): 1-8.
[39] Uno S N, Kamiya M, Yoshihara T, et al. A spontaneously blinking fluorophore based on intramolecular spirocyclization for live-cell super-resolution imaging[J]. Nature Chemistry, 2014, 6(8): 681-689.
[40] Grußmayer K, Lukes T, Lasser T, et al. Self-blinking dyes unlock high-order and multiplane super-resolution optical fluctuation imaging[J]. ACS Nano, 2020, 14(7): 9156-9165.
[41] Dedecker P, Mo G C H, Dertinger T, et al. Widely accessible method for superresolution fluorescence imaging of living systems[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(27): 10909-10914.
[42] Glogger M, Spahn C, Enderlein J, et al. Multi-color, bleaching-resistant super-resolution optical fluctuation imaging with oligonucleotide-based exchangeable fluorophores[J]. Angewandte Chemie, 2021, 60(12): 6310-6313.
[43] Kim M, Park C, Rodriguez C, et al. Superresolution imaging with optical fluctuation using speckle patterns illumination[J]. Scientific Reports, 2015, 5(1): 1-10.
[44] Yi X Y, Son S, Ando R, et al. Moments reconstruction and local dynamic range compression of high order superresolution optical fluctuation imaging[J]. Biomedical Optics Express, 2019, 10(5): 2430-2445.
[45] Geissbuehler S, Bocchio N L, Dellagiacoma C, et al. Mapping molecular statistics with balanced super-resolution optical fluctuation imaging (bSOFI)[J]. Optical Nanoscopy, 2012, 1(1): 1-7.
[46] Dedecker P, Duwé S, Neely R K, et al. Localizer: fast, accurate, open-source, and modular software package for superresolution microscopy[J]. Journal of Biomedical Optics, 2012, 17(12): 126008.
[47] Moeyaert B, Vandenberg W, Dedecker P. SOFIevaluator: a strategy for the quantitative quality assessment of SOFI data[J]. Biomedical Optics Express, 2020, 11(2): 636-648.
[48] Girsault A, Lukes T, Sharipov A, et al. SOFI simulation tool: a software package for simulating and testing super-resolution optical fluctuation imaging[J]. PLoS One, 2016, 11(9): e0161602.
[49] Geissbuehler S, Sharipov A, Godinat A, et al. Live-cell multiplane three-dimensional super-resolution optical fluctuation imaging[J]. Nature Communications, 2014, 5(1): 1-7.
[51] Sroda A, Makowski A, Tenne R, et al. SOFISM: super-resolution optical fluctuation image scanning microscopy[J]. Optica, 2020, 7(10): 1308-1316.
[52] Aßmann M. Quantum-optically enhanced STORM (QUEST) for multi-emitter localization[J]. Scientific Reports, 2018, 8(1): 1-12.
[53] Mo G C H, Ross B, Hertel F, et al. Genetically encoded biosensors for visualizing live-cell biochemical activity at super-resolution[J]. Nature Methods, 2017, 14(4): 427-434.
[54] Giovannetti V, Lloyd S, Maccone L. Quantum-enhanced measurements: beating the standard quantum limit[J]. Science, 2004, 306(5700): 1330-1336.
[55] Brida G, Genovese M, Ruo Berchera I. Experimental realization of sub-shot-noise quantum imaging[J]. Nature Photonics, 2010, 4(4): 227-230.
[56] Tsang M. Quantum imaging beyond the diffraction limit by optical centroid measurements[J]. Physical Review Letters, 2009, 102(25): 253601.
[57] Giovannetti V, Lloyd S, Maccone L, et al. Sub-Rayleigh-diffraction-bound quantum imaging[J]. Physical Review A, 2009, 79(1): 013827.
[58] D’Angelo M, Chekhova M V, Shih Y. Two-photon diffraction and quantum lithography[J]. Physical Review Letters, 2001, 87(1): 013602.
[59] Walls D F, Zoller P. Reduced quantum fluctuations in resonance fluorescence[J]. Physical Review Letters, 1981, 47(10): 709-711.
[60] Mandel L. Sub-Poissonian photon statistics in resonance fluorescence[J]. Optics Letters, 1979, 4(7): 205-207.
[61] Senellart P, Solomon G, White A. High-performance semiconductor quantum-dot single-photon sources[J]. Nature Nanotechnology, 2017, 12(11): 1026-1039.
[62] Kimble H J, Dagenais M, Mandel L. Photon antibunching in resonance fluorescence[J]. Physical Review Letters, 1977, 39(11): 691-695.
[63] Schwartz O, Oron D. Improved resolution in fluorescence microscopy using quantum correlations[J]. Physical Review A, 2012, 85(3): 033812.
[64] Schwartz O, Levitt J M, Tenne R, et al. Superresolution microscopy with quantum emitters[J]. Nano Letters, 2013, 13(12): 5832-5836.
[65] Cui J M, Sun F W, Chen X D, et al. Quantum statistical imaging of particles without restriction of the diffraction limit[J]. Physical Review Letters, 2013, 110(15): 153901.
[66] Monticone D G, Katamadze K, Traina P, et al. Beating the Abbe diffraction limit in confocal microscopy via nonclassical photon statistics[J]. Physical Review Letters, 2014, 113(14): 143602.
[67] Israel Y, Tenne R, Oron D, et al. Quantum correlation enhanced super-resolution localization microscopy enabled by a fibre bundle camera[J]. Nature Communications, 2017, 8: 14786.
[68] Classen A, von Zanthier J, Scully M O, et al. Superresolution via structured illumination quantum correlation microscopy[J]. Optica, 2017, 4(6): 580-587.
[69] Tenne R, Rossman U, Rephael B, et al. Super-resolution enhancement by quantum image scanning microscopy[J]. Nature Photonics, 2019, 13(2): 116-122.
[70] Li W W, Wang Z Y. Breaking the diffraction limit using fluorescence quantum coherence[J]. Optics Express, 2022, 30(8): 12684-12694.
[71] LeCun Y, Bengio Y, Hinton G. Deep learning[J]. Nature, 2015, 521(7553): 436-444.
[72] Alom M Z, Taha T M, Yakopcic C, et al. A state-of-the-art survey on deep learning theory and architectures[J]. Electronics, 2019, 8(3): 292.
[73] Fukushima K. Neocognitron: a hierarchical neural network capable of visual pattern recognition[J]. Neural Networks, 1988, 1(2): 119-130.
[74] Goodfellow I, Pouget-Abadie J, Mirza M, et al. Generative adversarial networks[J]. Communications of the ACM, 2020, 63(11): 139-144.
[75] Cheng J R, Yang Y, Tang X Y, et al. Generative adversarial networks: a literature review[J]. KSII Transactions on Internet and Information Systems, 2020, 14(12): 4625-4647.
[76] RonnebergerO, FischerP, BroxT. U-Net: convolutional networks for biomedical image segmentation[M]∥Navab N, Hornegger J, Wells W M, et al. Medical image computing and computer-assisted intervention-MICCAI 2015. Lecture notes in computer science. Cham: Springer, 2015, 9351: 234-241.
[77] Rivenson Y, Göröcs Z, Günaydin H, et al. Deep learning microscopy[J]. Optica, 2017, 4(11): 1437-1443.
[78] Wang H D, Rivenson Y, Jin Y Y, et al. Deep learning enables cross-modality super-resolution in fluorescence microscopy[J]. Nature Methods, 2019, 16(1): 103-110.
[79] Zhang H, Fang C Y, Xie X L, et al. High-throughput, high-resolution deep learning microscopy based on registration-free generative adversarial network[J]. Biomedical Optics Express, 2019, 10(3): 1044-1063.
[80] Li X Y, Zhang G X, Qiao H, et al. Unsupervised content-preserving transformation for optical microscopy[J]. Light: Science & Applications, 2021, 10(1): 1-11.
[81] Jin L H, Liu B, Zhao F Q, et al. Deep learning enables structured illumination microscopy with low light levels and enhanced speed[J]. Nature Communications, 2020, 11(1): 1-7.
[82] Qiao C, Li D, Guo Y T, et al. Evaluation and development of deep neural networks for image super-resolution in optical microscopy[J]. Nature Methods, 2021, 18(2): 194-202.
[84] Zhang Q N, Chen J W, Li J S, et al. Deep learning-based single-shot structured illumination microscopy[J]. Optics and Lasers in Engineering, 2022, 155: 107066.
[85] Ouyang W, Aristov A, Lelek M, et al. Deep learning massively accelerates super-resolution localization microscopy[J]. Nature Biotechnology, 2018, 36(5): 460-468.
[86] Gaire S K, Zhang Y, Li H Y, et al. Accelerating multicolor spectroscopic single-molecule localization microscopy using deep learning[J]. Biomedical Optics Express, 2020, 11(5): 2705-2721.
[87] Speiser A, Müller L R, Hoess P, et al. Deep learning enables fast and dense single-molecule localization with high accuracy[J]. Nature Methods, 2021, 18(9): 1082-1090.
[88] Nehme E, Weiss L E, Michaeli T, et al. Deep-STORM: super-resolution single-molecule microscopy by deep learning[J]. Optica, 2018, 5(4): 458-464.
[89] Fang L J, Monroe F, Novak S W, et al. Deep learning-based point-scanning super-resolution imaging[J]. Nature Methods, 2021, 18(4): 406-416.
Article Outline
卫奥尼, 秦成兵, 董帅, 孟新钦, 宋蕴睿, 李向东, 梁喜龙, 张国峰, 陈瑞云, 胡建勇, 杨志春, 霍建忠, 肖连团, 贾锁堂. 超分辨荧光显微成像的若干研究进展[J]. 激光与光电子学进展, 2023, 60(11): 1106012. Aoni Wei, Chengbing Qin, Shuai Dong, Xinqin Meng, Yunrui Song, Xiangdong Li, Xilong Liang, Guofeng Zhang, Ruiyun Chen, Jianyong Hu, Zhichun Yang, Jianzhong Huo, Liantuan Xiao, Suotang Jia. Research Progress of Super-Resolution Fluorescence Microscopy[J]. Laser & Optoelectronics Progress, 2023, 60(11): 1106012.






